Отобранные бактерии:
| Название | Мнемоника |
| Bacillus anthracis | BACAN |
| Clostridium botulinum | CLOB1 |
| Finegoldia magna | FINM2 |
| Lactobacillus acidophilus | LACAC |
| Listeria monocytogenes | LISMO |
| Staphylococcus aureus | STAA1 |
| Streptococcus pyogenes | STRP1 |
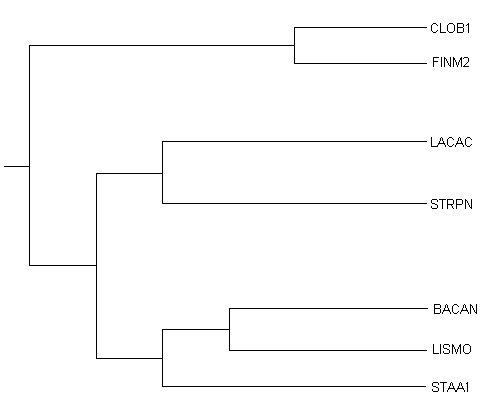
Скобочная формула дерева:
((CLOB1, FINM2),((LACAC, STRPN),((BACAN, LISMO), STAA1)));
Изображение дерева:
Ветви дерева:
Дерево содержит 3 нетривиальные ветви:
1) {BACAN, LISMO} против {STAA1, LACAC, STRPN, CLOB1, FINM2}
2) {BACAN, LISMO, STAA1} против {LACAC, STRPN, CLOB1, FINM2}
3) {BACAN, LISMO, STAA1, CLOB1, FINM2} против {LACAC, STRPN}
1. Пользуясь таксономическим сервисом NCBI, было определено, что данные бактерии относятся к следующим таксонам:
Bacillus anthracis : root; cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group
Clostridium botulinum : root; cellular organisms; Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium
Finegoldia magna : root; cellular organisms; Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiales incertae sedis; Clostridiales Family XI. Incertae Sedis; Finegoldia
Lactobacillus acidophilus : root; cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus
Listeria monocytogenes : root; cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria
Staphylococcus aureus : root; cellular organisms; Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus
Streptococcus pyogenes : root; cellular organisms; Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus
Если сопоставить изображение дерева и данные полученные сервисом NCBI, то видно, что на дереве отобранных бактерий есть ветви, выделяющие какие-нибудь из таксонов:
1) Отходящая от корня ветвь разделяется на 2 ветви, разделяющие классы Clostridia (верхняя) и Bacilli (нижняя).
2) Нижняя ветвь разделяется на 2 ветви, соответствующим порядкам Lactobacillales (верхняя) и Bacillales (нижняя).
3) Ветвь, соответствующая порядку Bacillales, разделяется на семейства Bacillaceae (верхняя), Listeriaceae (средняя) и Staphylococcaceae (нижняя).
2. Реконструкция дерева на основании выравнивания(с помощью программы muscle) выбранных ранее белков и фактор элонгации трансляции Ts программой fprotpars.
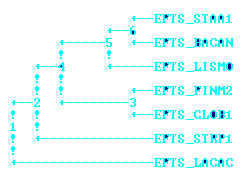
скобочная формула: (((((EFTS_STAA1,EFTS_BACAN),EFTS_LISMO),(EFTS_FINM2,EFTS_CLOB1)),
EFTS_STRP1),EFTS_LACAC);
Общих ветвей с правильным деревом 3:{LACAC, STRP1}против {BACAN, LISMO, STAA1,FINM2, CLOB1}, {FINM2, CLOB1}против {BACAN, LISMO, STAA1,LACAC, STRP1}, {BACAN, LISMO, STAA1} против {LACAC, STRP1, FINM2, CLOB1}.
3. Оценка эволюционных расстояний
EFTS_LACAC EFTS_STRP1 EFTS_CLOB1 EFTS_FINM2 EFTS_LISMO EFTS_BACAN EFTS_STAA1
EFTS_LACAC 0.000000 0.417534 1.030063 0.840702 0.799293 0.795829 0.746434
EFTS_STRP1 0.417534 0.000000 1.133174 0.869057 0.825243 0.871535 0.819417
EFTS_CLOB1 1.030063 1.133174 0.000000 0.640800 0.817756 0.854007 0.831040
EFTS_FINM2 0.840702 0.869057 0.640800 0.000000 0.623031 0.661217 0.633879
EFTS_LISMO 0.799293 0.825243 0.817756 0.623031 0.000000 0.449536 0.455759
EFTS_BACAN 0.795829 0.871535 0.854007 0.661217 0.449536 0.000000 0.374539
EFTS_STAA1 0.746434 0.819417 0.831040 0.633879 0.455759 0.374539 0.000000
Отклонение от ультраметричности
Рассмотрим LACAC, CLOB1, STRP1.
d(CLOB1,STRP1) = 1.133174 > d(STRP1, LACAC) = 0.417534 => d(CLOB1,LACAC) == d(CLOB1,STRP1) - так должно быть
Но: d(CLOB1,LACAC) = 1.030063, а d(CLOB1,STRP1) = 1.133174 - отклонение от ультраметричности на 0.103111
Отклонение от аддитивности
Рассмотрим LACAC, CLOB1, STRP1, LISMO.
d(LACAC,STRP1) = 0.417534 | Суммы:
d(LACAC,CLOB1) = 1.030063 | 1) 0.417534 + 0.817756 = 1,235290
d(LACAC,LISMO) = 0.799293 | 2) 1.030063 + 0.825243 = 1,855306
d(STRP1,CLOB1) = 1.133174 | 3) 0.799293 + 1.133174 = 1,932367
d(STRP1,LISMO) = 0.825243 |
d(CLOB1,LISMO) = 0.817756 | Отклонеие: 0,077061
4. Реконструкция дерева программой fneighbor
Алгоритм Neighbor-Joining
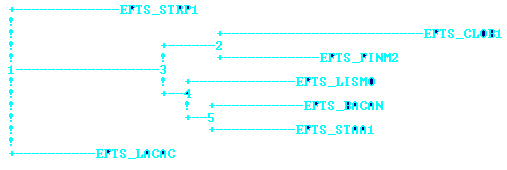
Общие ветви с правильным деревом:
{FINM2, CLOB1}против {BACAN, LISMO, STAA1,LACAC, STRP1}, {BACAN, LISMO, STAA1} против {LACAC, STRP1, FINM2, CLOB1}.
Алгоритм UPGMA

Корень дерева:{LACAC, STRP1}против {BACAN, LISMO, STAA1,FINM2, CLOB1}Полученный результат опять отличается от правильного дерева. общие ветви: {BACAN, LISMO, STAA1} против {LACAC, STRP1, FINM2, CLOB1}, {LACAC, STRP1}против {BACAN, LISMO, STAA1,FINM2, CLOB1}
Четвертый семестр
© Чернецова Даша



