Реконструкция филогении по нуклеотидным последовательнсотям
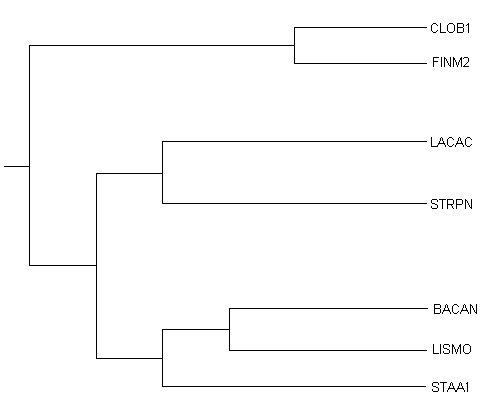
Правильное дерево:
Я реконструировала дерево программой fdnapars. Программа выдала одно дерево:
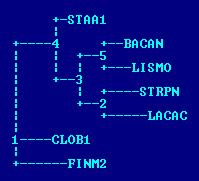
Как видно, дерево не совпадает с правильным. Количество правильных ветвей - 3. Не удалось воспроизвести ветвь {STAA1, BACAN, LISMO} против {LACAC, CLOB1, FINM2, STRPN}.Однако, что важно, только программе fdnapars удалось выделить BACAN, LISMO, STAA1 в правильную ветвь.
Реконструкция программой fprotpars воспроизводит 3 правильных ветвей. Получается, что одинаково.
ссылка на выравнивание
Построение и анализ дерева, содержащего паралоги
Находки Blast:
Subject id e-value bit score
sp|Q81UJ9|3MGH_BACAN e-119 423
sp|Q896H4|3MGH_CLOTE 1e-53 204
sp|A7FTE3|3MGH_CLOB1 5e-47 182
sp|P94378|3MGH_BACSU 3e-35 143
sp|O31544|YFJP_BACSU e-180 624
sp|Q833H5|3MGH_ENTFA 2e-149 130
sp|A7X5V7|3MGH_STAA1 1e-31 104
sp|Q5FMR0|3MGH_LACAC 2e-20 100
sp|O05413|2NPD_BACSU 0.0 715
Реконструкция дерева программой fprotpars
+--------3MGH_BACSU
!
+--------7 +--3MGH_CLOB1
! ! +--5
! +--4 +--3MGH_CLOTE
! !
+--3 +-----3MGH_BACAN
! !
! ! +--3MGH_ENTFA
! ! +--8
+--2 +-----------6 +--3MGH_STAA1
! ! !
! ! +-----3MGH_LACAC
1 !
! +--------------------YFJP_BACSU
!
+-----------------------2NPD_BACSU
Ортологи: 3MGH_ENTFA и 3MGH_STAA1; 3MGH_CLOB1 и 3MGH_ENTFA
Паралоги: 2NPD_BACSU и 3MGH_BACSU; 3MGH_BACSU и YFJP_BACSU
Четвертый семестр
© Чернецова Даша

