Чтобы начать использование программы соединяемся с сервером: export PATH=$PATH:/home/preps/grishin/open3dtools/bin
open3dalign.sh - запускаем программу.
Чтобы сделать выравнивание нужно загрузить ваш SDF файл со структурами веществ (команда import), выполнить выравнивание (чтобы использовать в качестве темплэйта, к которому выравниваются все вещества, первое вещество в списке, наберите align object_list=1), и записать выравнивание в файл (save).
Далее необходимо перекодировать из юникода в ascii:iconv -c -f utf-8 -t ascii aligned.sdf > aligned_ascii.sdf и удалить ненужную информацию из заголовков,добавив $$$$ в конец каждой записи:
sed -e 's/.*HEADER.*\([0-9][0-9]\).*/\1/' -e 's/\(.*M END.*\)/\1\n$$$$/' aligned_ascii.sdf > temp
sed -n '/^[0-9a-zA-Z \$\.-]*$/ p' temp > aligned_ok.sdf
rm temp
В итоге получили файл aligned_ok.sdf.
Далее 33DQSAR для регрессионной модели.
Использованные команды:
open3dqsar.sh - запуск программы
import type=sdf file=aligned_ok.sdf - загрузка файла со структурами
import type=dependent file=activity.txt - загрузка файла с данными об активности исследуемых соединений.
box - задание решетку вокруг исследуемых соединений
set object_list=60-85 attribute=TEST - соединения с 60 по 85 оставляем в качетсве тестового набора, для построения модели они не используются
set object_list=86-88 attribute=EXCLUDED - исключение соединений с неизвестной активностью
calc_field type=VDW force_field=MMFF94 probe_type=CR - рассчет значений энергии ван-дер-Ваальсовых взаимодействий в узлах решетки
cutoff type=max level=5.0 field_list=1 - ограничения на значения энергии
cutoff type=min level=-5.0 field_list=1 -ограничения на значения энергии
zero type=all level=0.05 - приравнивание слишком маленьких значений энергии к 0
sdcut level=0.1, nlevel, remove_x_vars type=nlevel - исключение из анализа ячейки, в которых вариабельность в энергии взаимодействия с зондом для разных соединений мала
pls - построение регрессионной модели
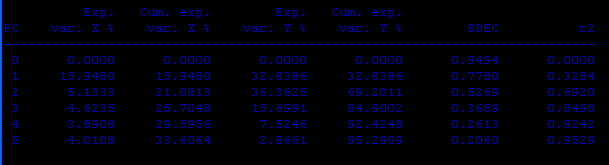
Некоторые коэффициенты (3 последних) близки к 1, что очень хорошо.
cv type=loo runs=20 - команда для выполнения кросс-валидации. Вот, что получили:
PC SDEP q2 -------------------------- 0 0.9658 -0.0348 1 0.9164 0.0683 2 0.9733 -0.0509 3 0.9667 -0.0368 4 0.9880 -0.0829 5 0.9497 -0.0006predict - предсказание активости для тестовой выборки. Получили:
PC r2(pred) SDEP -------------------------- 0 0.0000 1.0362 1 0.2655 0.8881 2 0.3296 0.8484 3 0.2353 0.9061 4 0.2754 0.8821 5 0.2536 0.8953В первом случае коэффициенты получились очень плохие, в случае тестовой выборки они далеки от идеала, но значительно лучше первого случая.
Теперь 33DQSAR для регрессионной модели с учетом структуры активного центра белка-мишени.
Команды используются все те же, файл с выравниваением - aligned_ok1.sdf
Регрессионная модель:
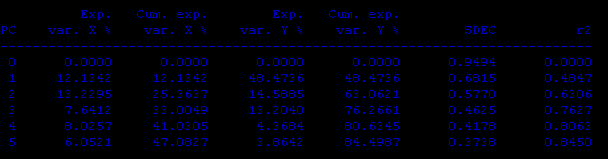
Кросс-валидация:PC SDEP q2 -------------------------- 0 0.9658 -0.0348 1 0.8027 0.2851 2 0.7664 0.3484 3 0.7061 0.4468 4 0.6735 0.4968 5 0.6401 0.5454Предсказание:PC r2(pred) SDEP -------------------------- 0 0.0000 1.0362 1 0.3451 0.8385 2 0.3226 0.8529 3 0.2998 0.8671 4 0.3012 0.8662 5 0.2693 0.8858Наблюдается улучшение коэффициентов, но незначительное.Предсказание активностей с помощью полученной модели.
Вначале переделаем модель с использованием всех имеющихся данных, а вещества с неизвестной активностью обозначим как тестовую выборку:
set object_list=60-85 attribute=TRAINING
set object_list=86-88 attribute=TEST Регрессионная модель:
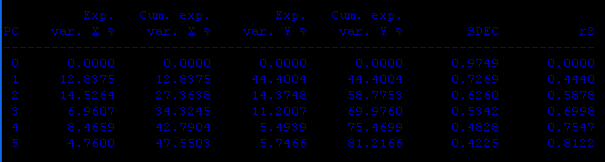
Кросс-валидация:
PC SDEP q2 -------------------------- 0 0.9865 -0.0240 1 0.8233 0.2868 2 0.7521 0.4049 3 0.7084 0.4720 4 0.6963 0.4899 5 0.7061 0.4754PC r2(pred) SDEP -------------------------- 0 0.0000 6.6604 1 0.0298 6.5603 2 -0.0155 6.7118 3 0.0082 6.6331 4 -0.0627 6.8660 5 -0.1011 6.9889External predictions for dependent variable 1 (activity) -------------------------------------------------------------------------------------------------------------------------------------- N ID Name Actual 1 2 3 4 5 Opt PC n -------------------------------------------------------------------------------------------------------------------------------------- 86 86 01 0.0000 7.1119 7.5466 7.4119 7.6262 7.7234 1 87 87 44 0.0000 6.9428 7.1202 7.0946 7.3278 7.5477 1 88 88 72 0.0000 5.5073 5.2436 5.1697 5.4378 5.4696 3Видно, что коэффициенты при кросс-валидации лучше, чем в предсказании,наилучший коэффициент (q2) у 4. Поэтому и смотрим для предсказания на столбец под цифрой 4.
Шестой семестр
© Чернецова Даша