Практикум 4
Список выбранных протеомов
ACICJ, BARHE, BURMA, NEIMA, POLAQ, PSEMY, SACD2
Составление списка белков
Объединяем эти протеомы в один файл
cat /P/y22/term4/Proteomes/ACICJ.fasta /P/y22/term4/Proteomes/BARHE.fasta /P/y22/term4/Proteomes/BURMA.fasta /P/y22/term4/Proteomes/NEIMA.fasta /P/y22/term4/Proteomes/POLAQ.fasta /P/y22/term4/Proteomes/PSEMY.fasta /P/y22/term4/Proteomes/SACD2.fasta > db.fasta
делаем базу данных для blastp
makeblastdb -in db.fasta -dbtype prot
Делаем бласт, в качестве запроса - белок CLPX_ECOLI. Сразу поставил порог на e-value
blastp -task blastp -query q.fasta -db db.fasta -out pr4.out -evalue 0.0001
Ссылка на файл с результатами
Список гомологичных белков
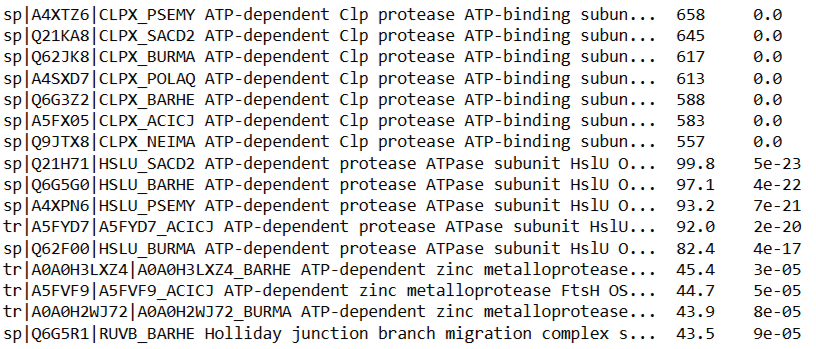
Список гомологичных белков
Реконструкция и визуализация
После BLAST я вытащил все значимые белки по мнемоникам, переименовал заголовки в фасте, оставив только эти короткие названия. Выравнял последовательности через MUSCLE, сконвертил выравнивание в PHYLIP. Построил дерево в FastME по модели MtREV с бутстрепом (100 повторов). Получил файл tree_mtrev.nwk для визуализации. Сделал визуализацию с помощью iTol.
Ссылка на файл в формате nwk
Получившееся дерево

Получившееся дерево
Примеры пар ортологов: CLPX_PSEMY и CLPX_SACD2, HSLU_PSEMY и HSLU_SACD2, CLPX_POLAQ и CLPX_BURMA.
Примеры пар паралогов: RUVB_BARHE и CLPX_BARHE, RUVB_BARHE и HSLU_BARHE, A5FYD7_ACICJ и CLPX_ACICJ.
Дерево с ортологическими группами
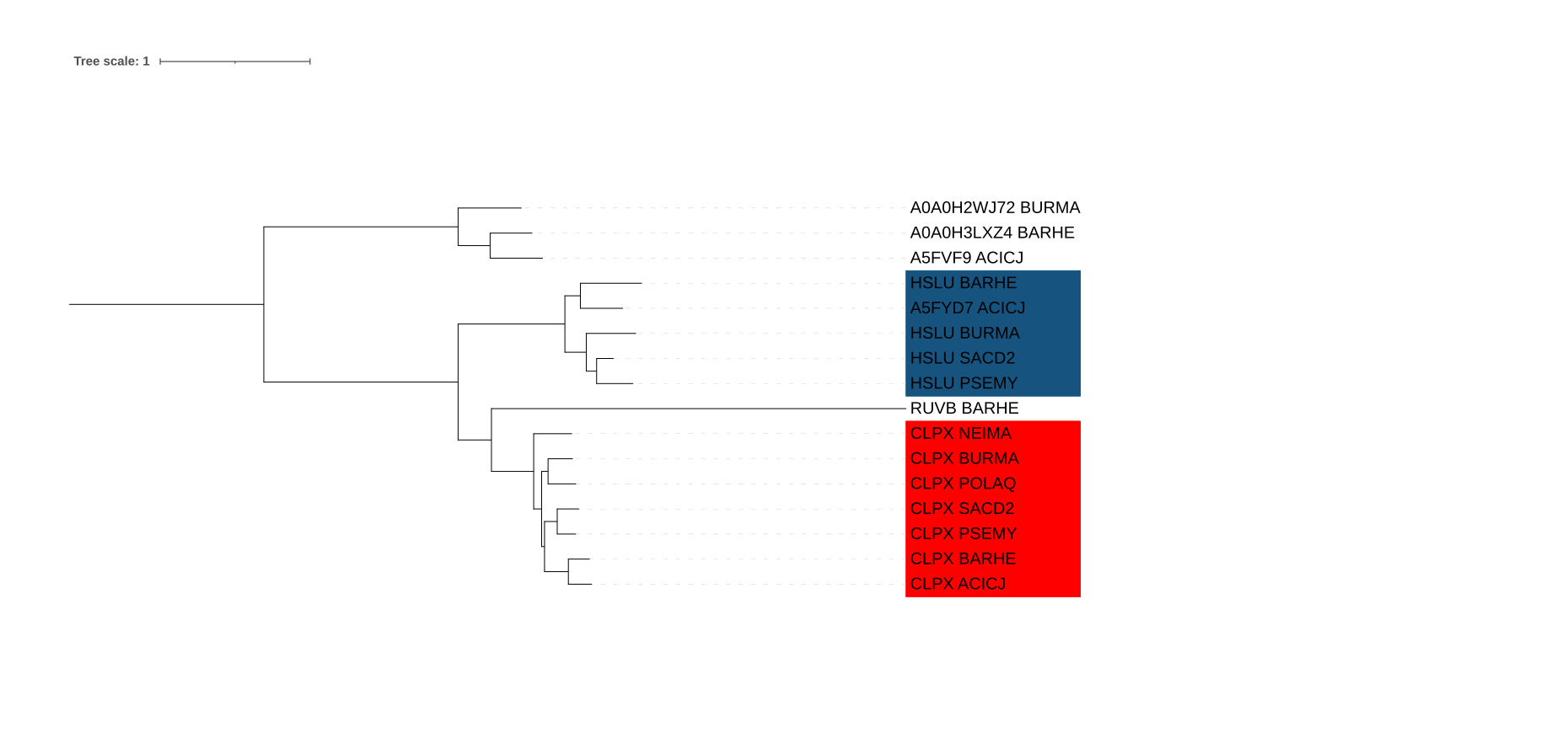
Дерево, где цветами указаны разные ортологические группы
В синей группе не хватает белков из бактерий: NEIMA и POLAQ. В красной есть все.
В красной группе клады (ACICJ, BARHE, NEIMA, POLAQ, BURMA, SACD2, PSEMY) и (SACD2, PSEMY, ACICJ, BARHE) не соотвутсвуют правильной филогении. Вместо них должны быть клады (ACICJ, BARHE) и (NEIMA, POLAQ, BURMA, SACD2, PSEMY). Синяя соответствует филогении.
Дерево со схлопнутыми кладами
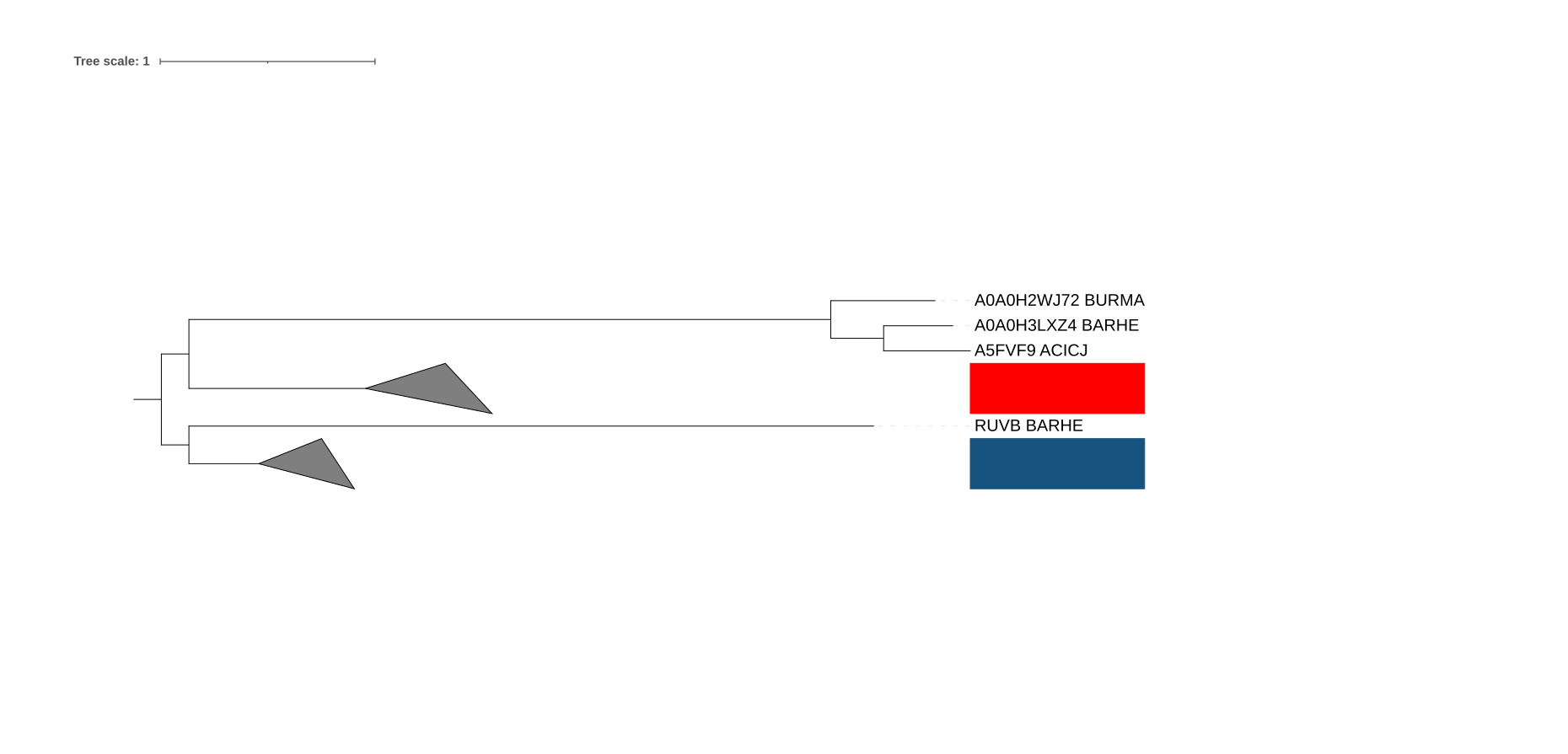
Дерево со схлопнутыми кладами. Красным: HSLU, Синим: CLPX