Задание 1
Были построены модели структур A-, B- и Z-формы ДНК с помощью программы fiber пакета 3DNA. Пакет 3DNA - один из популярных пакетов программ для анализа и простейшего моделирования структур нуклеиновых кислот. Работает под операционной системой LINUX.
Последовательность одной из нитей А- и В- форм дуплекса ДНК представляет собой 5 раз повторенную последовательность "gatc". Последовательность одной из нитей Z- формы представляет собой 10 раз повторенную последовательность "gc".
Полученные PDB - файлы:
A-форма
B-форма
Z-форма
Задание 2
Средства JMol для работы со структурами нуклеиновых кислот.
В программе Jmol, используя выделение разных атомов и химических группировок с помощью предопределенных множеств, было получено следующее изображение структуры А-формы дуплекса ДНК:
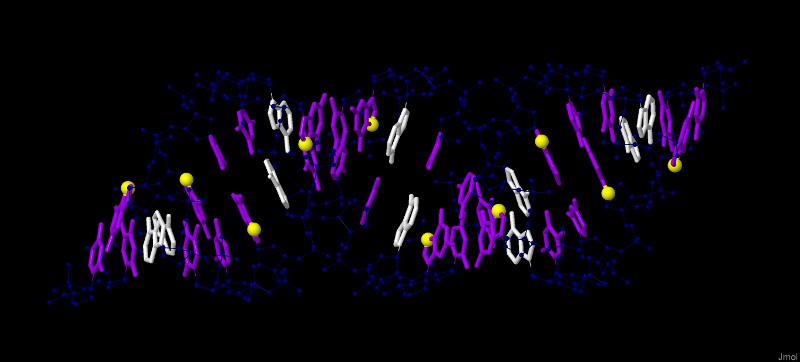
Рис.1. Структура А-формы дуплекса ДНК. Темно-синим выделен сахарофосфатный остов ДНК; все нуклеотиды выделены с помощью wireframe 50;
все аденины обозначены белым;атом N7 во всех гуанинах выглядит как крупная желтая сфера.
Рисунок был получен с помощью скрипта:
cartoons off
wireframe
select backbone
color navy
cpk 50
select not backbone
wireframe 50
select not backbone and A
color white
select [G]*:*.N7
color yellow
cpk 150
На сайте PDB были найдены следующие структуры:
ДНК-белковый комплекс ID:1I3J
тРНК ID:1N78
В обеих структурах разрывов в цепи нуклеиновой кислоты обнаружено не было. Проверка осуществлялась визуально.
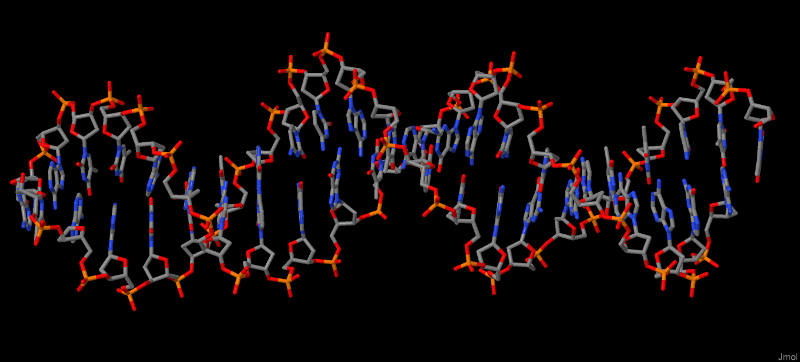
Рис.2. Цепь ДНК из комплекса 1I3J в проволочной модели(wireframe). Атомы окрашены в соответствии с химическим элементом.
Рис.3. Цепь ДНК из комплекса 1I3J в виде лент (ribbons).

Рис.4. Цепи тРНК из комплекса 1N78 в проволочной модели(wireframe). Атомы окрашены в соответствии с химическим элементом.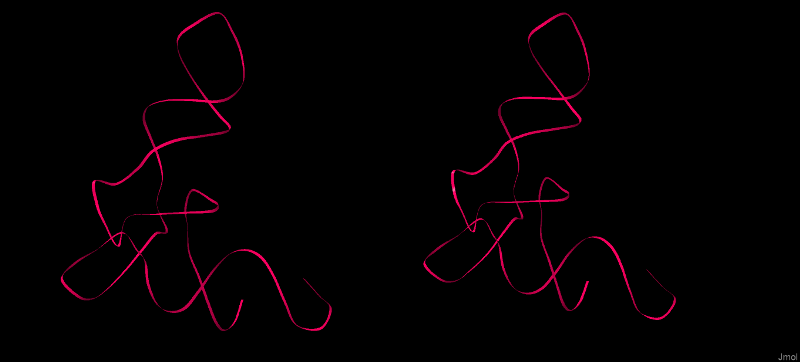
Рис.5. Цепи тРНК из комплекса 1N78 в виде лент (ribbons).
Координаты атомов только ДНК и РНК для дальнейшей работы были сохранены в отдельных файлах:
только ДНК ID:1I3J
только тРНК ID:1N78
Задание 3
Сравнение структур 3-х форм ДНК с помощью средств JMol.
В ранее полученной структуре В-формы двойной спирали ДНК визуально были определены большая и малая бороздки.

Рис.6. Структура В-формы дуплекса ДНК. Цепи представлены в виде лент (ribbons). Гуанины окрашены в соответствии с химическим элементом и представлены в виде соединенных проволокой сфер. Большая и малая бороздки подписаны.
Для иллюстрации взаимного расположения атомов азотистого основания и бороздок был выбран 9-ый по счету гуанин цепи А ([G]9:A) из ранее полученной структуры В-формы двойной спирали ДНК. С помощью ChemSketch было создано его изображение на котором отмечены атомы направленные в сторону большой и малой бороздок.

Рис.7.Структурная формула гуанина с пояснениями о расположении атомов относительно бороздок В-формы дуплекса ДНК.
В случае А- и Z-форм отличий в расположении атомов гуанина относительно бороздок нет.
Было проведено сравнение основных спиральных параметров разных форм ДНК, полученных в 1 задании. Результаты представлены в таблице:
|
A-форма |
B-форма |
Z-форма |
Тип спирали (правая или левая) |
правая |
правая |
левая |
Шаг спирали (Å) |
28,03 |
33,75 |
43,50 |
Число оснований на виток |
11 |
10 |
12 |
Ширина большой бороздки |
7,98 |
17,21 |
7,20 |
Ширина малой бороздки |
16,81 |
11,69 |
15,17 |
Средствами Jmol было проведено сравнение торсионных углов выбранного ранее нуклеотида в структурах А- и В-форм. Для этого использовался следующий скрипт:
restrict [G]9:A or 10:A
color cpk
cartoon off
wireframe
cpk 50
measure ([G]9:A.O1P) ([G]9:A.P) ([G]9:A.O5') ([G]9:A.C5') "alpha %0.1VALUE "
measure ([G]9:A.P) ([G]9:A.O5') ([G]9:A.C5') ([G]9:A.C4') "beta %0.1VALUE"
measure ([G]9:A.O5') ([G]9:A.C5') ([G]9:A.C4') ([G]9:A.C3') "gamma %0.1VALUE"
measure ([G]9:A.C5') ([G]9:A.C4') ([G]9:A.C3') ([G]9:A.O3')"delta %0.1VALUE"
measure ([A]10:A.P) ([G]9:A.O3') ([G]9:A.C3') ([G]9:A.C4') "epsilon %0.1VALUE"
measure ([A]10:A.O5') ([A]10:A.P) ([G]9:A.O3') ([G]9:A.C3') "zeta %0.1VALUE"
measure ([G]9:A.O4') ([G]9:A.C1') ([G]9:A.N9) ([G]9:A.C4) "chi %0.1VALUE"
Полученные результаты занесены в следующую таблицу:
| угол | α | β | γ | δ | ε | ξ | χ |
| A-форма (презентация) | 62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| A-форма (Jmol) | 64.1 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма (презентация) | 63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
| B-форма (Jmol) | 85.9 | 136.4 | 31.1 | 143.4 | -140.8 | -160.5 | -98.0 |
Задание 4
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
| form | alpha | beta | gamma | delta | epsilon | zeta | chi | |
| sum | A | -51,7 | 174,8 | 41,7 | 79,09 | -147,79 | -75,1 | -157,2 |
| sum | B | -29,9 | 136,34 | 31,14 | 143,34 | -140,8 | -160,5 | -98 |
| sum | Z | -43,78 | 21,12 | -61,47 | 116,25 | -100,05 | 8,58 | -47,8 |
| sum | delta AB | 21,8 | 38,46 | 10,56 | 64,25 | 6,99 | 85,40 | 59,21 |
| sum | delta AZ | 7,92 | 153,68 | 103,17 | 37,16 | 47,74 | 83,68 | 109,4 |
| sum | delta BZ | 13,88 | 115,22 | 92,61 | 27,09 | 40,75 | 169,08 | 50,19 |
Были определены значения торсионных углов в заданной структуре тРНК. Результаты представлены в таблице:
| form | alpha | beta | gamma | delta | epsilon | zeta | chi | |
| RNA | sum | -30,97 | 58,22 | 55,63 | 87,43 | -128,37 | -56,40 | -146,85 |
| A | sum | -51,7 | 174,8 | 41,7 | 79,09 | -147,79 | -75,1 | -157,2 |
| B | sum | -29,9 | 136,34 | 31,14 | 143,34 | -140,8 | -160,5 | -98 |
| Z | sum | -43,78 | 21,12 | -61,47 | 116,25 | -100,05 | 8,58 | -47,8 |
Также были определены торсионные углы в заданной структуре ДНК; с помощью Excel было подсчитано среднее значение каждого из торсионных углов (краевые нуклеотиды не рассматривались). Результаты представлены в таблице:
| base | alpha | beta | gamma | delta | epsilon | zeta | chi |
| sum | -24,59 | -18,88 | 10,44 | 148,78 | -84,59 | -116,18 | -98,05 |
| № | base | |
| 1 | T | |
| 2 | T | 368,0789 |
| 3 | C | 478,6211 |
| 4 | T | 247,3158 |
| 5 | T | 356,5947 |
| 6 | G | 392,4474 |
| 7 | G | 347,1158 |
| 8 | G | 408,7789 |
| 9 | T | 472,9895 |
| 10 | C | 644,9368 |
| 11 | T | 293,9105 |
| 12 | A | 304,5053 |
| 13 | C | 368,4947 |
| 14 | C | 393,1 |
| 15 | G | 473,4947 |
| 16 | T | 294,8526 |
| 17 | T | 569,5842 |
| 18 | T | 338,3789 |
| 19 | A | 530,7579 |
| 20 | A | 259,6368 |
| 21 | T | |
| 22 | A | |
| 23 | A | 358,1474 |
| 24 | T | 356,3684 |
| 25 | T | 324,1158 |
| 26 | A | 526,1579 |
| 27 | A | 464,7632 |
| 28 | A | 299,2474 |
| 29 | C | 387,3 |
| 30 | G | 468,2632 |
| 31 | G | 575,2842 |
| 32 | T | 295,9526 |
| 33 | A | 347,9053 |
| 34 | G | 334,2789 |
| 35 | A | 317,8211 |
| 36 | C | 453,3684 |
| 37 | C | 268,3105 |
| 38 | C | 348,1158 |
| 39 | A | 348,3158 |
| 40 | A | 349,1474 |
| 41 | G | 342,3105 |
| 42 | A |
Все вычисления проводились с помощью программы Excel. Файл с вычислениями можно посмотреть здесь
Были определены номера нуклеотидов, образующих стебли(stems) во вторичной структуре заданной тРНК. Стебли образуют нуклеотиды с номерами 501-507 и 572-566, 549-553 и 561-565, 538-544 и 532-526, 510-513 и 525-522.
Неканонические пары в структуре:
C:.502_:[..G]G-*---U[..U]:.571_:C C:.555_:[..U]Ux**+xG[..G]:.518_:C C:.544_:[..A]Ax*---G[..G]:.526_:C C:.538_:[..A]A-*---C[..C]:.532_:C C:.513_:[..U]U-*--xG[..G]:.522_:CВодородные связи между основаниями, которые не образуют стебли, необходимы для поддержания структуры тРНК.
C:.554_:[..U]U-**-xA[..A]:.558_:C C:.514_:[..A]A-**-xU[..U]:.508_:C C:.515_:[..G]Gx**+xC[..C]:.548_:C C:.519_:[..G]Gx---xC[..C]:.556_:C
Рис.8. Схема водородных связей в заданной структуре тРНК. Цветом выделены стебли.
Были найдены 2 последовательных пары азотистых оснований с наибольшими значениями площади "перекрывания":
Рис.9. Перекрывание пар азотистых оснований. Серым выделено наибольшее из них
Изображение перекрывания этих пар получено спомощью следующих комманд:
ex_str -2 stacking.pdb step2.pdb stack2img -cdolt step2.pdb step2.ps convert step2.ps fig_17.pngПолученное изображение приведено ниже:
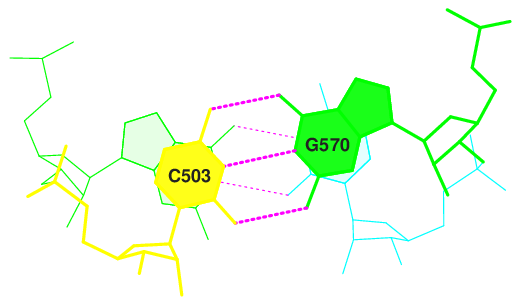
Рис.10. Пары азотистых оснований с наибольшим перекрыванием.
Взаимная ориентация оснований была проверена с помощью JMol:
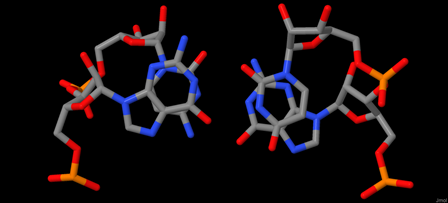
Рис.11. Пары азотистых оснований с наибольшим перекрыванием.