Комплексы ДНК-белок
Поиск ДНК-белковых контактов в заданной структуре 1I3J
C помощью команды define JMol были заданы множества атомов:
множество атомов кислорода 2'-дезоксирибозы (set1).
множество атомов кислорода в остатке фосфорной кислоты (set2).
множество атомов азота в азотистых основаниях (set3).
Для этого был написан следующий скрипт:
define set1 (*.O?')
define set2 (*.OP?)
define set3 (*.N? and DNA)
Был создан скрипт, вызов которого в JMol даст последовательное (с паузами!) изображение всей структуры, только ДНК в проволочной модели, той же модели, но с выделенными шариками множеством атомов set1, затем set2 и set3:
cartoons off
restrict DNA
color cpk
wireframe 50
define set1 (*.O?')
define set2 (*.OP?)
define set3 (*.N? and DNA)
pause
select set1
cpk 150
pause
cpk off
select set2
cpk 150
pause
cpk off
select set3
cpk 150
Ниже представлены полученные изображения:
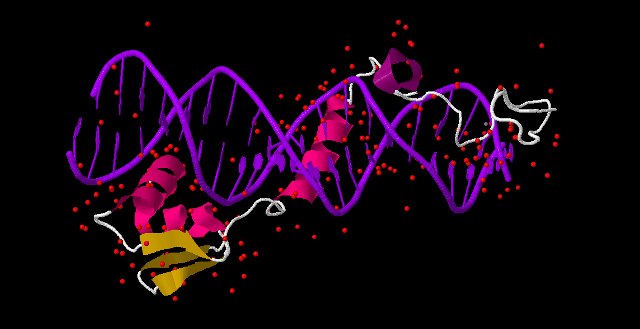
Рис.1. Изображение всей структуры.
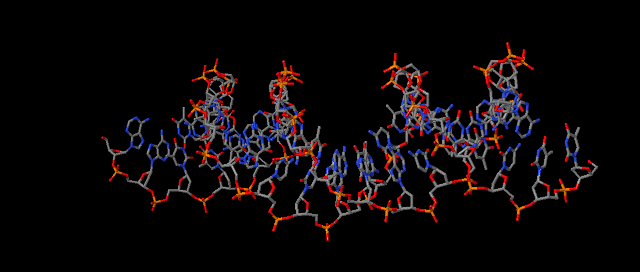
Рис.2. Только ДНК в проволочной модели.
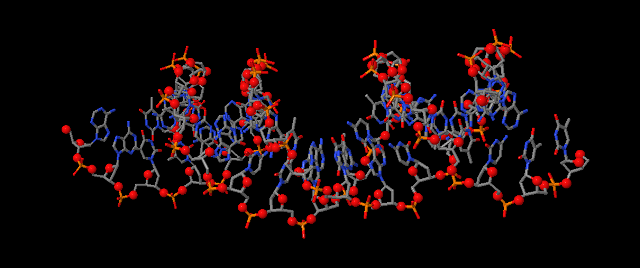
Рис.3. Та же модель, но с выделенными шариками множеством атомов set1.
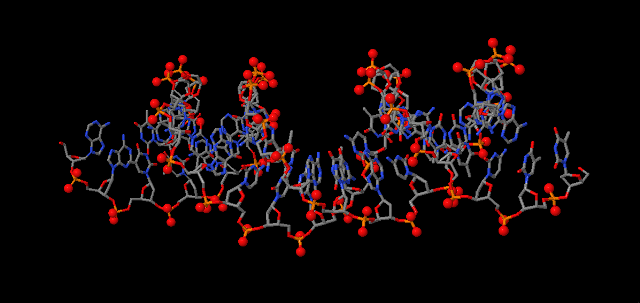
Рис.4. Та же модель, но с выделенными шариками множеством атомов set2.

Рис.5. Та же модель, но с выделенными шариками множеством атомов set3.
Опишем ДНК-белковые контакты в заданной структуре и сравним количество контактов разной природы. Будем считать полярными атомы кислорода и азота, а неполярными атомы углерода, фосфора и серы. Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5Å. Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5Å.
Для поиска ДНК-белковых контактов был написан следующий скрипт:
cartoons off
restrict DNA
color cpk
wireframe 50
define set1 (*.O?')
define set2 (*.OP?)
define set3 (*.N? and DNA)
define set4 (*.C?')
define set5 (*.P)
define set6 (*.O? and DNA)
define set7 (((*.C6 or *.N6 or *.C5 or *.N7 or *.C5) and A) or ((*.C6 or *.O6 or *.C5 or *.N7 or *.C8) and G) or ((*.C6 or *.C5 or *.C4 or *.N4) and C) or ((*.C6 or *.C5 or *.C4 or *.O4 or *.C7) and T))
define set8 (((*.C2 or *.N3 or *.C4 or *.N9) and A) or ((*.C2 or *.N2 or *.N3 or *.C4 or *.N9) and G) or ((*.O2 or *.N1 or *.C2) and C) or ((*.O2 or *.C2 or*.N1 or *.C6) and T))
select within (3.5, set1) and (*.N or *A.O)
select within (4.5, set4) and (*.S or *A.P or *A.C)
select within (3.5, set2) and (*.N or *A.O)
select within (4.5, set5) and (*.S or *A.P or *A.C)
select within (3.5, (set7 and (set6 or set3))) and (*.N or *A.O)
select within (4.5, (set7 and (not (set6 or set3)))) and (*.S or *A.P or *A.C)
select within (3.5, (set8 and (set6 or set3))) and (*.N or *A.O)
select within (4.5, (set8 and (not (set6 or set3)))) and (*.S or *A.P or *A.C)
В котором определяются множества:
множество атомов кислорода 2'-дезоксирибозы (set1).
множество атомов кислорода в остатке фосфорной кислоты (set2).
множество атомов азота в азотистых основаниях (set3).
множество атомов углерода 2'-дезоксирибозы (set4).
множество атомов фосфора в остатке фосфорной кислоты (set5).
множество атомов кислорода в азотистых основаниях (set6).
множество атомов азотистых оснований обращенных в сторону большой бороздки(set7).
множество атомов азотистых оснований обращенных в сторону малой бороздки(set8).
Информация об атомах, обращенных в сторону той или иной бороздки взята из презентации.
По результатам скрипта составим таблицу:
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 4 | 13 | 17 |
| остатками фосфорной кислоты | 14 | 2 | 16 |
| остатками азотистых оснований со стороны большой бороздки | 0 | 2 | 2 |
| остатками азотистых оснований со стороны малой бороздки | 3 | 0 | 3 |
remediator --old --pdb 1I3J_without_water.pdb >old_1I3J_without_water.pdb
nucplot old_1I3J_without_water.pdb
convert nucplot.ps nucplot.png
Полученная схема приведена ниже:
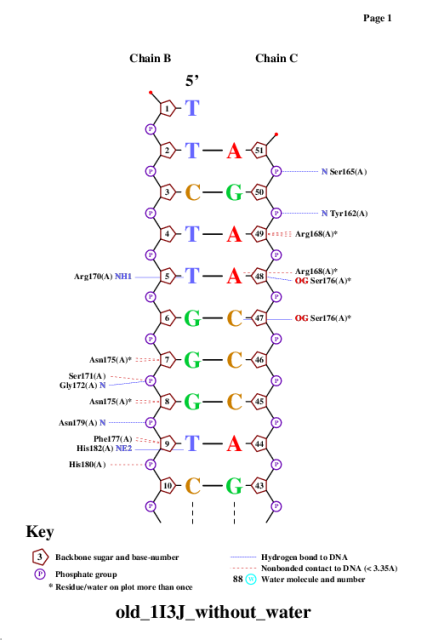
Рис.6. Популярная схема ДНК-белковых контактов страница 1.
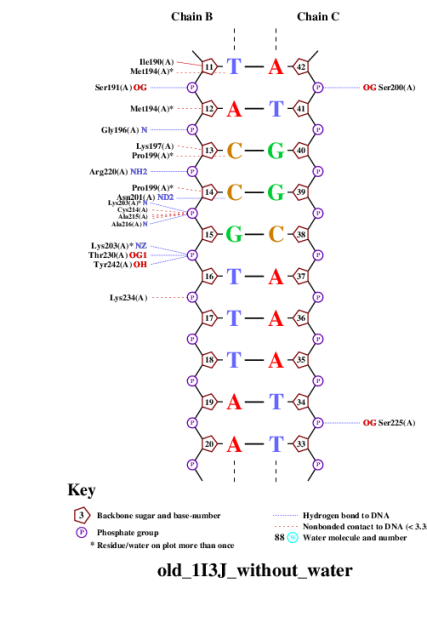
Рис.7. Популярная схема ДНК-белковых контактов страница 2.

Рис.8. Популярная схема ДНК-белковых контактов страница 3.
Наибольшее число указанных на схеме контактов с ДНК равно 2. Два контакта с ДНК имеют аминокислотные остатки с номерами:
168
175
176
194
199
203
Вероятно, в распознавании ДНК участвуют несколько остатков. Рассмотрим остатки 168Arg и 199Pro, посколку они оба имеют по два контакта с ДНК, один из которых непосредственно с азотистым основанием.
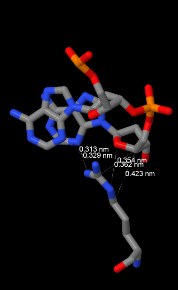
Рис.9. Контакты Arg168 c A48 и A49. Атомы раскрашены в соответствии с химическим элементом
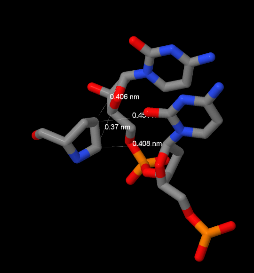
Рис.10. Контакты Pro199 c C13 и C14. Атомы раскрашены в соответствии с химическим элементом
Предсказание вторичной структуры заданной тРНК
Для предсказания вторичной структуры тРНК 1NV8 путем поиска инвертированных повторов использавалась программа einverted со следующими параметрами:Gap penalty [12]: Minimum score threshold [50]: 5 Match score [3]: Mismatch score [-4]:При других параметрах программа либо не находила ничего, либо предсказывала множество коротких неверных повторов.
Также было проделано предсказание вторичной структуры тРНК по алгоритму Зукера. Ниже приведена картинка с лучшим результатом:
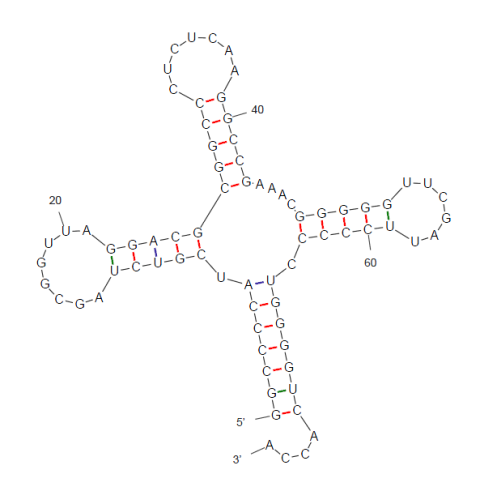
Рис.11. Лучшее предсказание вторичной структуры тРНК полученное по алгоритму Зукера.
В таблице ниже представлено сравнение реальной структуры с полученными предсказаниями.
| Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера | |
| Акцепторный стебель | 5'-501-507-3' 5'566-572-3' Всего 7 пар |
5'-903-907-3' 5'-965-969-3' предсказано 5 пар все неверны(сдвиг на один нуклеотид) |
Найдено верно все 7 пар |
| D-стебель | 5'-510-513-3' 5'-522-525-3' Всего 4 пары |
Не найдено | Найдены верно все 4 пары+1лишняя |
| T-стебель | 5'-549-553-3' 5'-561-565-3' Всего 5 пар |
Не найдено | Найдено 5 пар все неверны |
| Антикодоновый стебель | 5'-538-544-3' 5'-526-532-3' Всего 7 пар |
Не найдено | Найдено 5 пар из 7 |
| Общее число канонических пар нуклеотидов | 19 | 5 всего 0 верных | 19 всего 14 верных |