Реконструкция деревьев по нуклеотидным последовательностям. Деревья. содержащие паралоги
Построение дерева по нуклеотидным последовательностям
Для работы были найдены характеристики РНК малой субъединицы рибосомы (16S rRNA) каждой из исследуемых бактерий.
| № | Название | Мнемоника | AC EMBLE | Координаты | Цепь |
| 1 | Bacillus subtilis | BACSU | AF008220 | 485-2034 | Прямая |
| 2 | Clostridium tetani | CLOTE | AE015927 | 8715-10223 | Прямая |
| 3 | Finegoldia magna | FINM2 | AP008971 | 611796-613319 | Прямая |
| 4 | Lactobacillus acidophilus | LACAC | CP002559 | 447399..448973 | Прямая |
| 5 | Lactococcus lactis | LACLM | AM406671 | 511423..512971 | Прямая |
| 6 | Staphylococcus aureus | STAA1 | AP009324 | 531922:533476 | Прямая |
| 7 | Streptococcus pyogenes | STRP1 | AE004092 | 17170..18504 | Прямая |
Последовательности РНК малой субъединицы рибосомы (16S rRNA) были получены из соответствующих записей EMBLE с помощью команды seqret пакета EMBOSS. После чего было построено их выравнивание программой MUSCLE. В программе MEGA с помощью метода Max likelihood было реконструировано филогенетическое дерево.
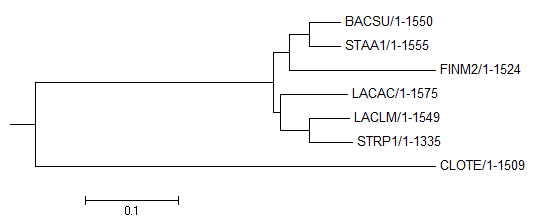
Нетривиальные ветви идеального дерева
{'BACSU', 'STAA1'}vs{'LACLM', 'LACAC', 'STRP1', 'CLOTE', 'FINM2'}
{'LACLM', 'STRP1'}vs{'LACAC', 'BACSU', 'CLOTE', 'STAA1', 'FINM2'}
{'CLOTE', 'FINM2'}vs{'LACLM', 'LACAC', 'BACSU', 'STAA1', 'STRP1'}
{'LACAC', 'LACLM', 'STRP1'}vs{'BACSU', 'CLOTE', 'STAA1', 'FINM2'}
Нетривиальные ветви реконструированного дерева
{'BACSU', 'FINM2', 'STAA1'}vs{'LACLM', 'LACAC', 'CLOTE', 'STRP1'}
{'BACSU', 'STAA1'}vs{'LACLM', 'LACAC', 'STRP1', 'CLOTE', 'FINM2'}
{'LACLM', 'STRP1'}vs{'LACAC', 'BACSU', 'CLOTE', 'STAA1', 'FINM2'}
{'LACAC', 'LACLM', 'STRP1'}vs{'BACSU', 'CLOTE', 'STAA1', 'FINM2'}
Ветви которые есть в идеальном дереве, но отсутствуют в реконструированном
{'CLOTE', 'FINM2'}vs{'LACLM', 'LACAC', 'BACSU', 'STAA1', 'STRP1'}
Ветви которые есть в реконструированном дереве, но отсутствуют в идеальном
{'BACSU', 'FINM2', 'STAA1'}vs{'LACLM', 'LACAC', 'CLOTE', 'STRP1'}
Построение и анализ дерева, содержащего паралоги
Был осуществлен поиск программой blastp гомологов (с порогом E-value 0,001). Из результатов поиска были отобраны по мнемонике видов только те находки, которые относятся к отобранным ранее бактериям. Затем по полученному программой MUSCLE выравниванию было построено дерево в программе MEGA с помощью метода Max likelihood.
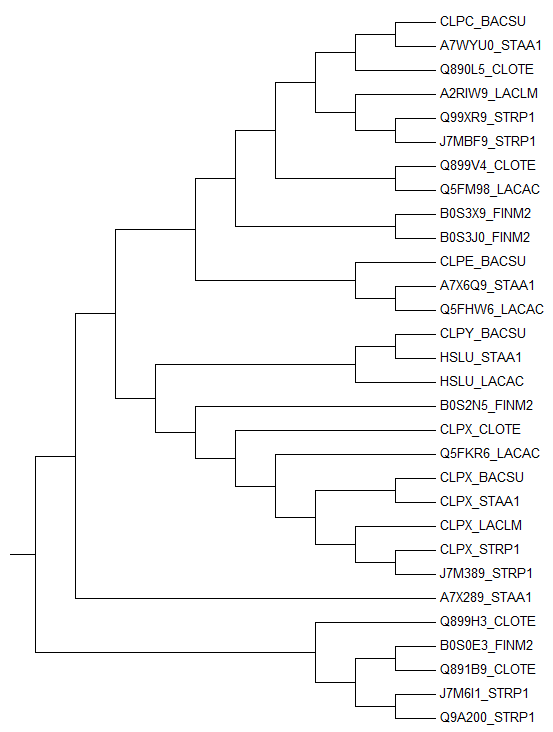
Примеры:
Паралоги : CLPC_BACSU и CLPX_BACSU; A2RIW9_LACLM и CLPX_LACLM. Ортологи :CLPX_BACSU и CLPX_STAA1; BS0E3_FINM2 и Q891B9_CLOTE.