Реконструкция филогенетических деревьев
Выбранные в рамках занятия 1 бактерии принадлежат следующим таксонам:
| № | Название | Мнемоника | Класс | Порядок | Семейство |
| 1 | Bacillus subtilis | BACSU | Bacilli | Bacillales | Bacillaceae |
| 2 | Clostridium tetani | CLOTE | Bacilli | Lactobacillales | Streptococcaceae |
| 3 | Finegoldia magna | FINM2 | Clostridia | Clostridiales | Clostridiales Family XI. Incertae Sedis |
| 4 | Lactobacillus acidophilus | LACAC | Bacilli | Lactobacillales | Lactobacillaceae |
| 5 | Lactococcus lactis | LACLM | Bacilli | Lactobacillales | Streptococcaceae |
| 6 | Staphylococcus aureus | STAA1 | Bacilli | Bacillales | Staphylococcaceae |
| 7 | Streptococcus pyogenes | STRP1 | Bacilli | Lactobacillales | Streptococcaceae |
Скобочная формула:
(( Clostridiaceae_Clostridium_tetani ,Clostridiales_Family_XI._Incertae Sedis-Finegoldia_magna)Clostridia_ Clostridiales,((Lactobacillaceae_Lactobacillus_acidophilus,(Lactococcus_lactis, Streptococcus_pyogenes )Streptococcaceae)Lactobacillales,(Staphylococcaceae_Staphylococcus_aureus ,Bacillaceae_Bacillus_ subtilis)Bacillales)Bacilli)Firmicutes;
Изображение:
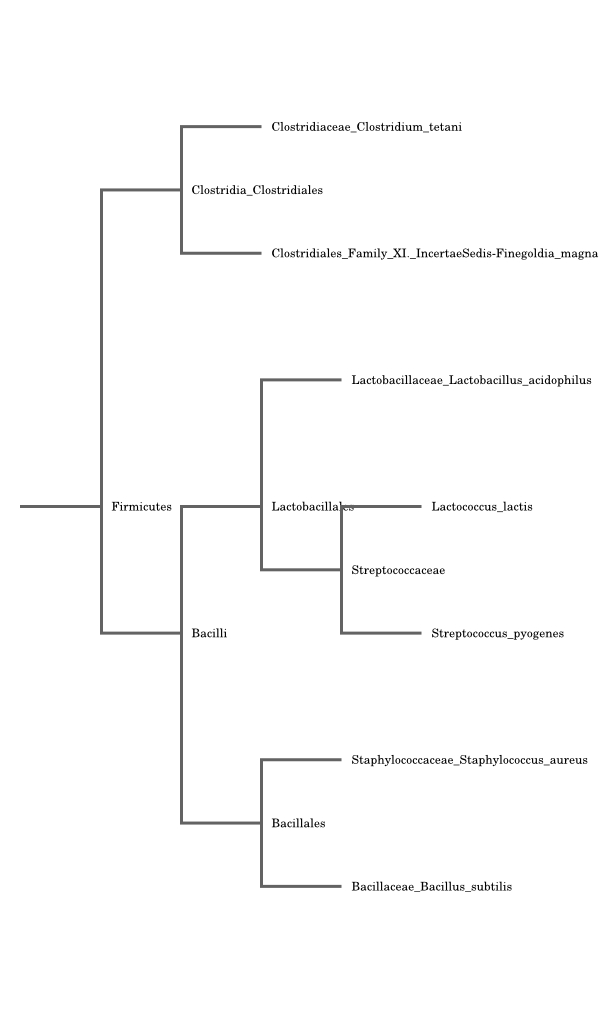
Выбранные бактерии принадлежат двум различным классам и трем порядкам.
Для реконструкции филогенетического дерева был выбран Рибосомный белок L3. Его мнемоника: RL3
С помощью команды seqret пакета EMBOSS из Swiss-Prot были получены последовательности белков с данной функцией из отобранных ранее бактерий.
Последовательности были пощены в один fasta файл. С помощью программы MUSCLE было построено выравнивание выбранных белков.
Выравнивание было импортировано в JalView. 
Рис. 1. Выравнивание рибосомальных белков L3 из отобранных бактерий, в блочной форме, с раскраской по проценту идентичности.
Четырьмя методами, доступными из JalView было реконструировано филогенетическое дерево.
.jpg)
Идеальное дерево
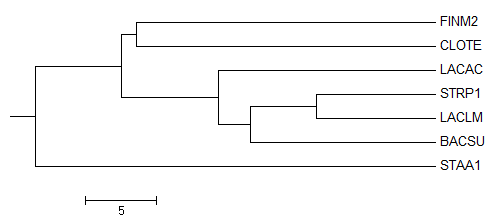
Average Distance Using %Identity.
Нетривиальные ветви идеального дерева
{'BACSU', 'LACAC', 'LACLM', 'STAA1', 'STRP1'}vs{'CLOTE', 'FINM2'}
{'BACSU', 'STAA1'}vs{'LACLM', 'LACAC', 'STRP1', 'CLOTE', 'FINM2'}
{'LACLM', 'STRP1'}vs{'LACAC', 'BACSU', 'CLOTE', 'STAA1', 'FINM2'}
{'CLOTE', 'FINM2'}vs{'LACLM', 'LACAC', 'BACSU', 'STAA1', 'STRP1'}
{'LACAC', 'LACLM', 'STRP1'}vs{'BACSU', 'CLOTE', 'STAA1', 'FINM2'}
Нетривиальные ветви дерева Average Distance Using %Identity.
{'BACSU', 'LACAC', 'LACLM', 'STRP1'}vs{'CLOTE', 'STAA1', 'FINM2'}
{'BACSU', 'LACLM', 'STRP1'}vs{'LACAC', 'CLOTE', 'STAA1', 'FINM2'}
{'LACLM', 'STRP1'}vs{'LACAC', 'BACSU', 'CLOTE', 'STAA1', 'FINM2'}
{'CLOTE', 'FINM2'}vs{'LACLM', 'LACAC', 'BACSU', 'STAA1', 'STRP1'}
{'BACSU', 'CLOTE', 'FINM2', 'LACAC', 'LACLM', 'STRP1'}vs{'STAA1',}
Ветви которые есть в Первом дереве, но отсутствуют в Average Distance Using %Identity дереве.
{'LACAC', 'LACLM', 'STRP1'}vs{'BACSU', 'CLOTE', 'STAA1', 'FINM2'}
{'BACSU', 'LACAC', 'LACLM', 'STAA1', 'STRP1'}vs{'CLOTE', 'FINM2'}
{'BACSU', 'STAA1'}vs{'LACLM', 'LACAC', 'STRP1', 'CLOTE', 'FINM2'}
Ветви которые есть в Average Distance Using %Identity дереве, но отсутствуют в Первом
{'BACSU', 'LACLM', 'STRP1'}vs{'LACAC', 'CLOTE', 'STAA1', 'FINM2'}
{'BACSU', 'LACAC', 'LACLM', 'STRP1'}vs{'CLOTE', 'STAA1', 'FINM2'}
{'BACSU', 'CLOTE', 'FINM2', 'LACAC', 'LACLM', 'STRP1'}vs{'STAA1',}
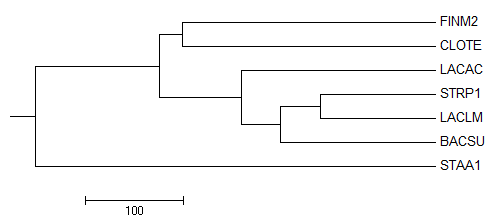
Average Distance Using BLOSUM62 такое же как предыдущее
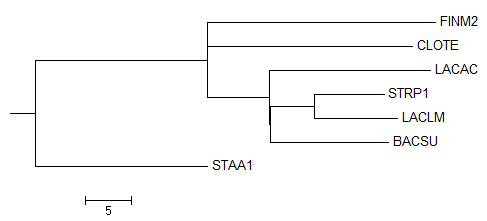
Neighbour Joining Using %Identity
Нетривиальные ветви идеального дерева
{'BACSU', 'LACAC', 'LACLM', 'STAA1', 'STRP1'}vs{'CLOTE', 'FINM2'}
{'BACSU', 'STAA1'}vs{'LACLM', 'LACAC', 'STRP1', 'CLOTE', 'FINM2'}
{'LACLM', 'STRP1'}vs{'LACAC', 'BACSU', 'CLOTE', 'STAA1', 'FINM2'}
{'CLOTE', 'FINM2'}vs{'LACLM', 'LACAC', 'BACSU', 'STAA1', 'STRP1'}
{'LACAC', 'LACLM', 'STRP1'}vs{'BACSU', 'CLOTE', 'STAA1', 'FINM2'}
Нетривиальные ветви дерева Neighbour Joining Using %Identity
{'BACSU', 'LACAC', 'LACLM', 'STRP1'}vs{'CLOTE', 'STAA1', 'FINM2'}
{'BACSU', 'CLOTE', 'LACAC', 'LACLM', 'STRP1'}vs{'STAA1', 'FINM2'}
{'BACSU', 'LACLM', 'STRP1'}vs{'LACAC', 'CLOTE', 'STAA1', 'FINM2'}
{'LACLM', 'STRP1'}vs{'LACAC', 'BACSU', 'CLOTE', 'STAA1', 'FINM2'}
{'BACSU', 'CLOTE', 'FINM2', 'LACAC', 'LACLM', 'STRP1'}vs{'STAA1',}
Ветви которые есть в идеальном дереве, но отсутствуют в Neighbour Joining Using %Identity
{'LACAC', 'LACLM', 'STRP1'}vs{'BACSU', 'CLOTE', 'STAA1', 'FINM2'}
{'CLOTE', 'FINM2'}vs{'LACLM', 'LACAC', 'BACSU', 'STAA1', 'STRP1'}
{'BACSU', 'LACAC', 'LACLM', 'STAA1', 'STRP1'}vs{'CLOTE', 'FINM2'}
{'BACSU', 'STAA1'}vs{'LACLM', 'LACAC', 'STRP1', 'CLOTE', 'FINM2'}
Ветви которые есть в Neighbour Joining Using %Identity дереве, но отсутствуют в идеальном
{'BACSU', 'LACLM', 'STRP1'}vs{'LACAC', 'CLOTE', 'STAA1', 'FINM2'}
{'BACSU', 'LACAC', 'LACLM', 'STRP1'}vs{'CLOTE', 'STAA1', 'FINM2'}
{'BACSU', 'CLOTE', 'LACAC', 'LACLM', 'STRP1'}vs{'STAA1', 'FINM2'}
{'BACSU', 'CLOTE', 'FINM2', 'LACAC', 'LACLM', 'STRP1'}vs{'STAA1',}
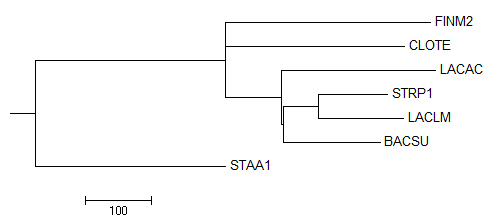
Neighbour Joining Using BLOSUM62 такое же как предыдущее
В программе Mega было реконструируировано дерево методом "Maximum Parsimony" и укоренено в наиболее правильную ветвь.
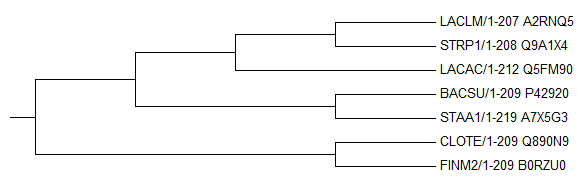
Maximum Parsimony
Дерево полностью совпало с правильным.