
Для выполнения данного задания я взяла грамотрицательную бактерию Campylobacter coli,
с которой я работала в первом семестре, и её близкого "родственника" - очень распространённую
патогенную бактерию Campylobacter jejuni. Поиск производился в банке "Nucleotides" NCBI,
с указанием "chromosome" в графе "Title".
Campylobacter coli: NZ_CP046317.1 (на графиках - по оси X)
Campylobacter jejuni: NZ_CP136638.1 (на графиках - по оси Y)
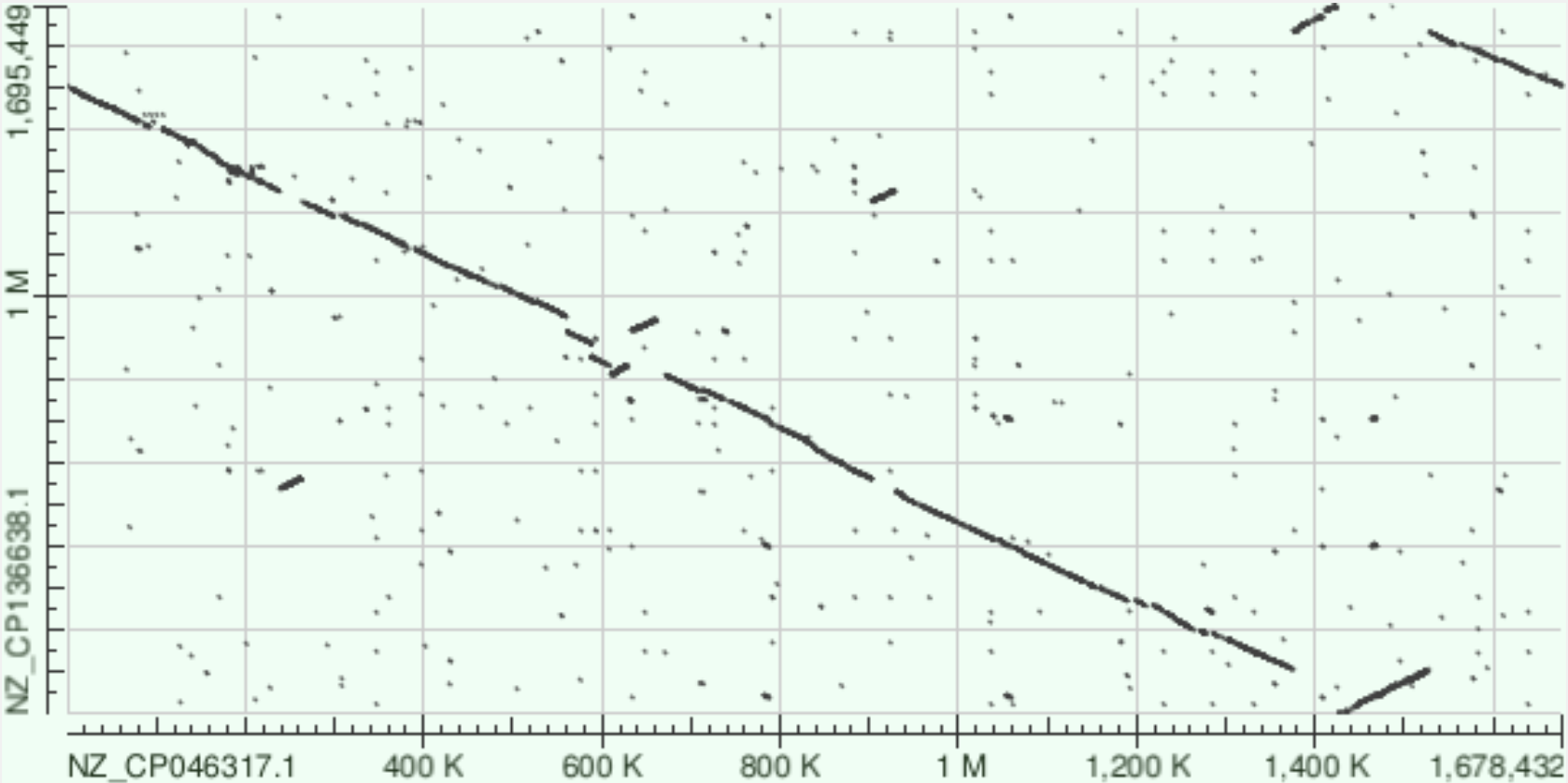
Для получения карты локального сходства был использован blastn с подключением алгоритма megablast. Получившийся Dot Plot прикреплён ниже:

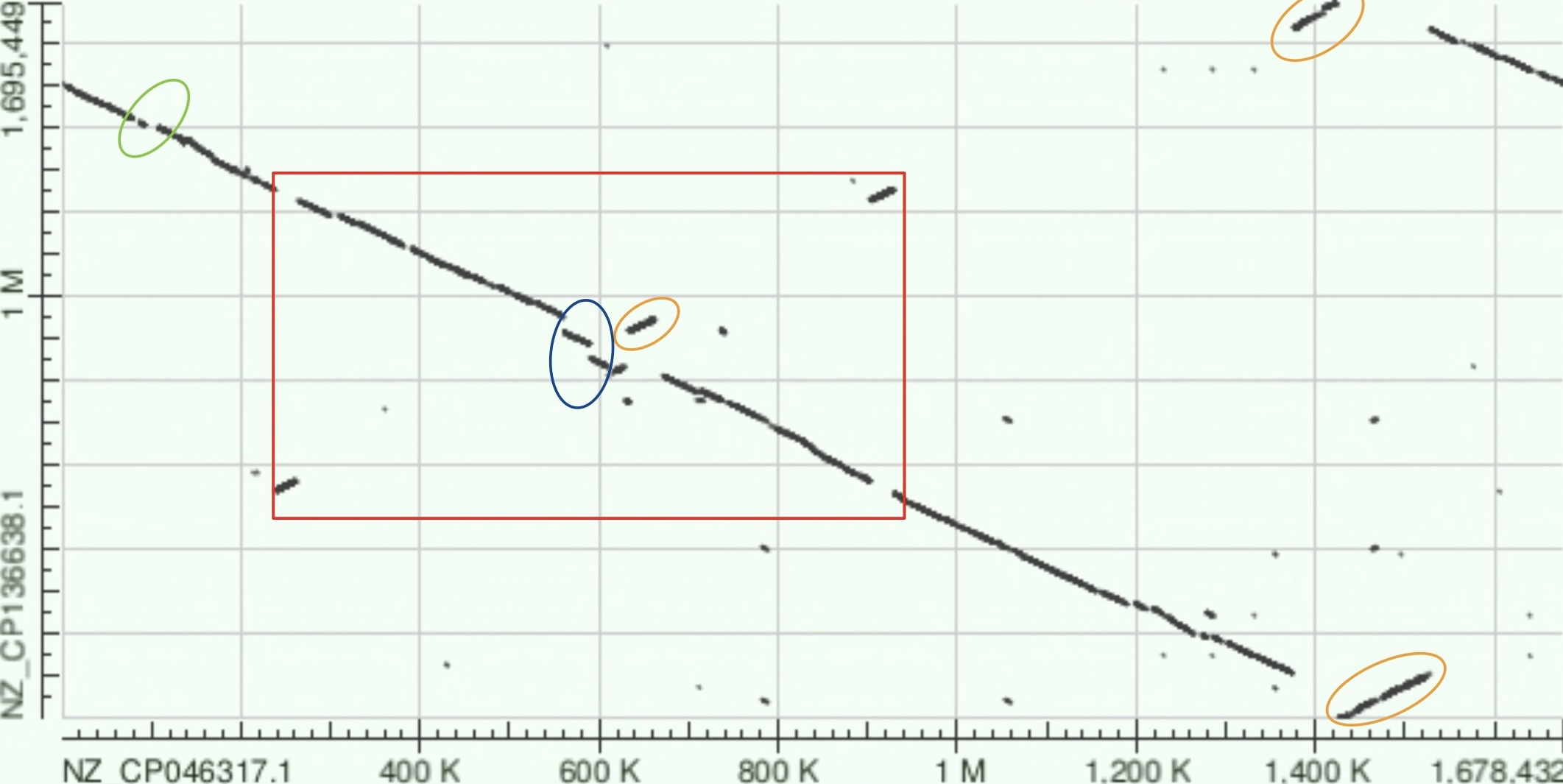
[1] Зелёным цветом отмечен участок, в котором, вероятно, произошла делеция или инсерция. На это указывает характерный "разрыв" прямых.
[2] Оранжевым цветом отмечены инвертированные участки (комплементарные обратной цепи другой последовательности).
[3] Синим цветом обведена транслокация/релокация участка.
[4] Красным обведён участок, в котором, как мне кажется, произошла серия инверсий (скорее всего, их было две; на это указывают короткие участки по углам красного бокса).
Иными словами, после первой инверсии ориентация участка была изменена, после второй - восстановлена, но не полностью.
Ниже приведён Dot Plot, построенный blastn (без подключения megablast). Как можно увидеть, результаты очень похожи, однако в данном случае программа выявила множество участков локальных сходств в последовательностях (разбросанные точки). Получается, blastn оказался чувствительнее.