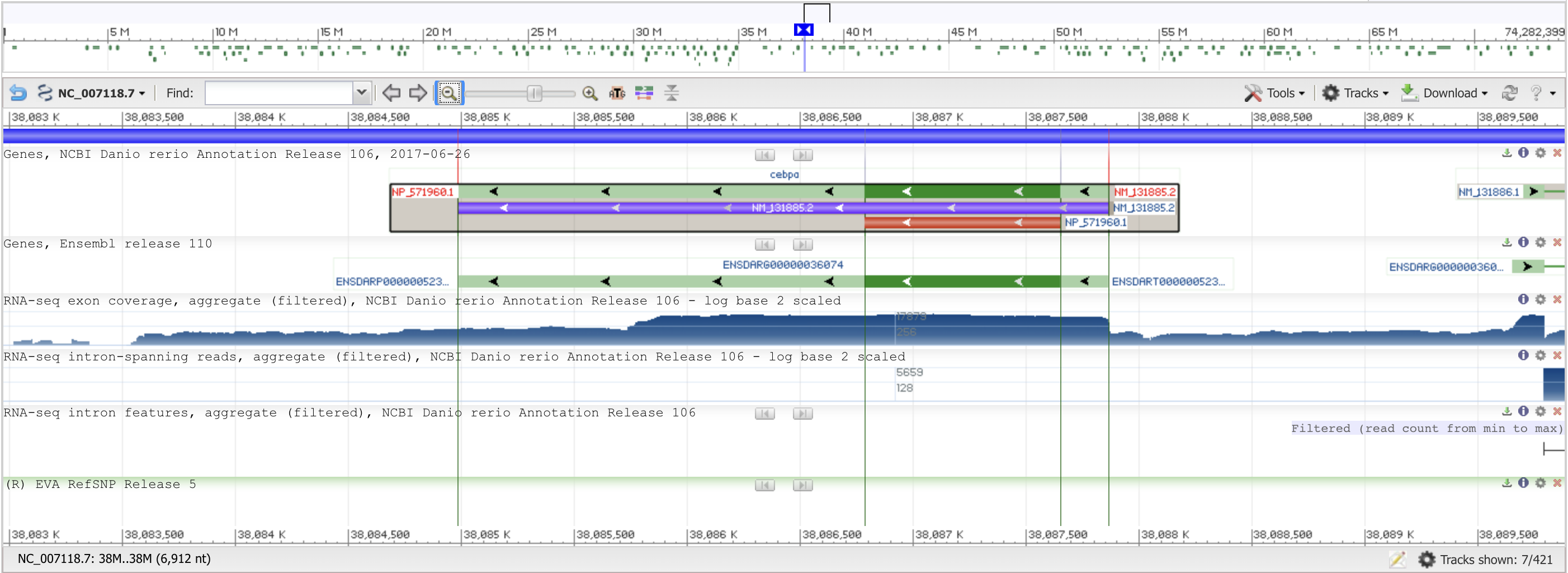

Я выбрала контиг с идентификатором NC_007118.7.
NC_007118.7:
Danio rerio strain Tuebingen chromosome 7
GRCz11 Primary Assembly
[38,084,698 - 38,088,153]
3,456 bp, positive strand
gene - cebra.

Зелёным на фотографии отмечен сам ген (один, cebra), светло-зелёным - 5'- и 3'-нетранслируемые
области, фиолетовым - соответствующая мРНК (одна). Красным выделена кодирующая последовательность (одна, CDS),
длиной в 867 bp.
Fasta файл можно найти здесь.
Для поиска BLAST был выбран класс Птиц (Aves, taxid:8782).
blastn
Длина слова - 11, expect treshhold (далее сохраняется везде) - 0.05, количество находок - 349/1000 (файл тут). Средний процент покрытия по всем находкам - 15%, значения e-value соответствуют
ожидаемым (очень маленькие числа, что хорошо). Отмечу, что часть находок - также сборки организмов (птиц). Также много предсказанных структур.
Для чего используется: определение принадлежности организма к тому или иному таксону, сравнивание неблизкородственных организмов.
megablast
Длина слова - 28, количество находок - 100/1000 (файл тут). Среди находок -
различные сборки организмов, отдельные хромосомы (в основном, различных птиц), однако большинство найденных записей - белок CCAAT (CEBPA).
Для чего используется: быстрый поиск среди схожих последовательностей; можно использовать, когда мы уверены,
что последовательность найдётся в каком-то конкретном организме.
blastx
Длина слова - 5, количество находок - 821/1000 (файл тут). В отличие от находок blastn и megablast - предсказанных последовательностей единицы. Среди находок - также белок CCAAT (CEBPA), но есть и IHE44, белок, агрегирующий с образованием микротрубчатых структур (также он инфицируется в печени зараженных вирусом гепатита).
Для чего используется: определение кодирующих последовательносте/аннотация последовательностей; последовательность
переводится из нуклеотидной в аминокислотную и поиск производится по белковой базе данных.
tblastx
Длина слова - 3. Я пробовала запустить программу со всевозможными комбинациями параметров, и, к сожалению, во всех
случаях она выдавала ошибку (превышение лимита использования CPU).
Для чего используется: предсказание генов в последовательностях; ищем гомолога в геномах, в которых еще даже не предсказаны гены.
Последовательность генома Danio rerio была проиндексирована командой:
makeblastdb -in cebra.fna -dbtype nucl
О рРНК:
23S рРНК входит в каталитический пептидилтрансферазный центр на рибосоме,
16S рРНК необходима для правильной посадки 30S субчастицы
рибосомы на мРНК - чтобы Р-сайт рибосомы размещался точно на инициирующем кодоне АУГ на мРНК.
Также 16S рРНК овтечает для связывание с последовательностью Шайна-Дальгарно.
Так как поиск производится по нуклеотидным последовательностям, я выбрала blastn. Были использованы следующие команды:
blastn -task blastn -evalue 0.05 -query ecoli16.txt -db cebra.fna -out blast_rRNA16.fna -outfmt 7
blastn -task blastn -evalue 0.05 -query ecoli23.txt -db cebra.fna -out blast_rRNA23.txt -outfmt 7*был выставлен порог e-value в 0.05, чтобы убрать случайные находки
В результате, поиск по 16S рРНК выдал 13, а по 23S рРНК - 76 находок blast:
Находки для 16S рРНК
Находки для 23s рРНК
*в файлах напротив каждой строчки написано, чем является находка - я вручную перепроверила каждую; если не написано
ничего - это не гомлог/является ncRNA.
16S rRNA
Среди 13 находок - одна случайная (большой e-value), одна - рибосомальная РНК (5 хромосома, NC_007116.7, [824924-824966]).
Все остальные - либо не принадлежали кодирующей части генома (lncRNA/ncRNA), либо входили в ген/mRNA.
23S rRNA
Половина (ровно) всех находок - случайные, у них очень большой e-value. Самая последняя находка попала в митохондриальный геном: это ген mt-rnr2 (NC_002333.2, [3027-3162]).
Митохондриально кодируемая 16S РНК представляет собой митохондриальную большую субъединицу рибосомной РНК, которая у человека кодируется геном MT-RNR2. Таким образом, значимая находка одна (по 16S и 23S рРНК) - это можно назвать гомологией.
Также, попался участок гена цинкового пальца (mRNA-gastrula zinc finger protein XlCGF57.1-like, NC_007115.7, [53400556-53400488]).