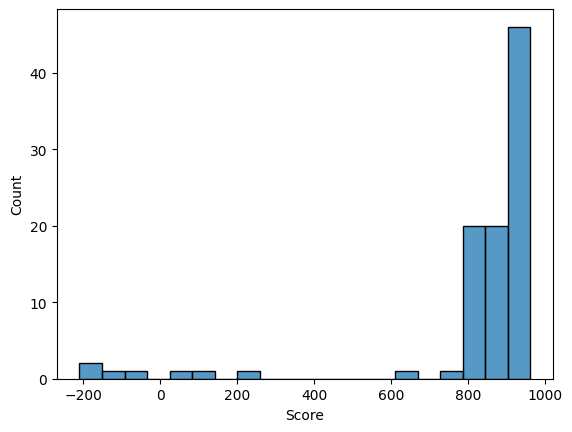

Я выбрала домен PF11471 (Sugarporin_N), который, по-видимому, является периплазматическим N-концевым продолжением мальтопоринов внешней мембраны (Pfam:PF02264, Lam).
У этого домена 5 архитектур, я выбрала одну из них (представлена белком Q7MB44, 97 последовательностей, длина – 468), содержащую домены Sugarporin_N и Porin_8.
Позитивная выборка: последовательности всех белков подсемейства.
Далее я провела "очистку" белков: выровняла их, оставила только участок от N-конца первого домена до C-конца второго, выровняла вновь, удалила последовательности с крупными
делециями внутри доменов, дубликаты и высокосходные последовательности (остановилась на 95%).
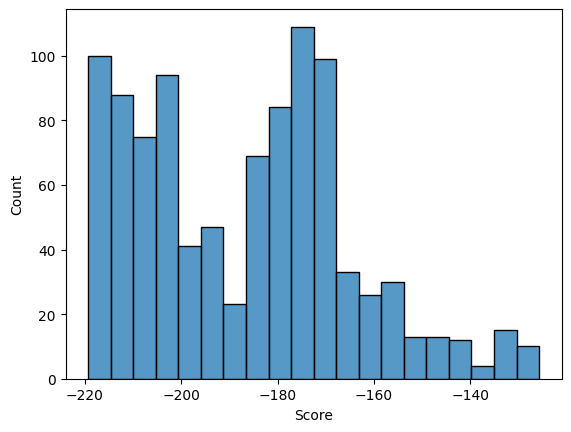
Обучающая выборка: выравнивание оставшихся последовательностей исходного подсемейства.
Далее, для формирования калибровочной выборки я добавила к своему подсемейству еще одну архитектуру, представленную белком
P22340 и содержащую 1058 белков (длина – 505). Она состоит из доменов Sugarporin_N и LamB.
Калибровочная выборка: выборка, состоящая из отрицательного контроля, положительного и обучающей выборки.
Далее был построен и откалиброван HMM–профиль:
hmm2build -g hmm_out.txt dom2_fin.fa
hmm2calibrate hmm_out.txt
hmm2search --cpu 1 hmm_out.txt full.fasta > hmm2search_out.txt
Тут можно найти профиль, а тут – находки в итоговой выборки.
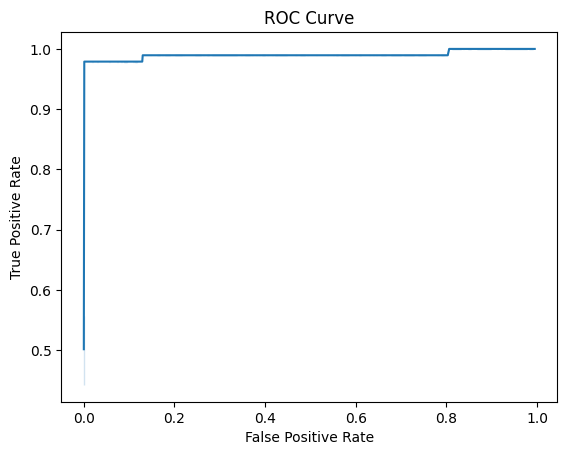
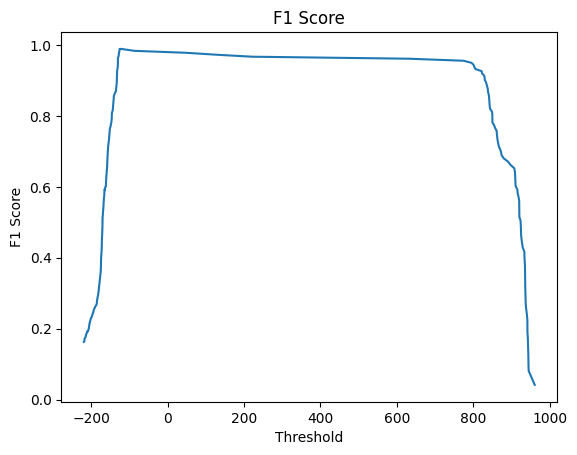
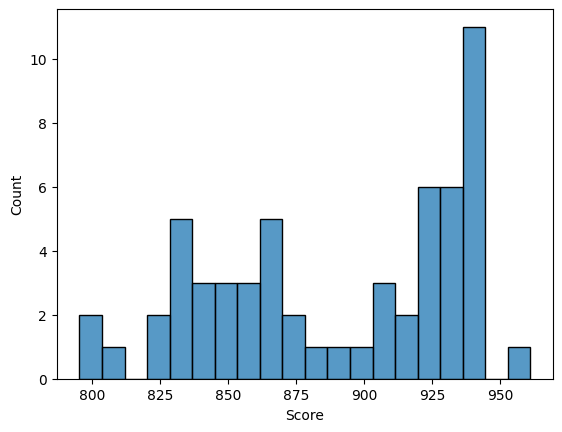
Для дальнейшего анализа я использовала скрипт, написанный Каримовой Кариной (202 группа). Ссылку на него можно найти тут. Он преобразовал нашу таблицу с находками, построил графики ROC Curve и F1 score. Результаты приведены ниже: