
Мнемоника функции белков: PURA
Я составила список идентификаторов белков по схеме: PURA_ACICJ, PURA_AROAE, PURA_HAEIN, PURA_PARDP, PURA_POLAQ, PURA_ROSDO, PURA_YERPE
Поиск проводила тут: форма загрузки из Uniprot.
Далее сохранила файлы в единый fasta: лежит тут.
Отредактировала файл, оставив после ‘>’ только мнемонику вида бактерии
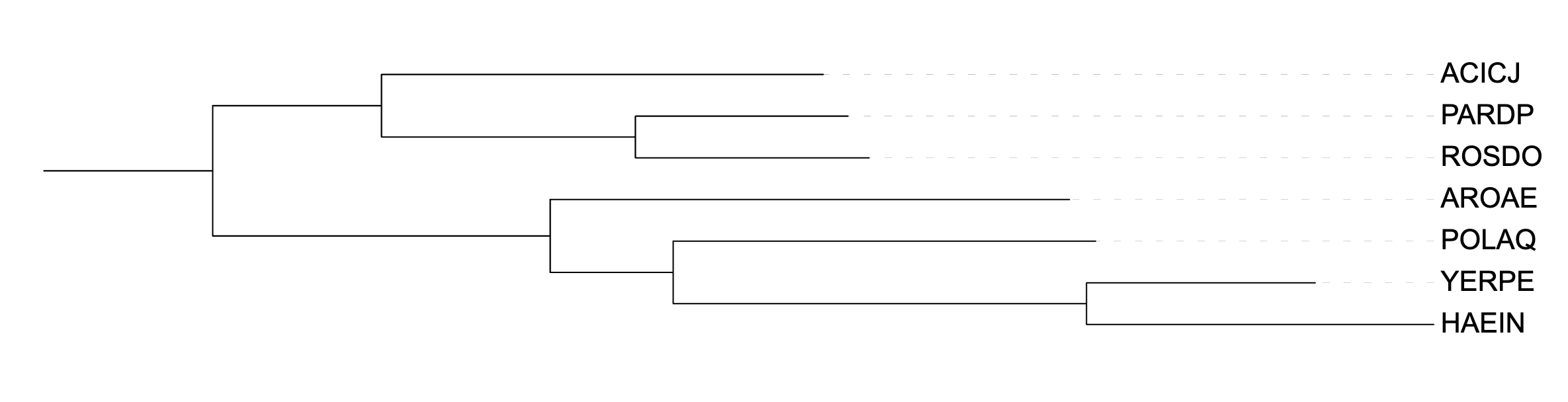
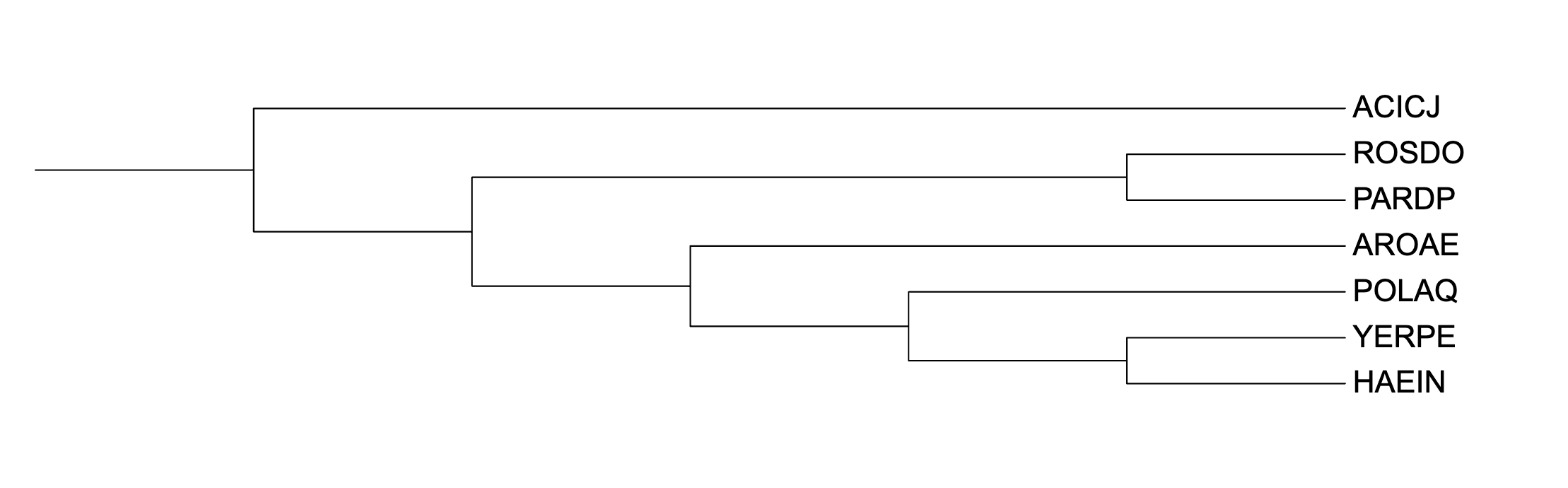
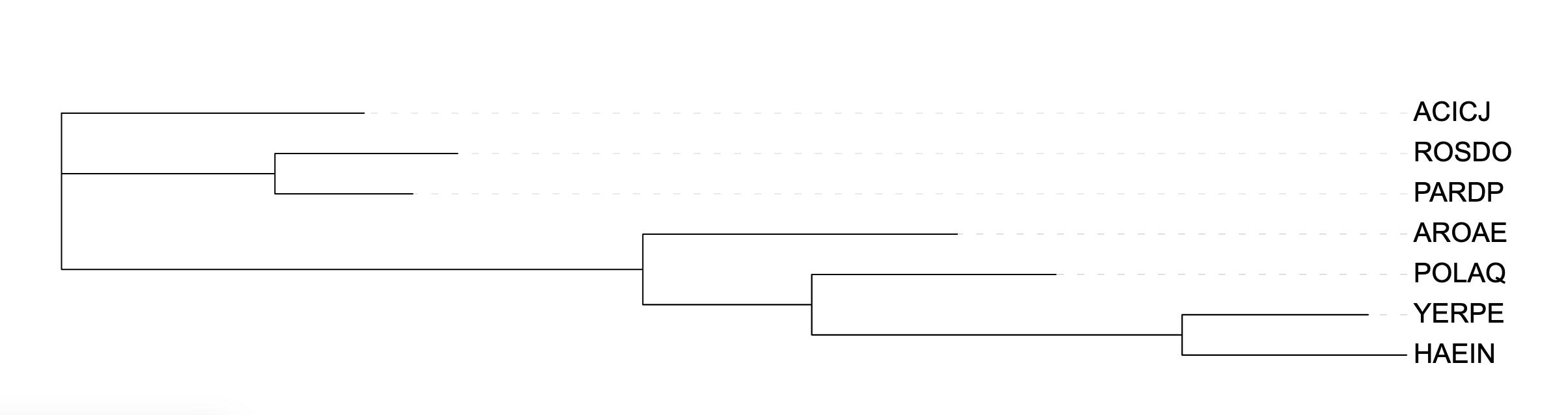
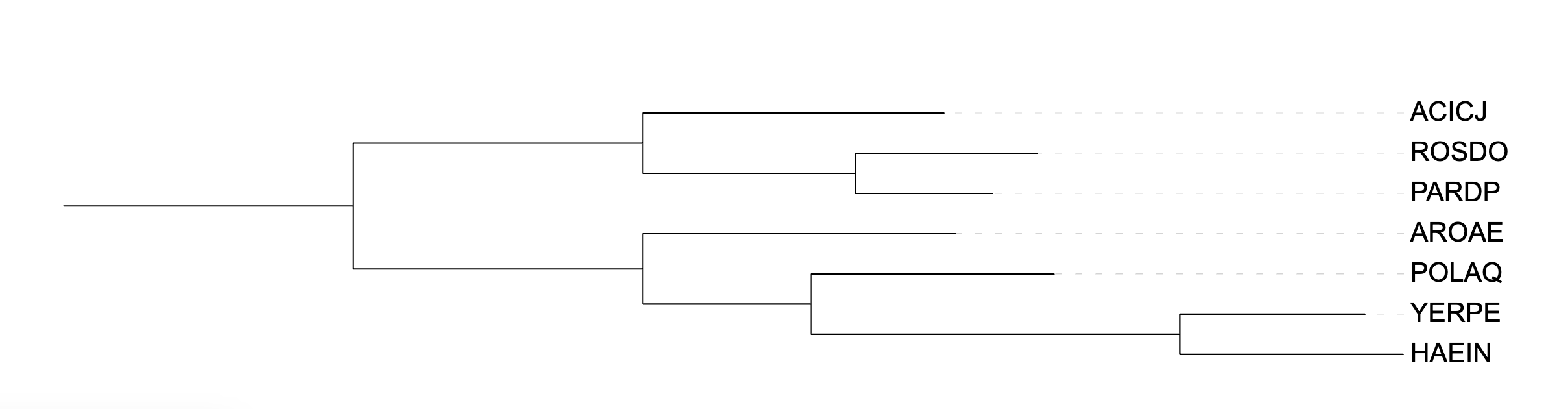
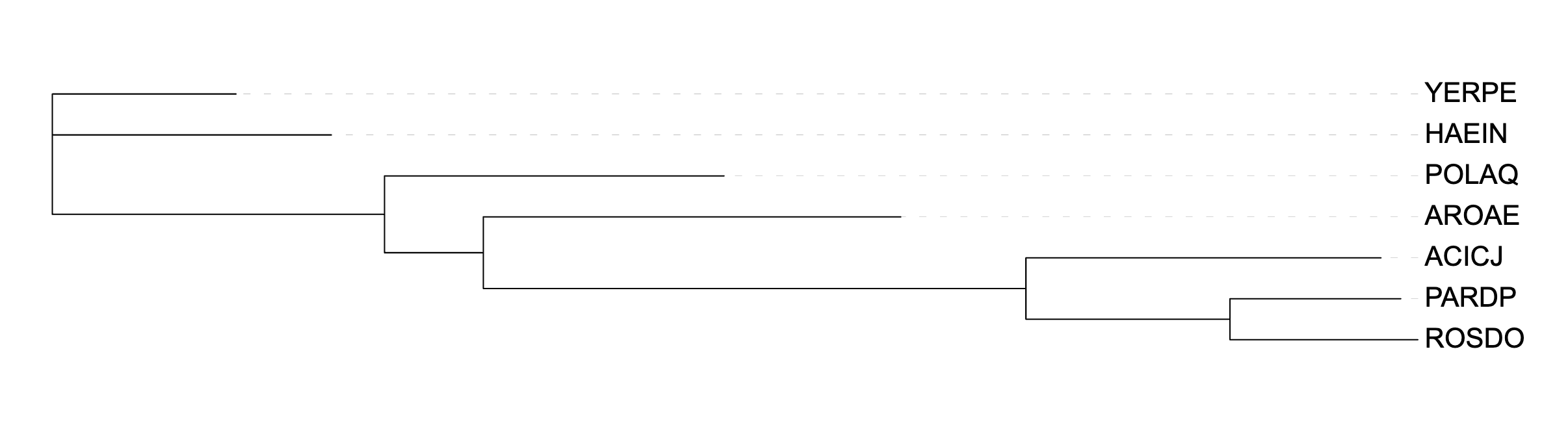
Далее я провела реконструкцию филогении тремя способами: MAFFT → FastME, MAFFT → TNT,
MAFFT → PhyML.
FastME — минимальная эволюция, TNT — максимальная экономия, PhyML — максимальное правдоподобие.
Ниже приведены деревья.