
Для выполнения данного задания я скопировала в свою директорию протеомы выбранных мною бактерий.
Затем объединила их в один файл - all.fasta командой:
cat ACICJ.fasta AROAE.fasta HAEIN.fasta PARDP.fasta POLAQ.fasta ROSDO.fasta YERPE.fasta > prots.fasta
Поиск гомологов белка CLPX_ECOLI осуществлялся при помощи следующих команд:
makeblastdb -dbtype prot -in prots.fasta -out bac_db
blastp -query CLPX_ECOLI.fasta -num_threads 4 -db bac_db -evalue 0.001 -out blast_res.txt
Найдено 27 гомологов (для удобства объединила их последовательности в один файл).
Находки можно найти здесь.
Последовательности находок лежат тут.
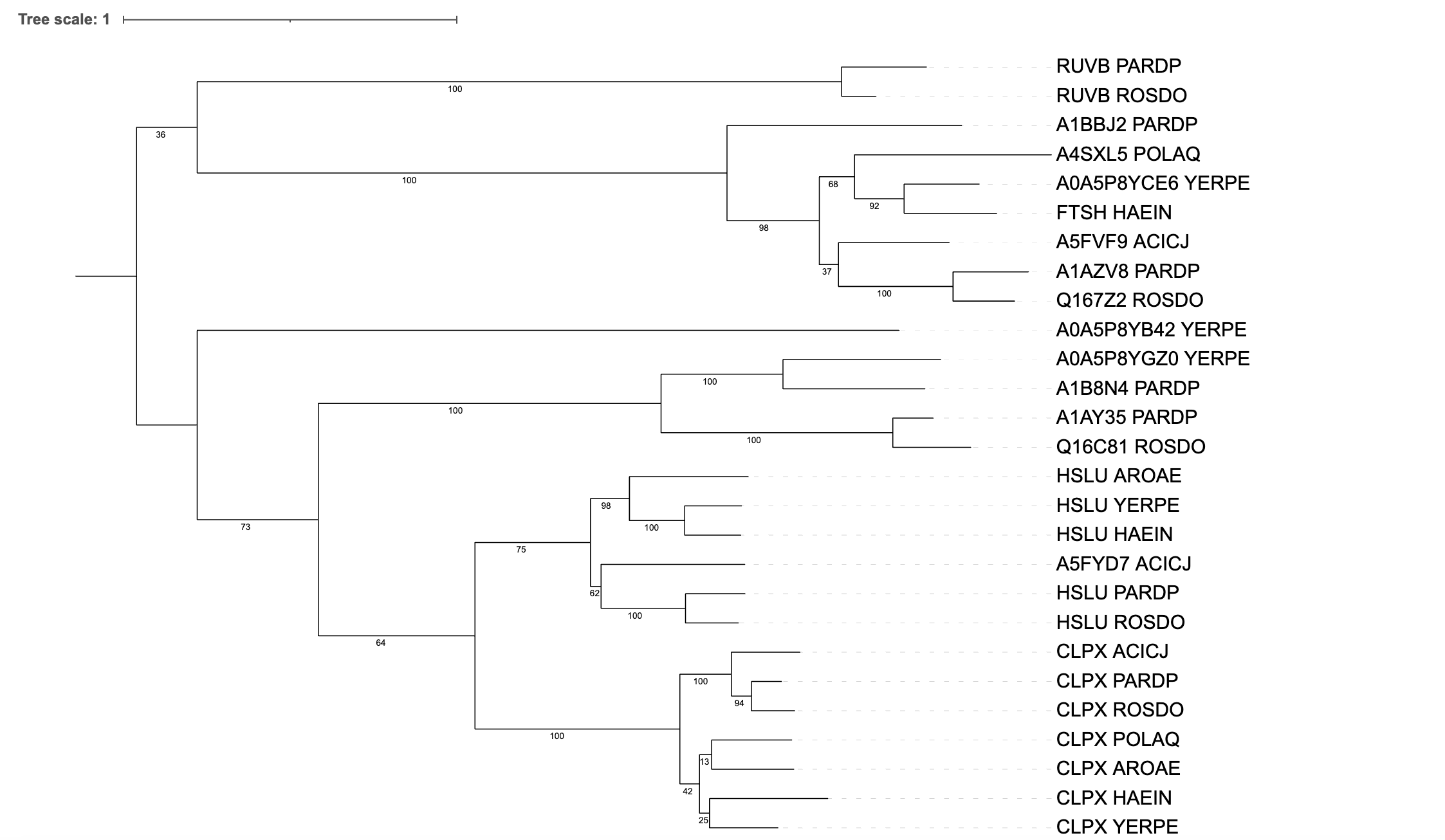
Дерево было реконструировано программой FastME с параметрами: 'Gamma distributed rates across sites' — No,
'Starting tree' — BIONJ, 'No refinement', 100 бутстреп реплик. Newick формула тут.
По реконструкции мы нашли ортологов и паралогов.
Ортологи:
CLPX_PARPD и CLPX_ROSDO, CLPX_YERPE и CLPX_HAEIN, HSLU_ROSDO и HSLU_PARDP
Паралоги:
CLPX_ACICJ и A5FYD7_ACICJ, A1B8N4_PARDP и A1AY35_PARDP, A1BBJ2_PARDP и A1AZV8_PARDP

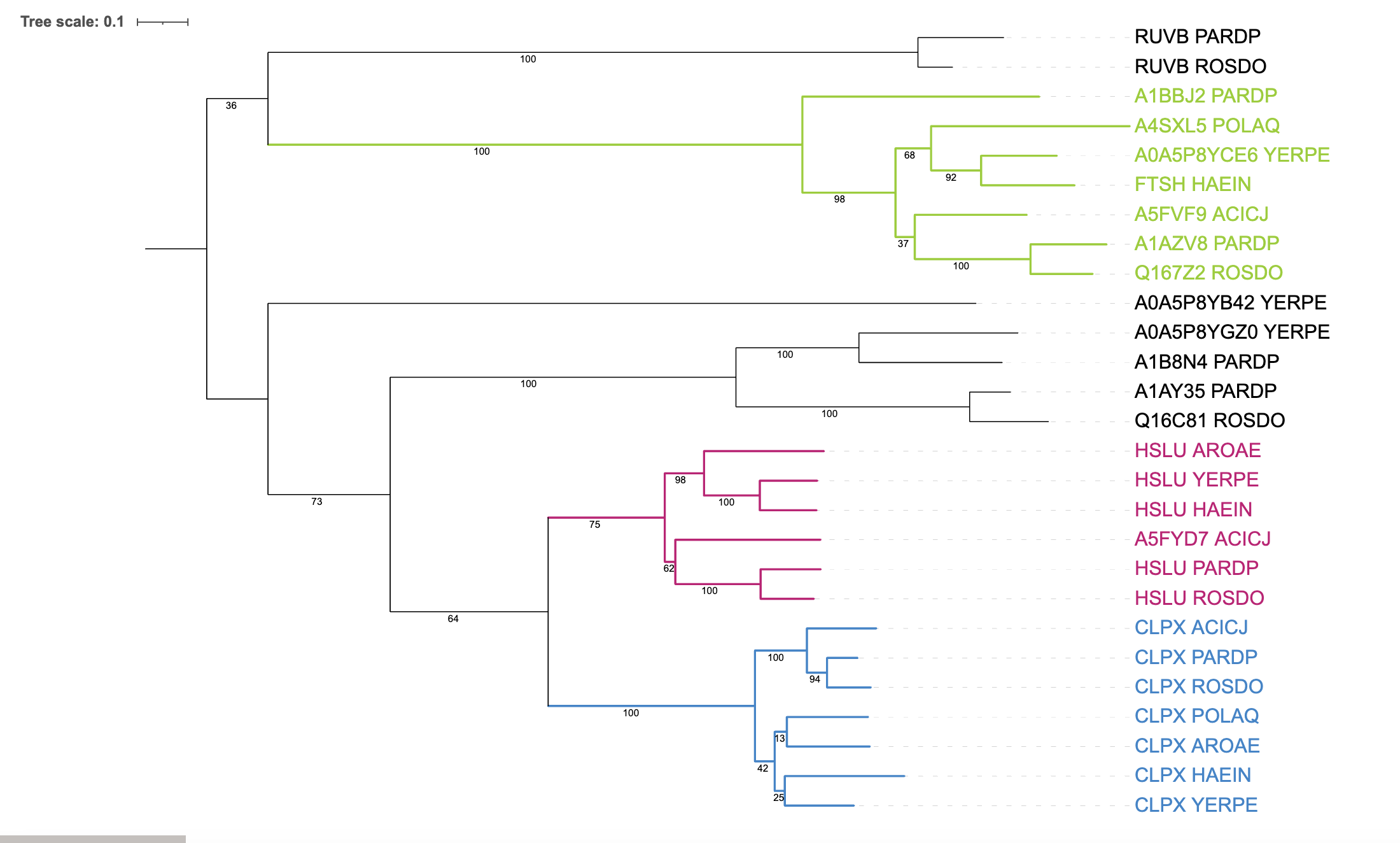
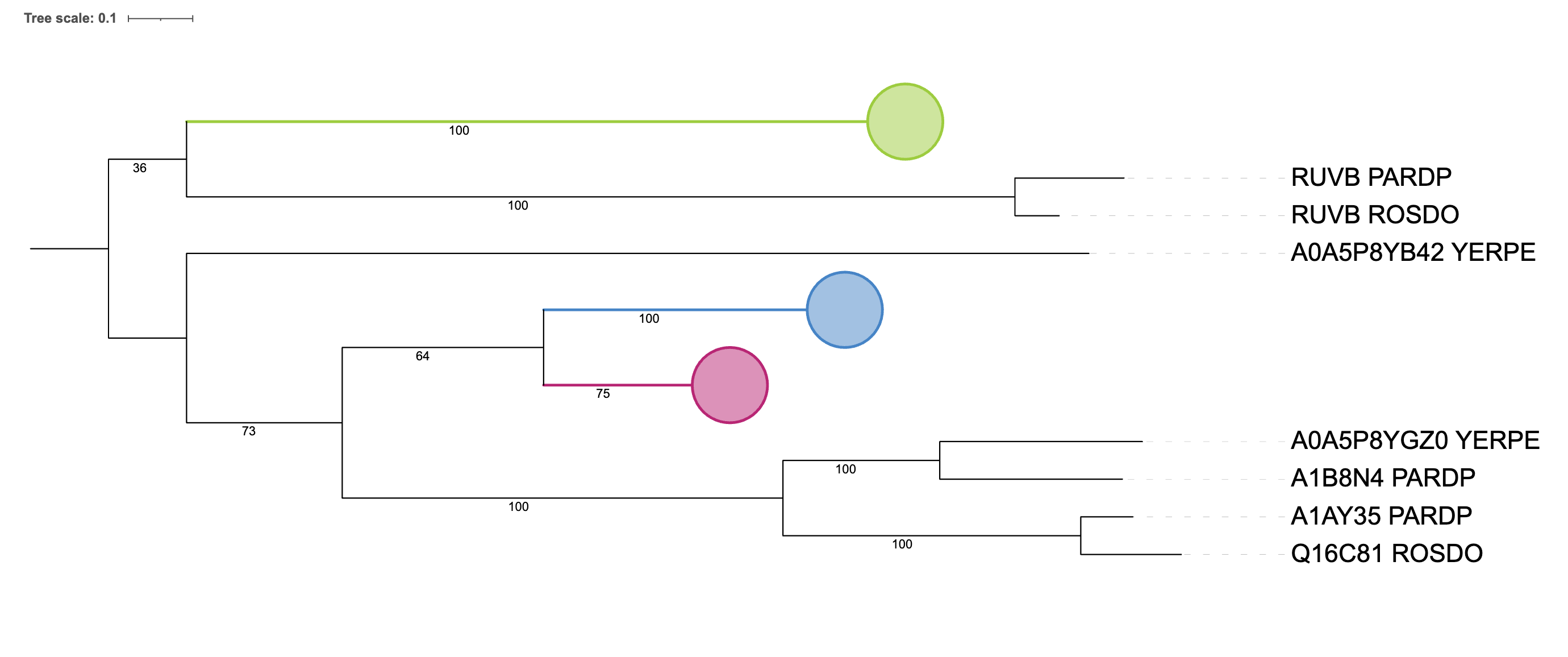
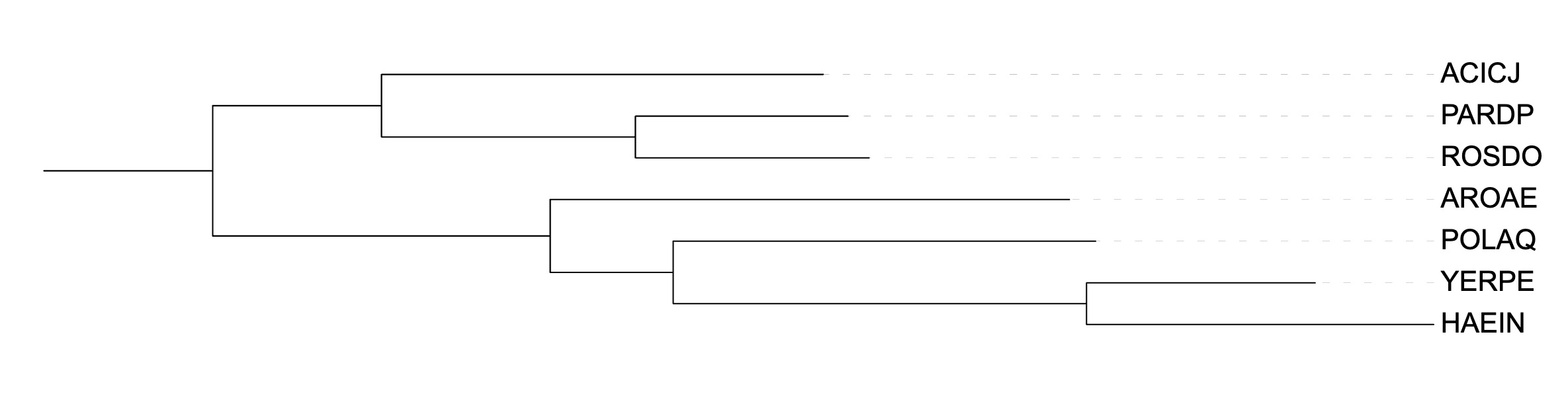
Сравним ортологические группы с эталонным деревом (Рис. 1). Во всех группах часть нетривиальных ветвей правильная, общая закономерность "ACICJ, PARDP, ROSDO против AROAE, POLAQ, YERPE, HAEIN" сохраняется.