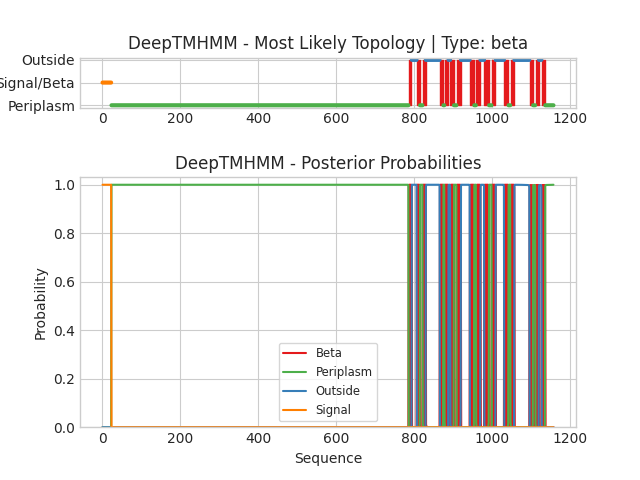

Название: Cellulose synthase operon protein C (channel BcsC) (Белок C оперона целлюлозосинтазы)
Организм: Escherichia coli
Функция: Необходим для максимального синтеза бактериальной целлюлозы
Выдача ОРМ: тут
Идентификатор Uniprot: BCSC_ECOLI
PDB: тут
Толщина трансмембранной части белка: 24.2 Å
Координаты трансмембранных участков (по OPM): 1(785- 793), 2(807- 815), 3(821- 829), 4(866- 873), 5(880- 884), 6(894- 901), 7(907- 917), 8(944- 952), 9(960- 968),10(980- 989),11(999-1008),12(1030-1040),13(1046-1057),14(1094-1104),15(1112-1119),16(1125-1134)
На вход DeepTHHMM подала fasta-файл с последовательностью амbнокислот (взяла в UniProt). Выдача программы:

Мне был выдан белок SAP_MYCTU (ID: O07802) (экспортер сульфолипида-1).Он необходим для транспорта через внутреннюю мембрану сульфолипида-1
(SL-1), который является основным липидом клеточной стенки патогенных микобактерий.
Ниже приведена выдача программы DeepTMHMM. Последовательность была взята из UniProt. Координаты можно найти тут.
Было предсказано 6 трансмембранных альфа-спиралей (в PPM - 7).
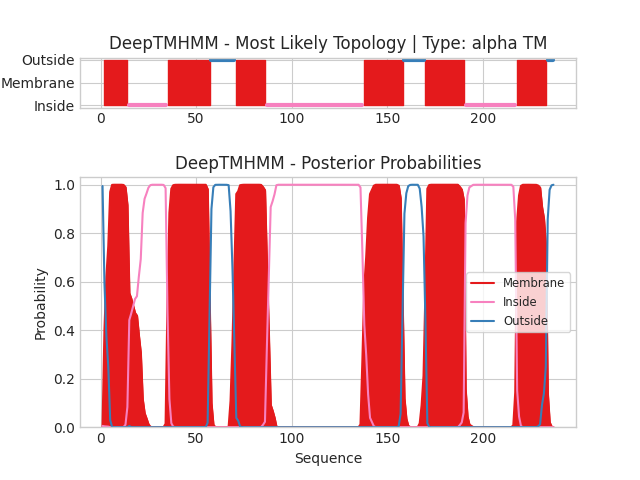

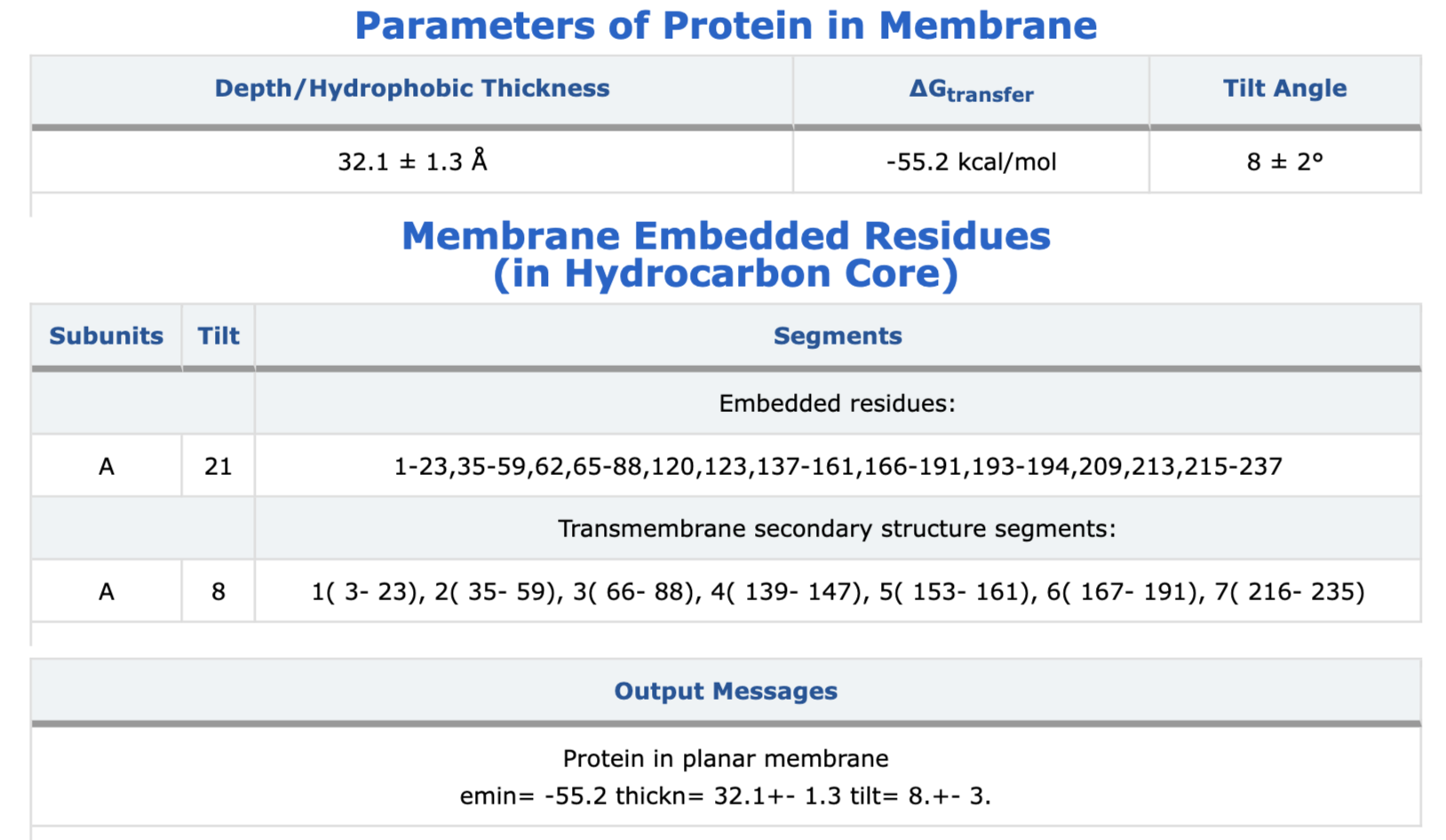
Можно заметить, что выдачи двух программ отличаются. Одна из них, если посмотреть на координаты, объединила два участка в один (может, участок был сложным?). Это объясняет полученные результаты.
Также важно отметить предсказание AlphaFold. Я изучила модель, которую он предложил (и которая размещена в UniProt), и, к сожалению, очень высокий pLDDT (confidence level) есть у одной единственной аминокислоты (что составляет 0,4% от общего количества). Качество предсказания, иными словами, не очень хорошее, и это совершенно точно могло оказать влияние на результаты предсказания сервера PPM.