Мне досталась довольно большая выборка белков (70 штук), поэтому сначала я хотела работать исключительно с базами данных, похожих на Panther (они строят bar-graph’ы, что довольно удобно в условиях большой выборки), однако в итоге я остановилась на двух других базах данных.
На вход я подала список ID белков, поиск проводился по таксону Homo sapiens (по-другому програмам отказывалась работать). Выдача – очень страшный (на первый взгляд) граф:

Далее я рассмотрела Reactome.
Тут также не нашёлся один белок: PGAM4. Думаю, его действительно нет в базах данных/неверно записан ID.
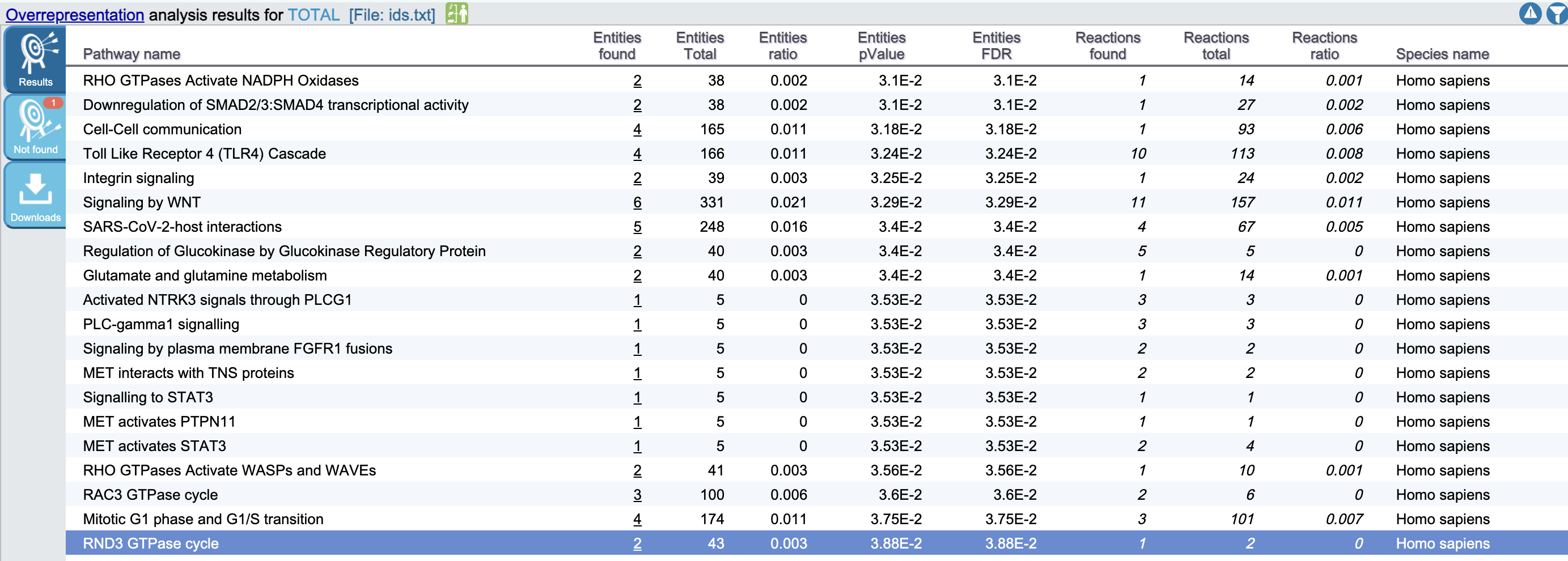
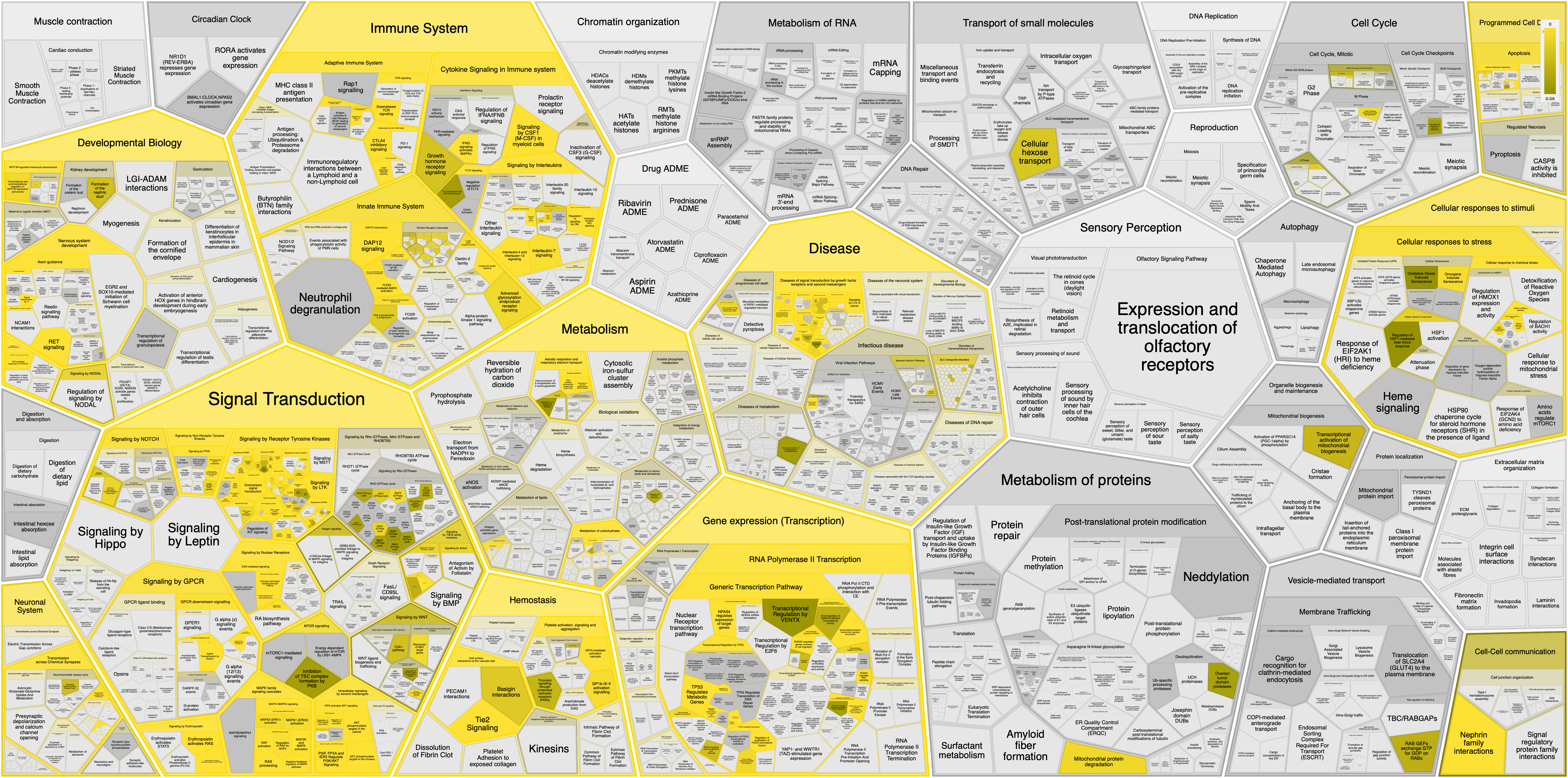
Далее приведены отдельно "области" (к сожалению, оформить их по-другому не удалось), в которых встречаются исследуемые белки.
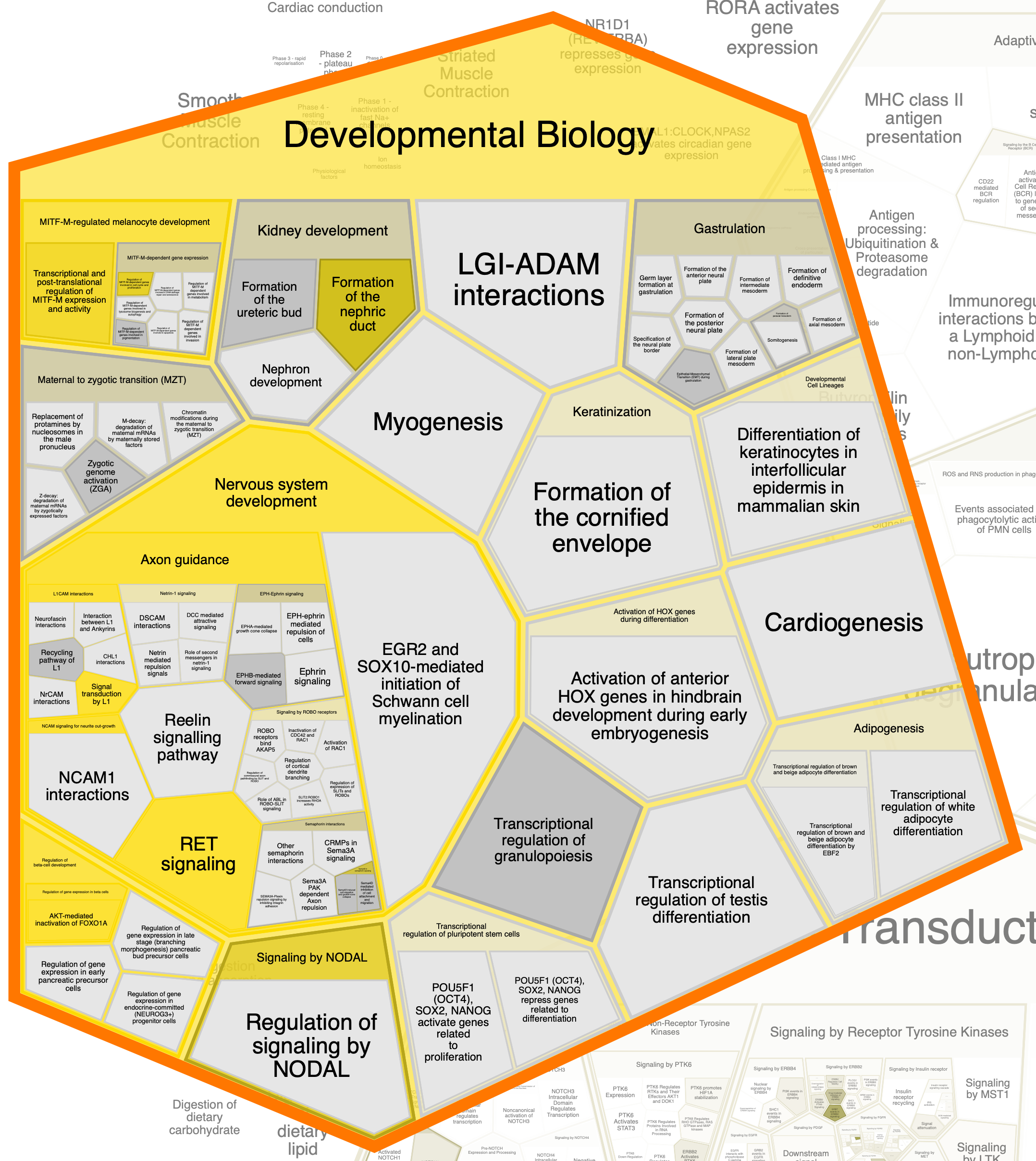
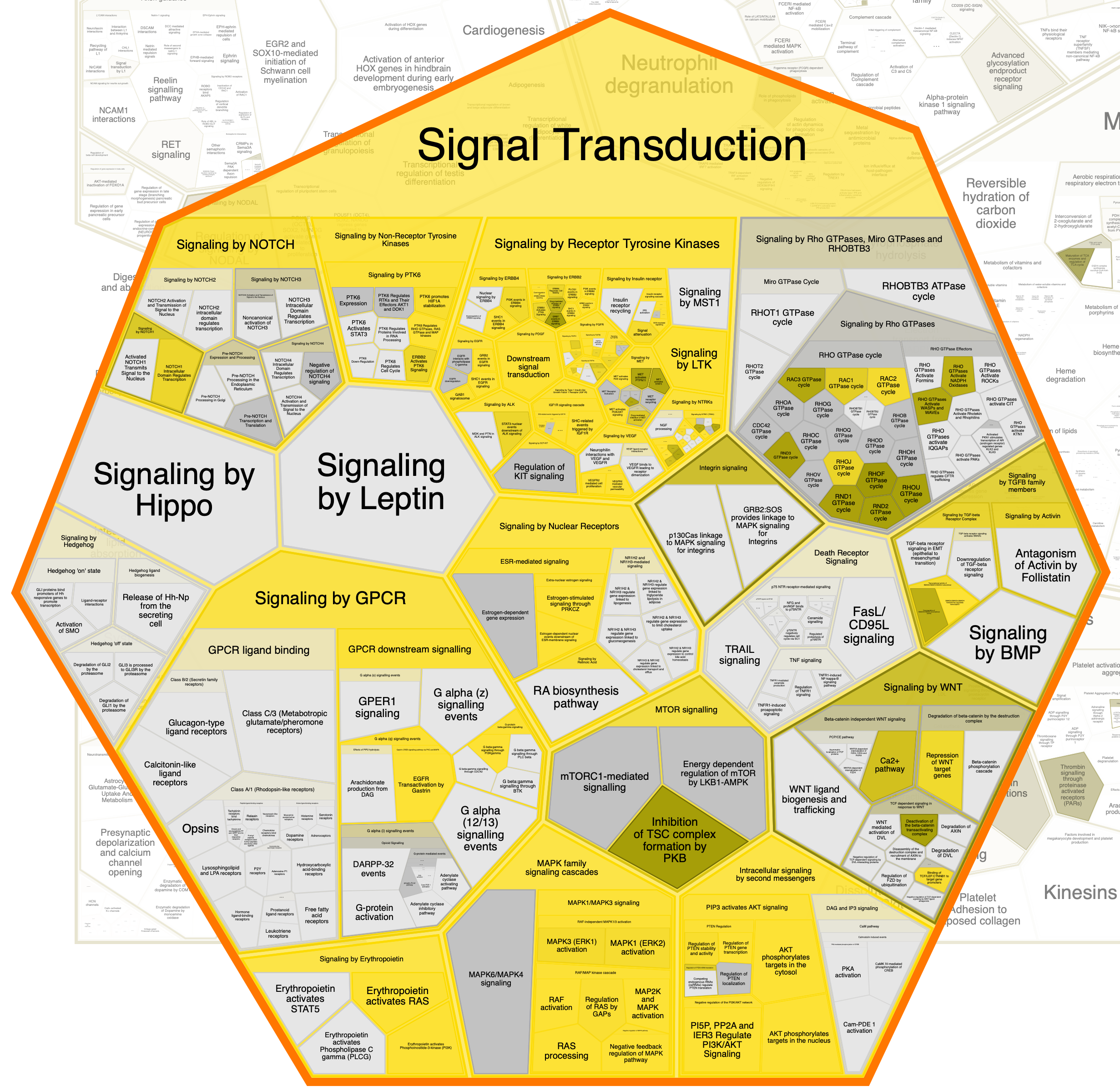
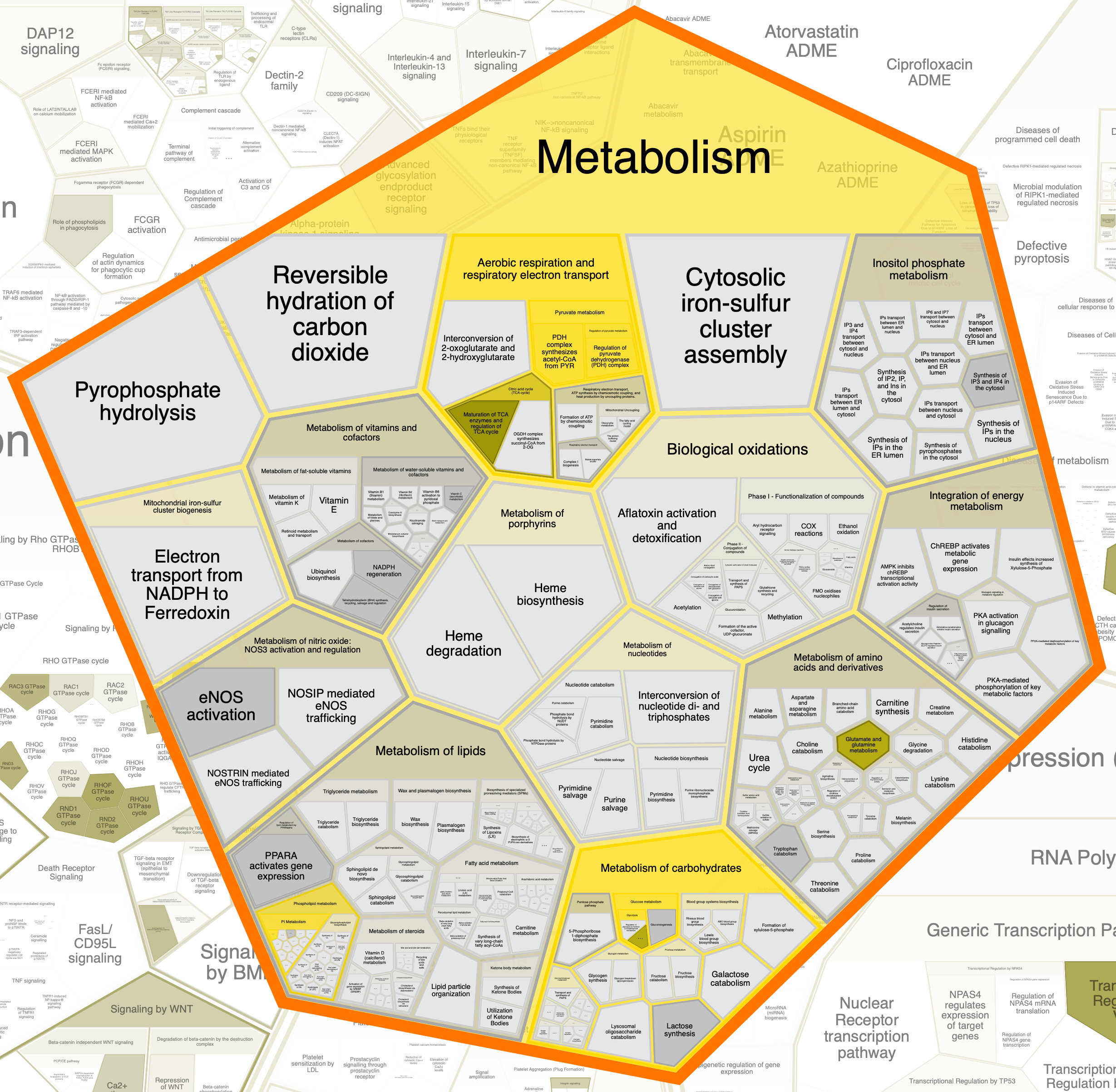
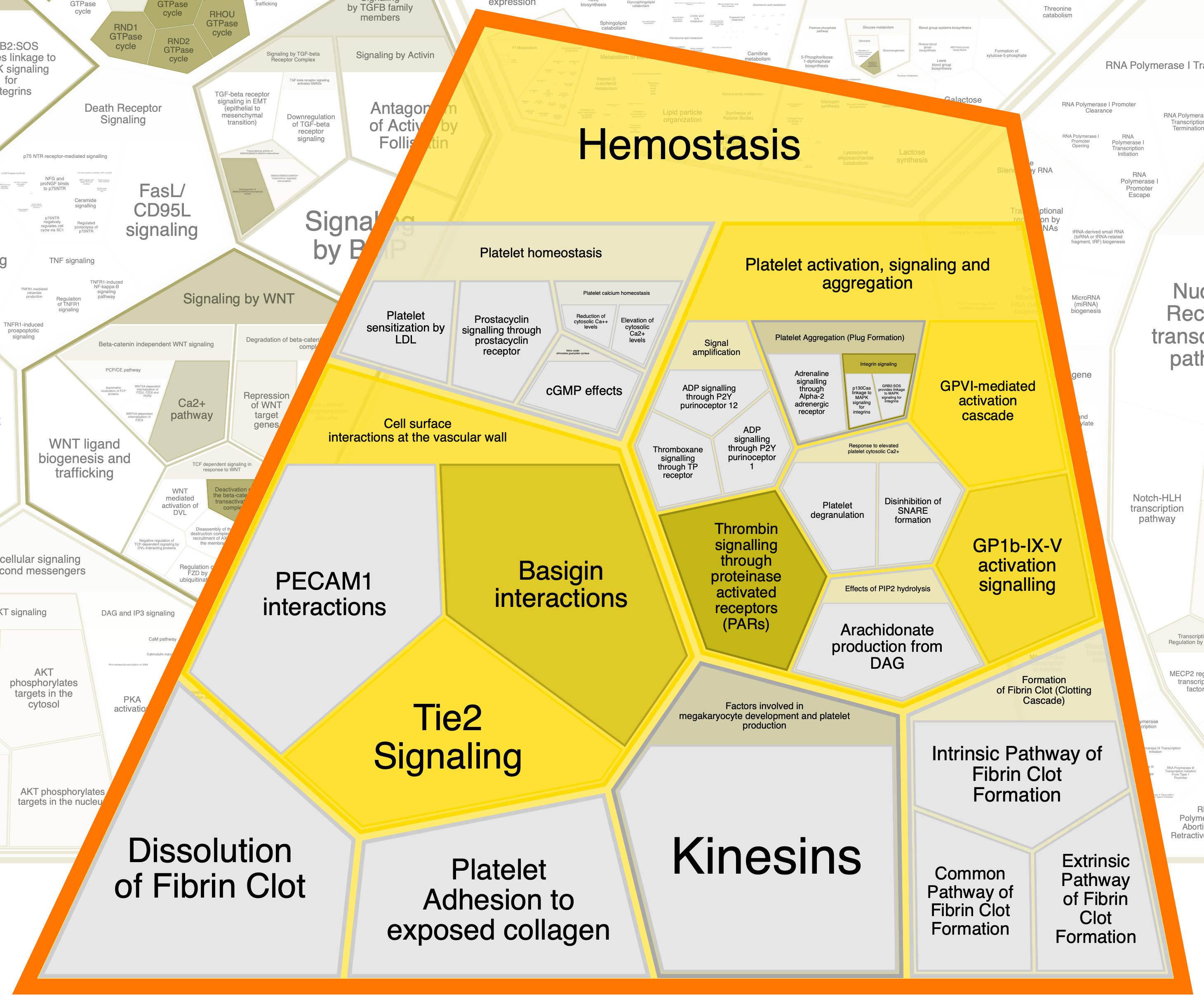
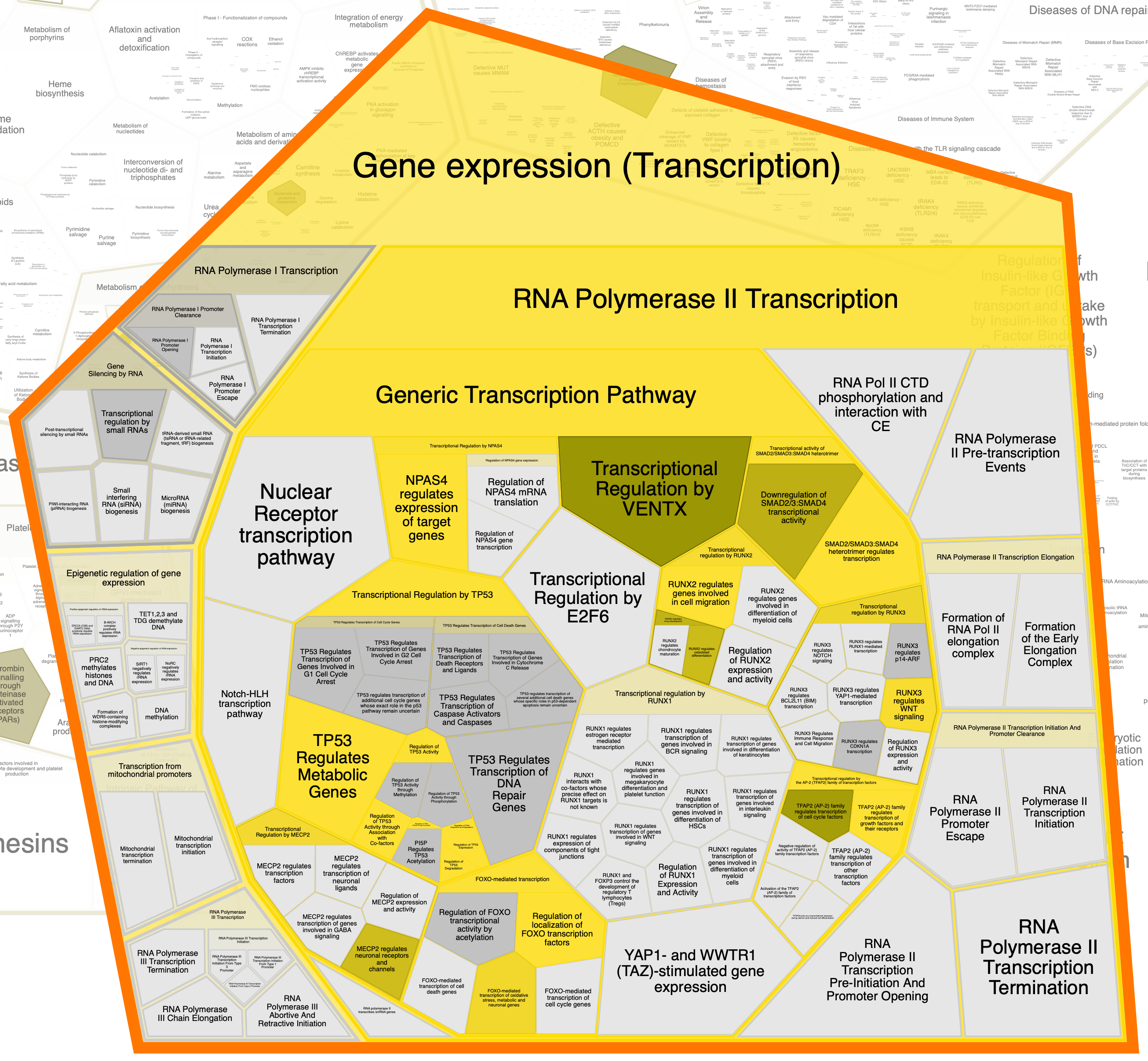
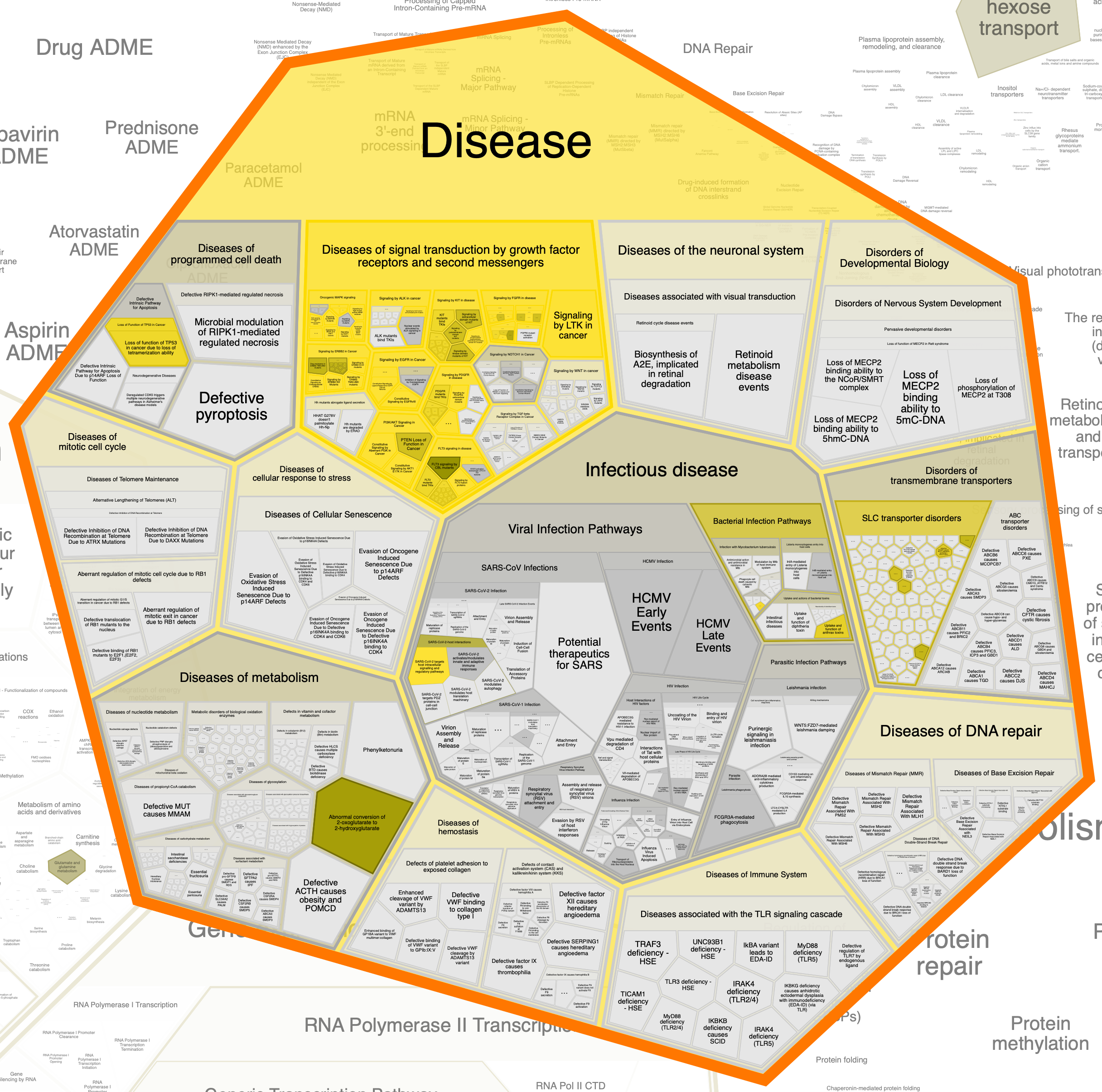
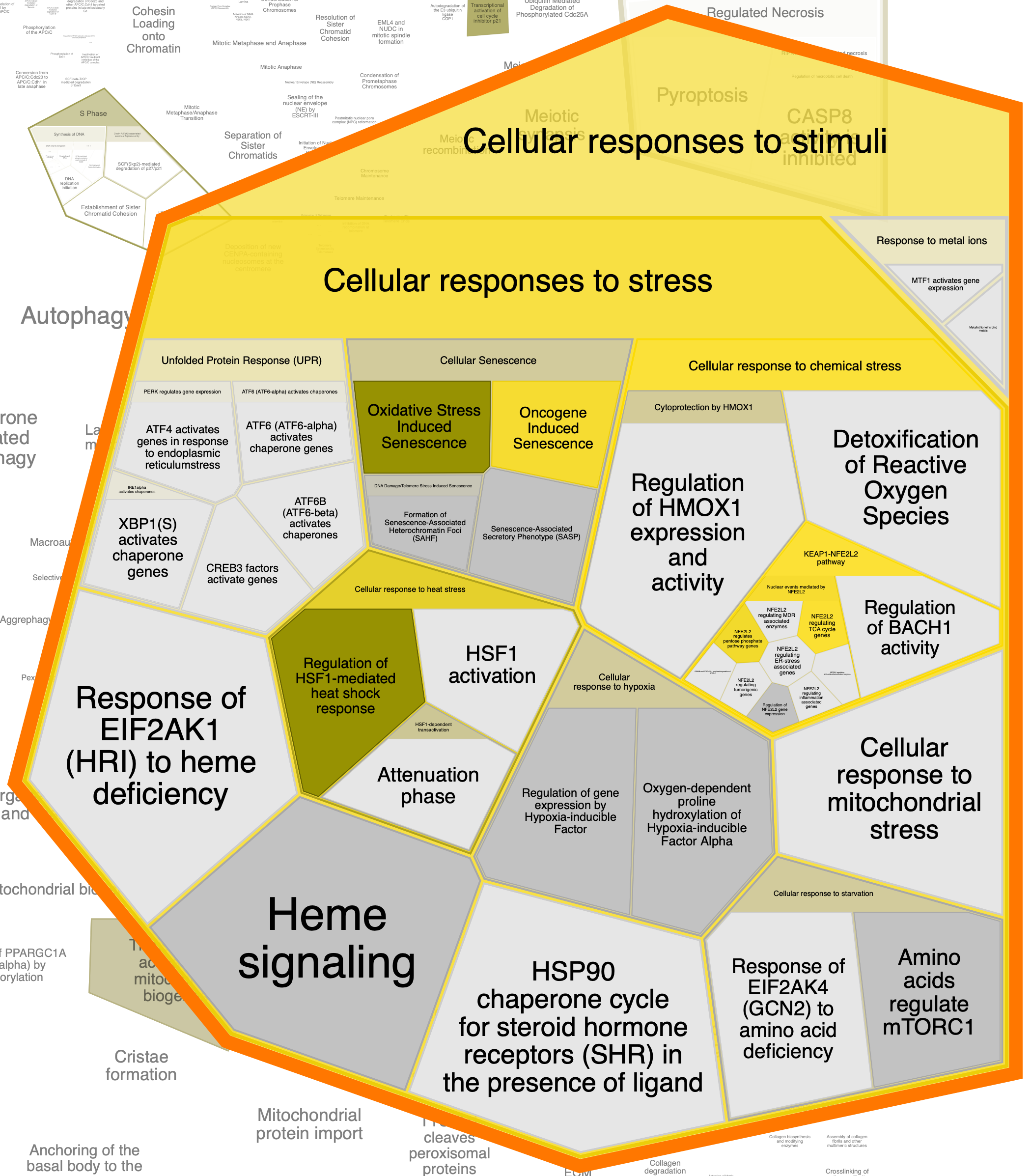
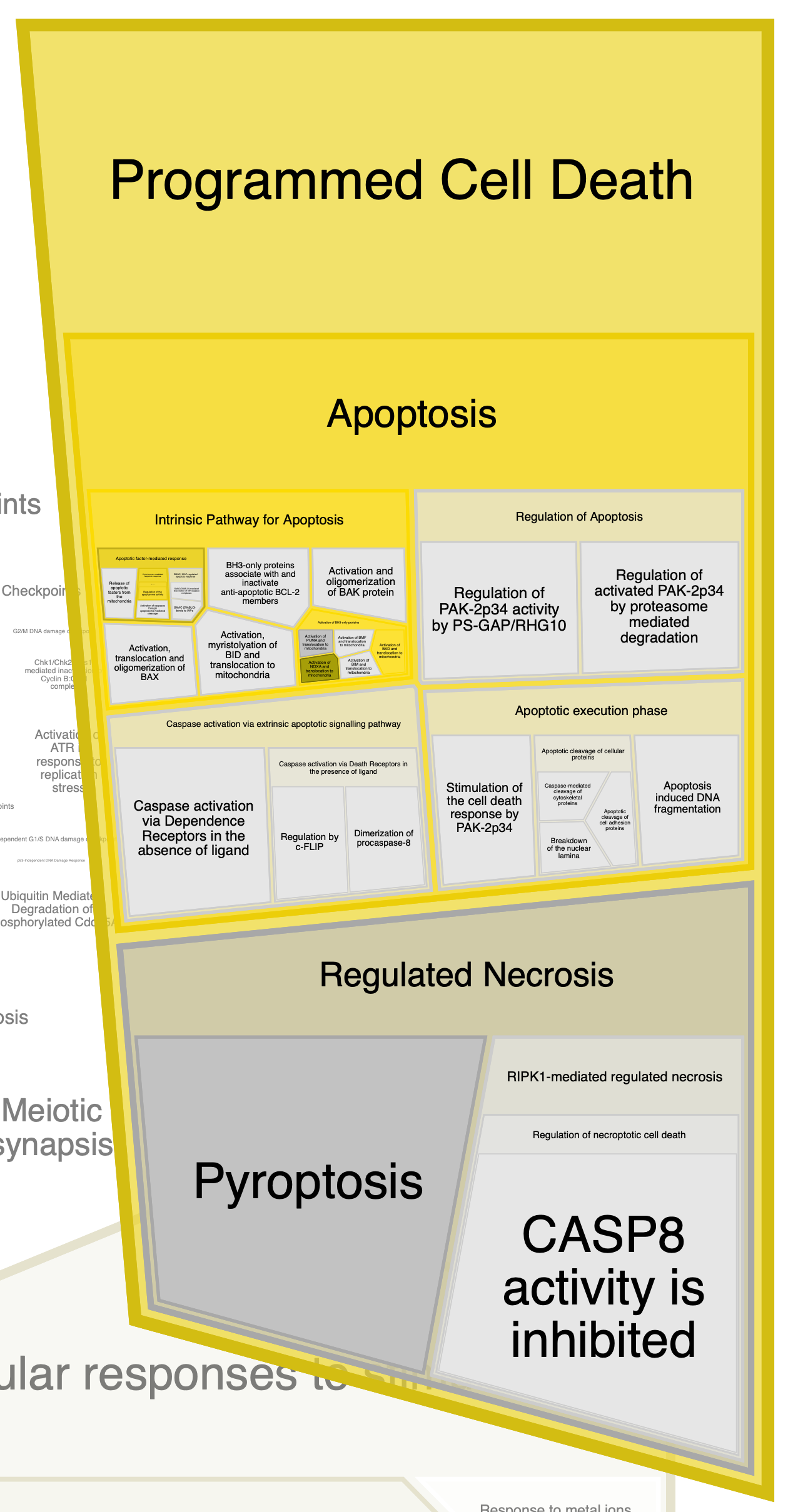
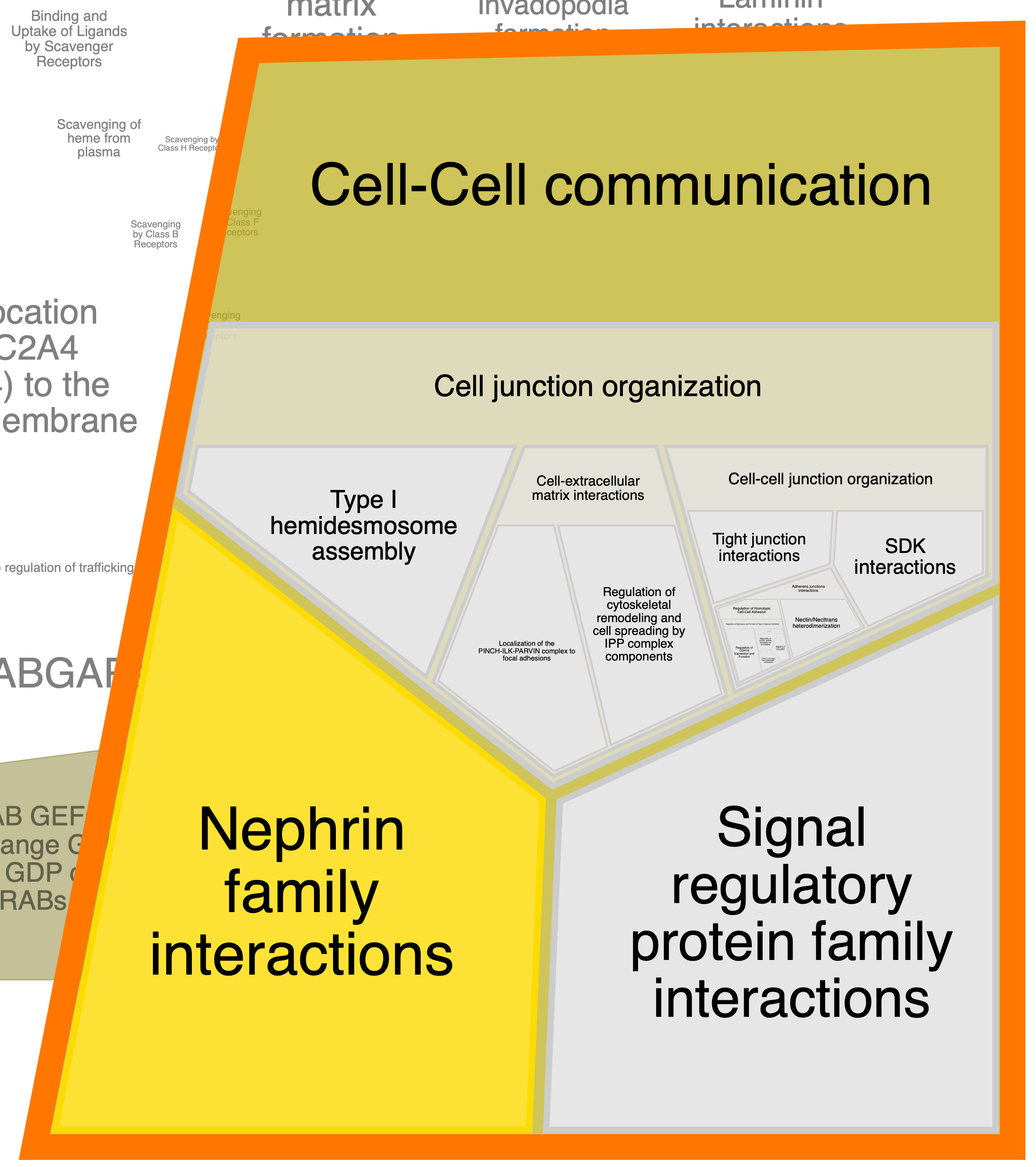
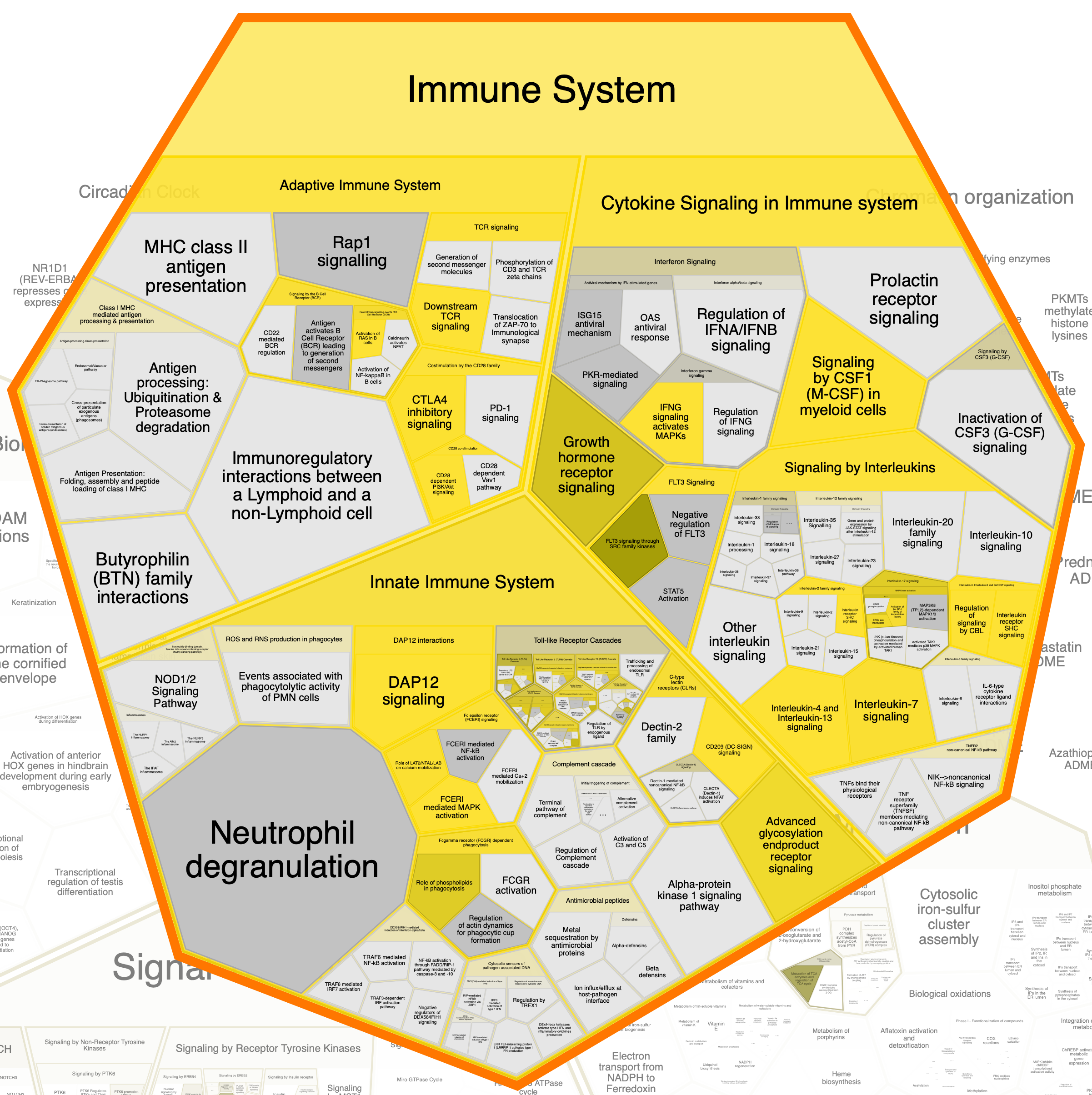
Глобально, облако слов, составленное мной, совпадает с областями, выделяемыми этой программой: также мы видим слова
«receptors», «signal transduction», «metabolism», «immune system», «developmental biology».
Кластеризация String теперь кажется более понятной.
Белки были сгруппированы на основе их функциональной активности и участия в определённых биологических процессах:
Первая группа содержит белки, которые больше всего участвуют в передаче сигналов в клетках через различные каскады фосфорилирования и активации киназ. Эти белки играют ключевую роль в таких процессах, как рост клеток, выживание, дифференцировка, развитие и ангиогенез. В эту группу входят различные рецепторы ростовых факторов, рецепторные и не-рецепторные тирозиновые киназы, а также некоторые важные сигнальные молекулы, как Ras и PI3K.
Вторая группа объединяет ферменты, прямо участвующие в метаболических путях, таких как гликолиз, пентозофосфатный путь и цикл трикарбоновых кислот (цитратный цикл). Эти ферменты включают киназы, дегидрогеназы и фосфатазы, которые катализируют ключевые реакции в энергетическом метаболизме и биосинтезе.
Третья группа в основном связана с регуляцией метаболизма на системном уровне и ответами на изменения в окружающей среде, включая гипоксию и питание. Эта группа включает такие белки, как серин/треониновые киназы AKT, которые регулируют усвоение глюкозы и клеточное выживание, а также ключевые регуляторы метаболизма и стресса, такие как mTOR и HIF-1α.