

Для работы был выбран референсный геном Campylobacter coli (GCF_009730395.1_ASM973039v1) – бактерии, с которой я работала в первом семестре.
Геном скачала NCBI. Далее, при помощи сервиса Operon-mapper я получила список оперонов
(можно найти тут).
Далее этот список вместе с геномом бактерии я подала на вход скрипту,
который был написан Георгием Муравьёвым. Я получила три выборки: обучающую (гены домашнего хозяйства),
тестовую (промоторы) и отрицательный контроль.
Далее был использован локальный MEME:
meme housekeeping.fasta -dna -nmotifs 3 -minw 6
Выдачу можно найти тут


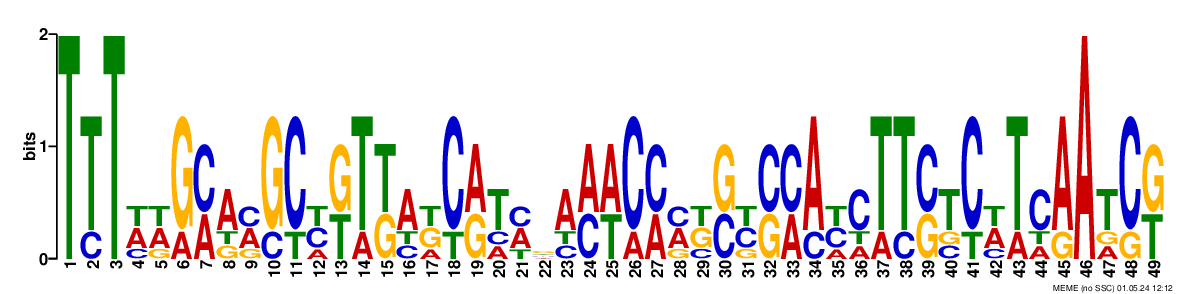
Для поиска второго найденного мотива в положительном и отрицательном контролях я использовала следующие команды:
fimo --norc -motif TTCCTYTT -thresh 0.001 ./meme_out/meme.txt ./promotors.fasta
fimo --norc -motif TTCCTYTT -thresh 0.001 ./meme_out/meme.txt ./negative.fasta
В результате работы программы я получила две таблицы c находками: для promotors и negative control соответственно. Мотив нашёлся в 457 промоторах положительного контроля и 21 негативного.