В начале были даны 2 файла с хроматограммами 22_F.ab1 и 22_R.ab1 прямой и обратной цепочками ДНК. Анализ хроматограмм был проведен в программе UGENE. При первом просмотре хроматограмм были примерно определены концевые нечитаемые участки последовательностей. Для прямой последовательности с 1 по 23 и с 691 по 721, для обратной - с 1 по 23 и с 694 по 700. Уровень шума, как и ожидалось, был наиболее высок вначале и в конце хроматограмм. В середине же последовательностей уровень шума в среднем был около 5%. Были получены прямая и обратная консенсусные последовательности и их выравнивание, а так же выравнивание исходных последовательностей. В результате работы также были обнаружены проблемные нуклеотиды (примеры 1-3):
Пример 1
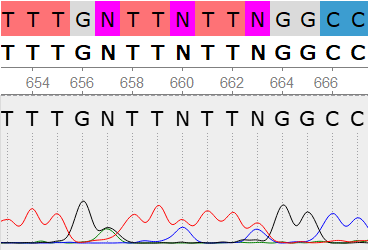
На 657, 660 и 663 позициях программа поставила N, хотя, согласно хроматограмме, там должны быть полиморфизмы R, Y и Y соответсвенно. Однако из-за невозможности сравнения с обратной последовательностью и близости конца хроматограммы данное явление можно также списать на увеличение уровня шума.
Пример 2
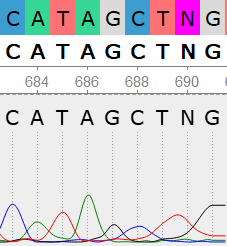
На 690 позиции - пересечении хромотограмм G и T, поэтому ранее поставленный лишний нуклеотид N был удален из консенсуса.
Пример 3
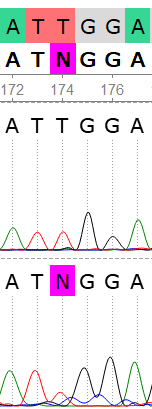
На 174 позиции в обратной последовательности произошло повышение уровня шума из-за чего программа не смогла определить нуклеотид. В прямой последовательности этого не наблюдалось, поэтому поставленный программой нуклеотид N был заменен на T.
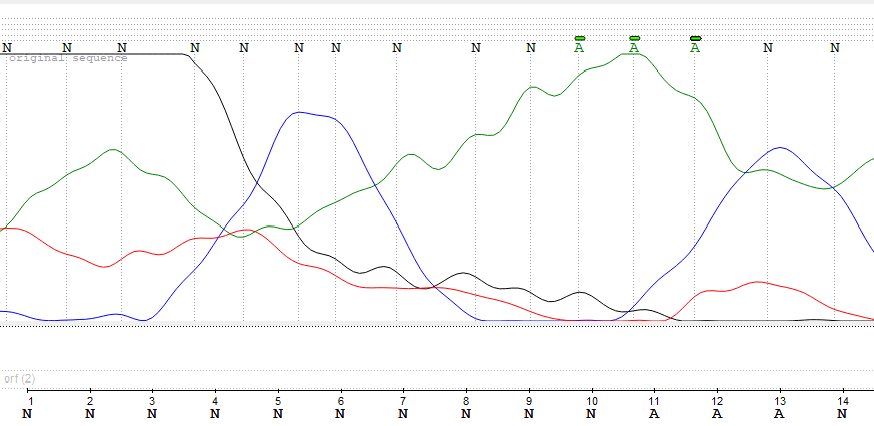
Снизу представлен нечитаемый фрагмент из начала (3'-конца) хроматограммы первоначальной обратной последовательности последовательности. В нем видно сильное перекрывание аденином остальных нуклеотидов, и также отсутствуют четко выделяющиеся пики, что мешает определить последовательность ДНК.