Были определены предполагаемые функция и таксономия для полученной в практикуме 6 консенсусной последовательности. Для поиска был использован blastn с алгоритмом megablast, так как данный алгоритм оптимизирован для поиска очень близких гомологов. В качестве базы данных поставлен параметр Nucleotide collection. Часть находок представлена на Рисунке 1.
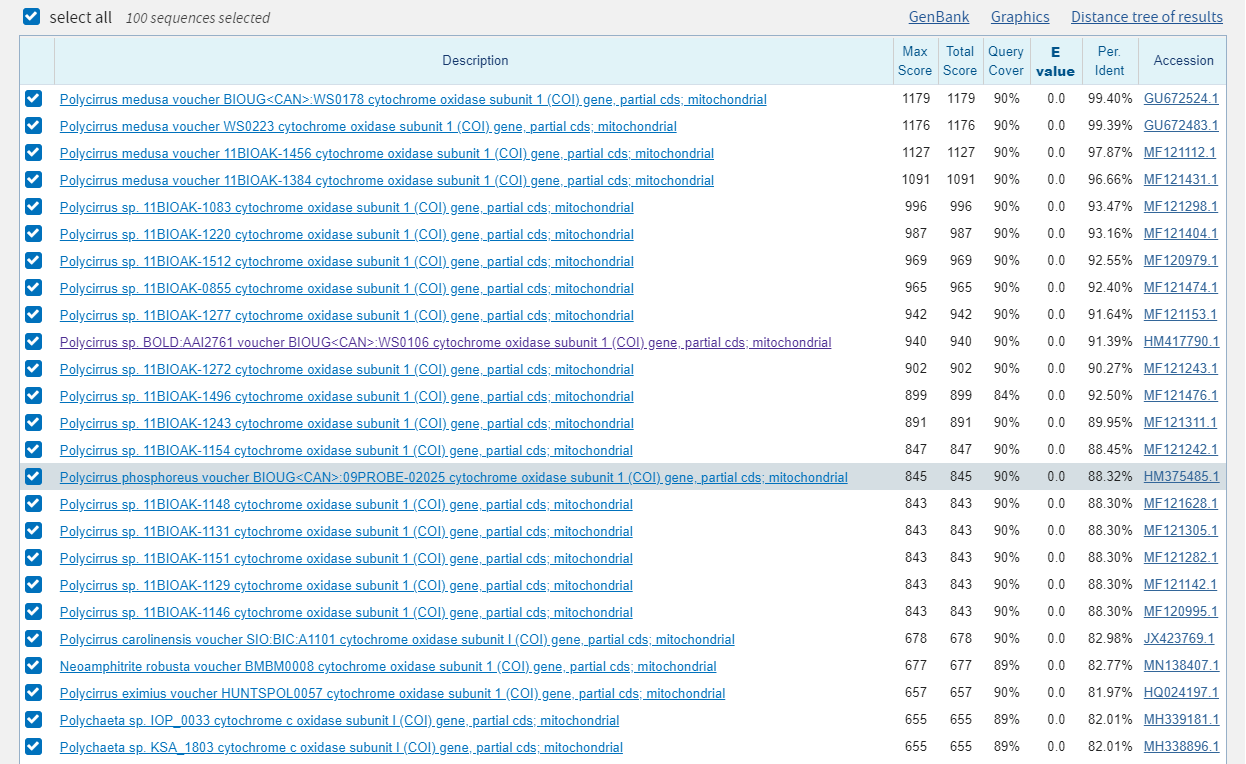
Рисунок 1. Находки megablast с наибольшим весом
Выдача BLAST показывает, что консенсусная последовательность скорее всего является участком гена, кодирующего первый субъюнит цитохром-оксидазы (COI). Для определения таксономии последовательности было выбрано несколько находок, принадлежащих к разным родам.Общая таксономия приведена на рисунке 2. Также было построено множественное выравнивание выбранных находок (fasta-файл). По выравниванию видно, что последовательности возможно являются гомологами из-за протяженных консервативных участков. Это подтверждает данные о таксономии видов. Следовательно, последовательность характерна для представителей Polycirrus medusa (также об этом можно судить по сравнительно большому весу выравниваний консенсусной последовательности с последовательностями данного вида).
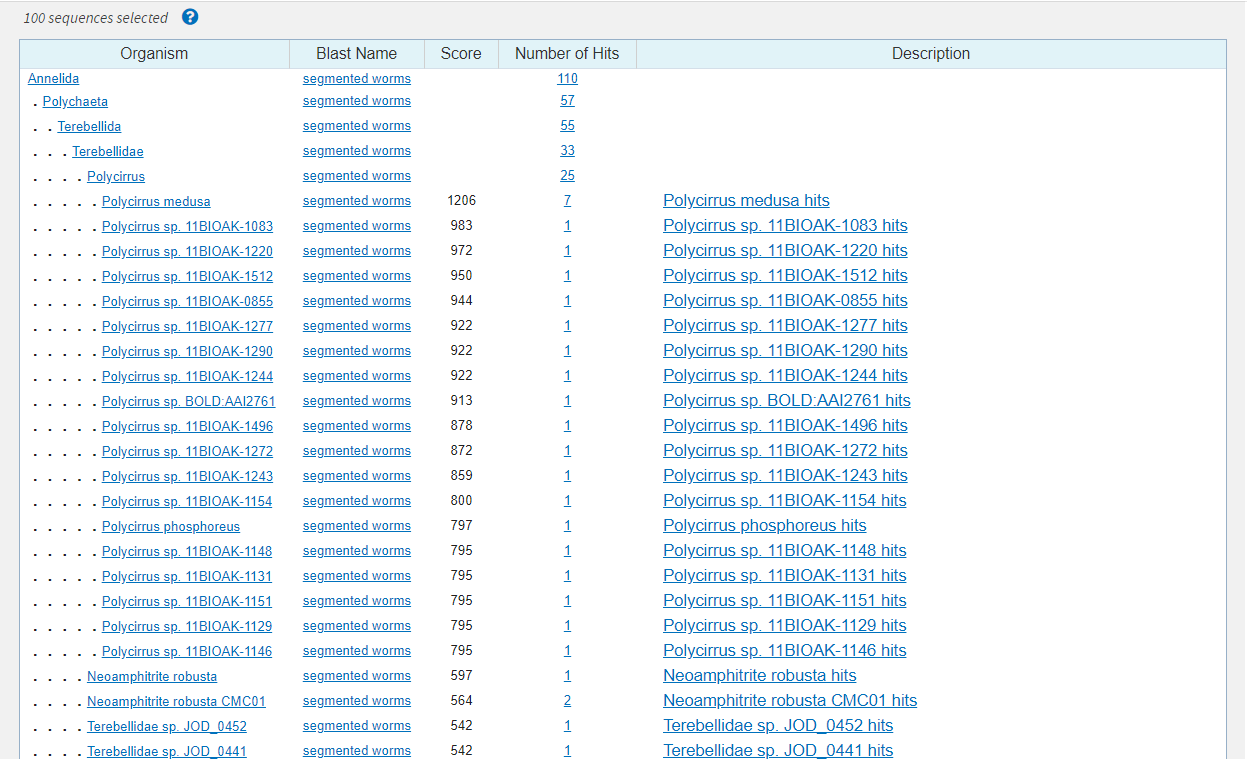
Рисунок 2. Общая таксономия
Было проведено сравнение трёх алгоритмов нуклеотидного BLAST. Поиск осуществлялся по консенсусной последовательности прошлого задания и по CDS вируса из прошлого практикума. Информация о задаваемых параметрах для BLAST и о количестве находок содержится в таблицах 1 и 2.
| Алгоритм | Word size | Match/Mismatch scores | Количество находок |
| megablast | 28 | 1, -2 | 16 |
| blastn по умолчанию | 11 | 2, -3 | 51 |
| blastn по умолчанию | 7 | 1, -4 | 53 |
Для консенсусной последовательности было выставлено ограничение по таксону Polycirrus (taxid:516039)с помощью исключения Polycirrus medusa (taxid:1037304). Выдача blastn отличилась от выдачи megablast на 35 находок, 11 из которых имели E-value, равное нулю, оставшиеся находки имели уже ненулевой E-value. С изменением параметров blastn добавилось ещё две плохие (с 1% покрытия) находки (к 7 имеющимся в blastn с параметрами по умолчанию).

Рисунок 3. Конец списка находок blastn с параметрами по умолчанию
Рисунок 4. Конец списка находок blastn с измененными параметрами
Из предыдущего практикума произвольно был выбран CDS вируса с GeneID: 40096679. Ограничением являлся род Sulfolobus (taxid:2284). Результаты вышли во многом аналогичными первой части задания. Изменение megablast (рисунок 5) на blastn по умолчанию (рисунок 6) привело к появлению лишь одной стоящей находки, так как большинство находок имело большой E-value. Уменьшение длины слова не принесло значимых находок.
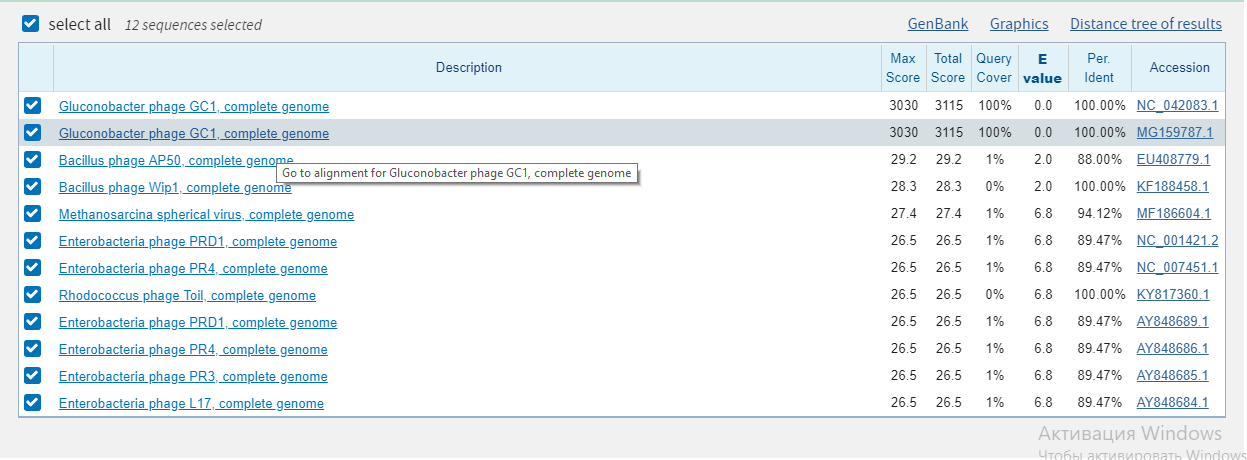
Рисунок 5. Находки megablast по CDS вируса
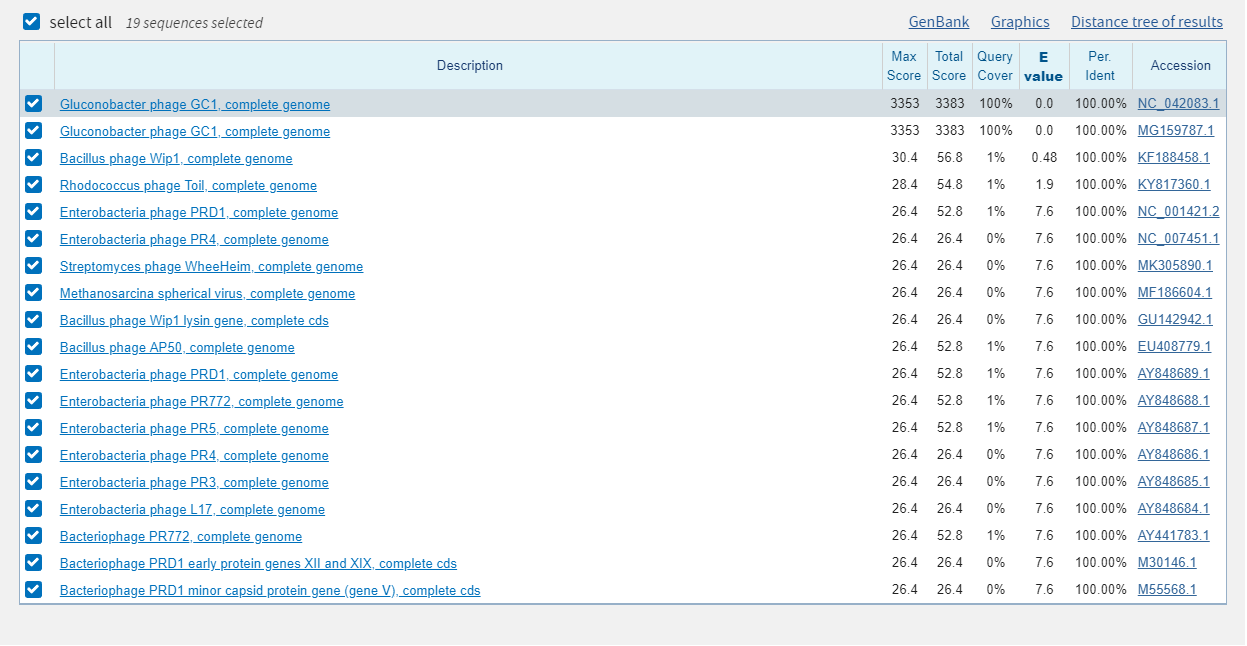
Рисунок 6. Находки blastn с параметрами по умолчанию по CDS вируса
Выбор алгоритма blast оказал небольшое влияние на выдачу значимых находок. Большинство находок было обнаружено еще megablast, blastn с параметрами по умолчанию добавил находки с покрытием около 1%, поэтому об их гомологичности запросу трудно судить. Уменьшение длины слова не принесло значимых результатов.
| Алгоритм | Word size | Match/Mismatch scores | Количество находок |
| megablast | 28 | 1, -2 | 12 |
| blastn по умолчанию | 11 | 2, -3 | 19 |
| blastn по умолчанию | 7 | 1, -4 | 34 |
Для задания были взяты 3 консервативных белка, информация о которых приведена ниже. Данные получены из UniProt.
Актин: ACT1_DICDI. Актины являются высококонсервативными белками для эукариот. Они ключевая составляющая в структуре цитоскелета и в процессе подвижности клетки.
Тубулин (бета-цепь): TBB5_HUMAN. Тубулин - основной компонент микротрубочек. Сложно вообразить эукариотичекую клетку без этого белка.
ДНК-лигаза дрожжей: A0A0L8VHL9_9SACH. У дрожжей отсутствуют гомологи ДНК-лизаз III млекопитающих, а гомолог ДНК-лигазы IV кодируется
геном DNL4/LIG4 и также участвует в негомологическом соединении концов ДНК.
Для выбранных белков был осуществлён поиск гомологов по предоставленной сборке генома. Используемые команды для BLAST+:
makeblastdb -in X5.fasta -dbtype nucl -out pr8_db
tblastn -query act.fasta -db pr8_db -out pr8_act.out
tblastn -query tbl.fasta -db pr8_db -out pr8_tbl.out
tblastn -query dld.fasta -db pr8_db -out pr8_dld.out
В таблице 3 приведена основная информация о результатах.
| TBB5_HUMAN | ACT1_DICDI | A0A0L8VHL9_9SACH | |
| Количество находок | 6 | 16 | 8 |
| Лучшая находка | unplaced-665 | scaffold-444 | scaffold-100 |
| Количество гэпов | 22 (5%) | нет | 44 (9%) |
| E-value лучшей находки | 0 | 0 | 6e-38 |
| Identity лучшей находки | 85% | 93% | 28% |
| Score лучшей находки | 1970 | 1911 | 154 |
| Вывод | Гомологичен | Гомологичен | Не гомологичен |
Процент покрытия у лучших находок первых двух белков (%95 у тубулина, 100% у актина), а также относительно высокие показатели идентичности дают нам возможность предположить, что в неаннотированном геноме закодированы гомологичные белки со схожими функциями. Напротив, имея высокий процент покрытия (91%), у ДНК-лигазы дрожжей низкий процент идентичности, что указывает на негомологичность лучшей находке.
Для поиска гена был взят произвольный контиг из сборки организма в предыдущем практикуме. Последовательность контига была взята в качестве запроса в blastx, ограничением были БД RefSeq и царство Metazoa. Полученная выдача представлена на рисунке 7.
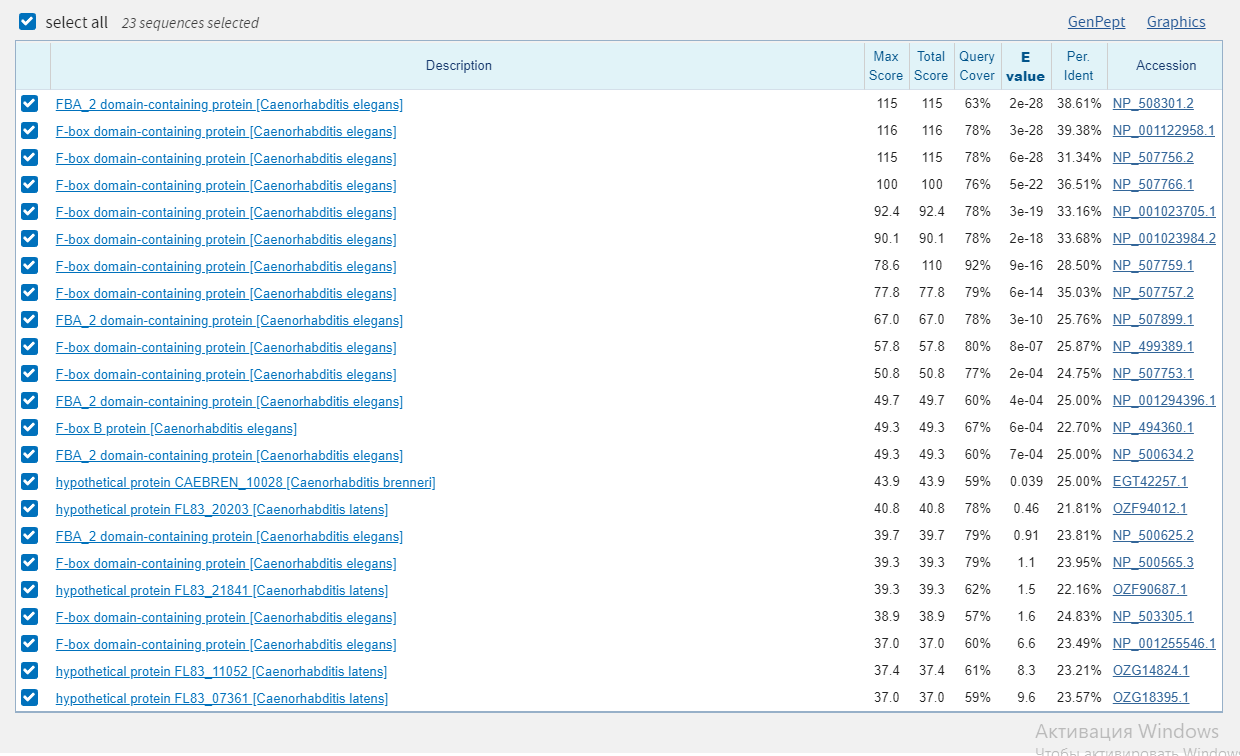
Рисунок 7.Результат поиска гена белка с помощью blastx
По полученным данным можно предположить, что в контиге содержится ген, кодирующий белок с F-box доменом. У растений многие F-box-белки участвуют в регуляции экспрессии генов и сайленсинге генов путём РНК-интерференции с микроРНК. Первый обнаруженный F-box-белок был одним из трёх компонентов SCF-комплекса, осуществляющего убиквитинирование белков, которые после этого разрушаются в протеосомах. Однако, низкие веса и процент идентичности, а так же немалые Е-vаlue не позволяют с достаточной уверенностью заявить о наличии данного гена в контиге.