Сборка генома de novo
Для того, чтобы скачать файл с чтениями, воспользовался следующей командой:wget ftp://ftp.sra.ebi.ac.uk/vol1/fastq/SRR424/000/SRR4240360/SRR4240360.fastq.gz
Скопируем адаптеры из директории ./NGS/adapters/ и объединим их в один файл с помощью команд:
cp /mnt/scratch/NGS/adapters/* /mnt/scratch/NGS/dimabosov/pr15/
cat *.fa >> adapters.fa
1. Подготовка чтений программой trimmomatic.
Удалим остатки адаптеров:java -jar /usr/share/java/trimmomatic.jar SE -threads 20 SRR4240360.fastq.gz trimadapt_SRR4240360.fastq.gz ILLUMINACLIP:adapters.fa:2:7:7 2> log_er.txt
После удаления адаптеров осталось 8212774 (99.49%) чтений.
Далее удалим с правых концов чтений нуклеотиды с качеством ниже 20, оставим чтения, длина которых не меньше 32 с помощью команды:
java -jar /usr/share/java/trimmomatic.jar SE -threads 20 trimadapt_SRR4240360.fastq.gz trim_SRR4240360.fastq.gz MINLEN:32 TRAILING:20 2> log_trim.txt
Судя по данным в log_trim.txt, осталось 8131995 (99.02%) чтений от чтений, оставшихся после удаления адаптеров.
Изменение размера файла:
До фильтрации - 194Mb
После удаления адаптеров - 193Mb
После удаления низкокачественных нуклеотидов и коротких чтений - 188Mb
2. Velveth.
Создадим k-меры наших чтений длиной 31 нуклеотид, использовав команду:velveth velveth 31 -fastq -short trim_SRR4240360.fastq.gz
-fastq - формат файла
-short - короткие чтения
velveth - директория для сборки.
3. Velvetg.
Запустим программу velvetg:velvetg velveth
Из вывода программы можно узнать, что значение параметра N50 = 43070.
Характеристика самых длинных контигов:
| Номер | Длина | Покрытие |
|---|---|---|
| 1 | 113474 | 33.528738 |
| 5 | 83603 | 33.649127 |
| 4 | 64155 | 35.850487 |
Контиг с ID 562 с длиной 2 имеет покрытие равное 1.
4. Анализ.
Я взял 3 самых длинных контига из прошлого пункта. Разберу их по отдельности(так проще и понятнее). Первый контиг имел ID 1, сравнил его с хромосомой Buchnera aphidicola имеющей AC - CP009253(то дано в задании) с помощью megablast: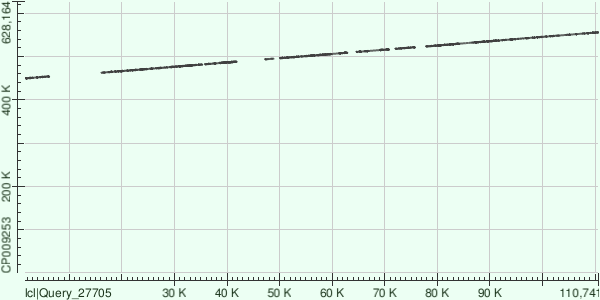
Координаты хромосомы, на которые ложится контиг с 449411 по 555905. Было найденно 15 диапазонов для выравнивания. Суммарно у них было 2791 гэп. Число однонуклеотидных различий - 68166. Хочется понять, что это за разрывы. Для этого проанализирую разрыв между 1 и 2 диапазонами. Контиг обрывается на 6226 нуклеотиде и во втором диапазоне начинается с 16117. Тогда как хромосома - 454тыс до 462тыс. Выходит, что это с большей вероятность неконсервативный участок, а не какая-нибудь делеция(хотя там можло всё что угодно произойти но с меньшей вероятностью). И между 6 и 7 диапазонами(45тыс. по горизонтальной оси) наблюдается нечто похожее(то есть там терпится разрыв в 6 тыс п.о. у обоих последовательностей).
E-value у этого выравнивая, как и 3 других - равно нулю.
Второй контиг:
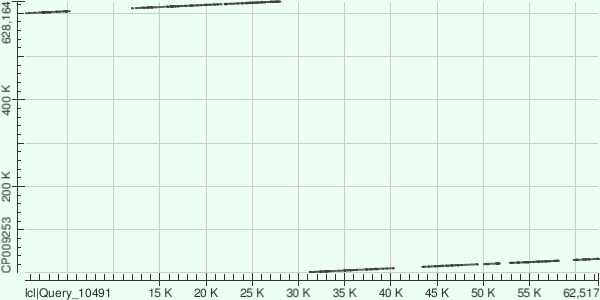
На первый взгляд - выглядит странно. Но если вспомнить, что рассматриваемый организм - бактерия, то становится очевидно, что данный график получен из-за кольцевой хромосомы.
По сути никакого разрыва, как это могло бы показаться, нет. 1407 гэпов и 36123 нуклеотидных замен.
Положение на геноме: с 599832 по 32745 нуклеотиды. 12 диапазонов.
Ну и последний - третий контиг:
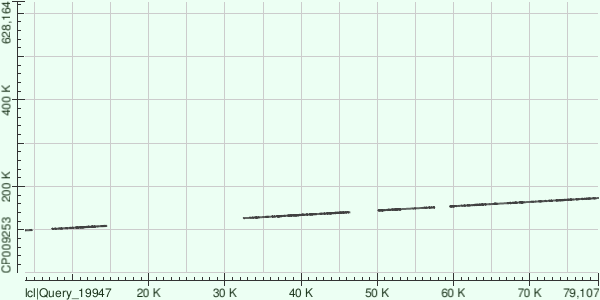
Он лёг на хромосому по координам: 98408 - 173180. Содержит 8 диапазонов, в которых 1561 гэп и 38145 однонуклеотидных замен.