1. Получение последовательности ДНК на основании данных, полученных из капиллярного секвенатора, и ее анализ

У нас было 2 хроматограммы: прямой последовательности - 03_F.ab1, и обратной -
03_R.ab1, веб платформы для работы с хроматограммами:
Pearl и Chromas. Помимо этого для выравнивая
двух последовательностей пришлось воспользоваться Jalview. Не то чтобы это был необходимый запас для прака, но раз уж начал делать биоинформатику, становится трудно остановиться.
Результат выравнивания с помощью Jalview в формате fasta по ссылке. Также Jalview выдал
выравнивание, по которому легко можно было сравнить и взаимодополнить последовательности: выравнивание.
Фаста файл конечным результатом: Ссылка.
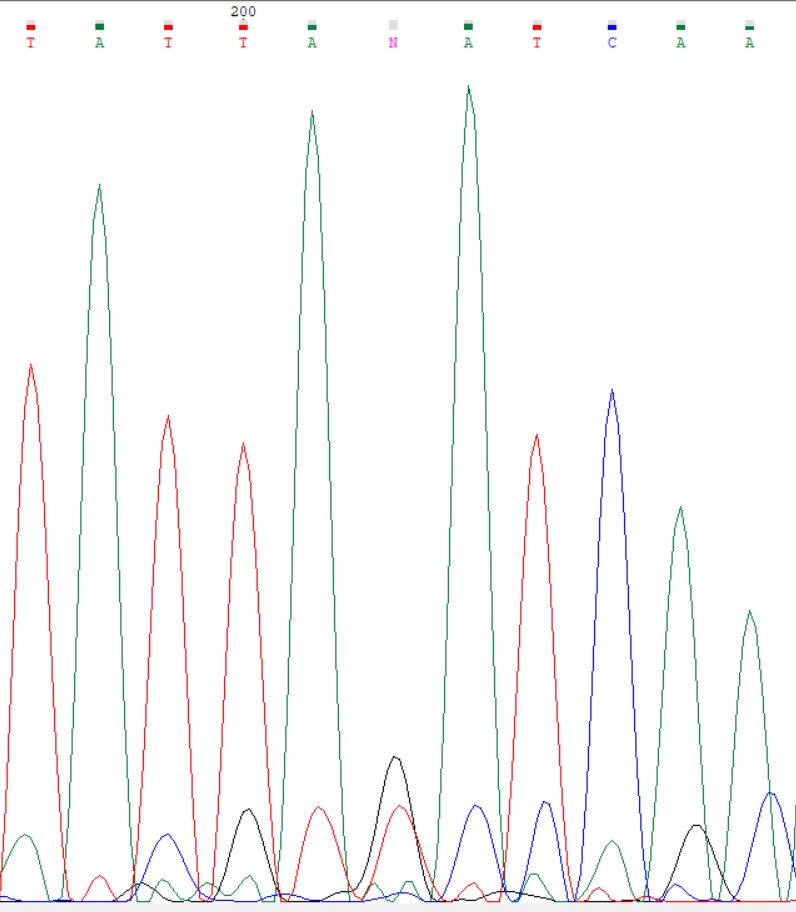

Слева- прямая последовательность, справа - обратная. Проблемный нуклеотид находился в прямой последовательности, а точнее - в 202 положении. Как я понимаю, секвенатор посчитал уровень сигнала слишком низким, чтобы зафиксировать в этой точке гуанин. Но благодаря обратной последовательности можно убедиться, что там действительно гуанин (в обратной последовательности, как можно заметить, он находится в 207 положении).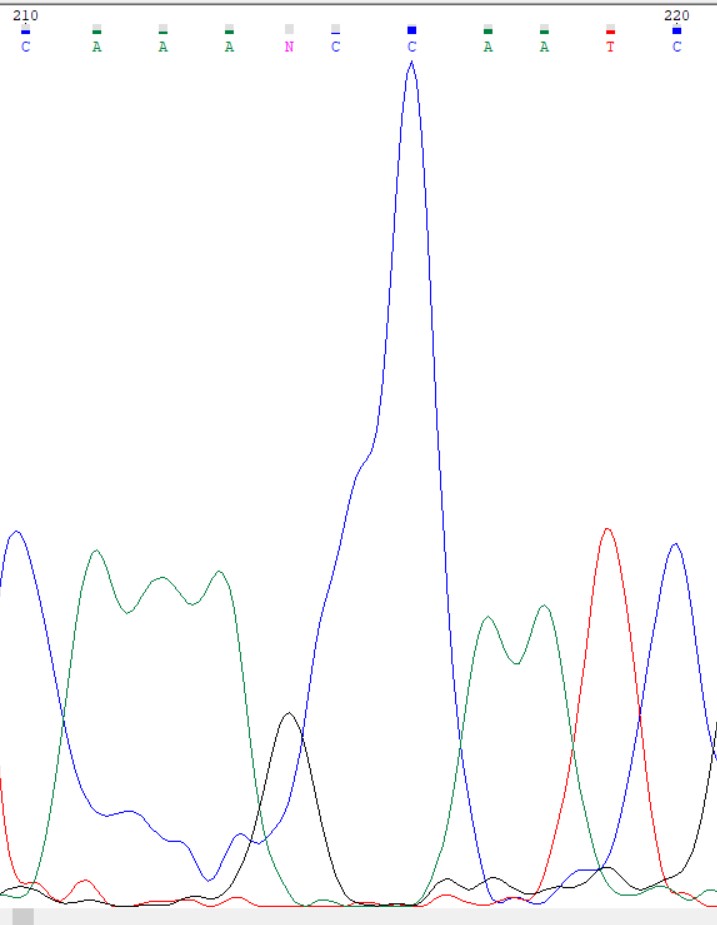

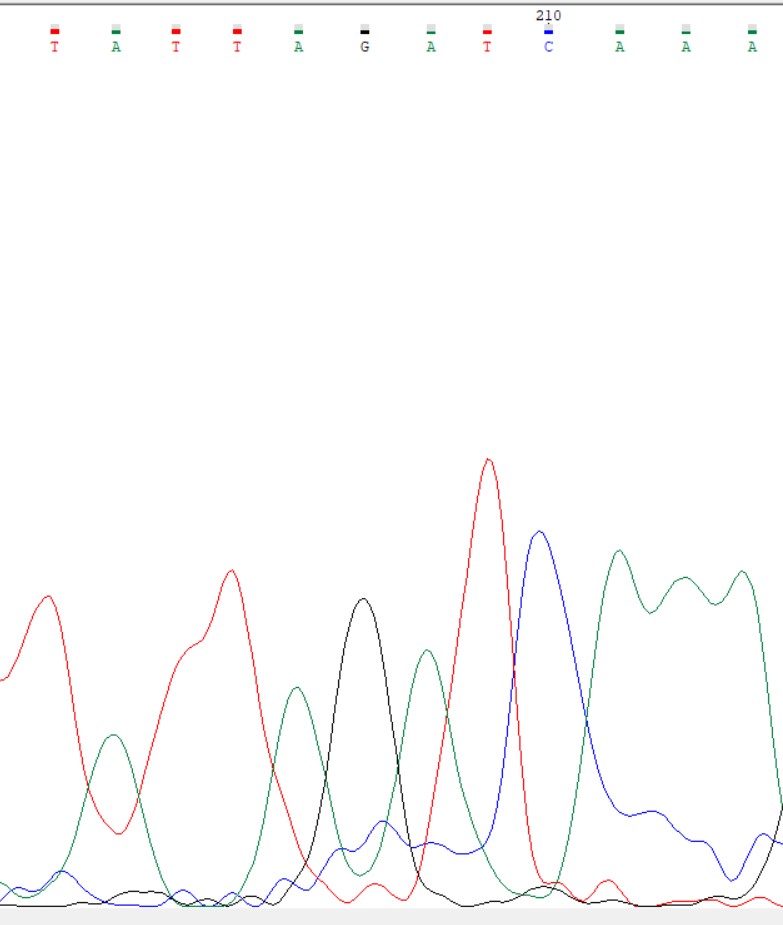
Теперь обратная последовательность слева. Проблема заключается в 214 положении. Ситуация похожа на то, что было с первым проблемным нуклеотидом. Опять не стал учитываться сигнал от гуанина. В прямой последовательности в 219 положении находится гуанин, и с помощью выравнивания полученного раннее, мы можем отредактировать нуклеотид.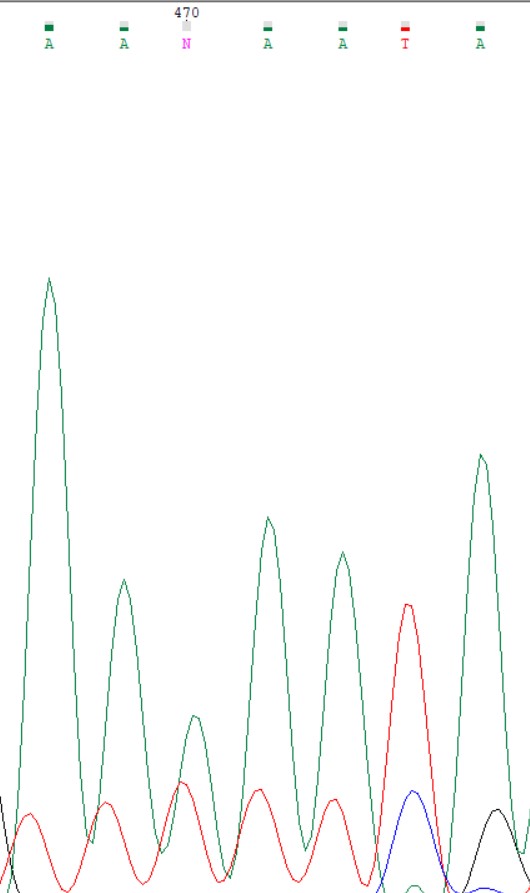

Слева - прямая последовательность ДНК, а справа - обратная. Достаточно интересные "скачки" красного на обоих хроматограммах. Хотелось бы предположить, что это полиморфизм, но подобные скачки в диапазоне 20 нуклеотидов достаточно часто встречаемы у обоих последовательностей. Предположу, что это либо ошибка при считывания последовательностей секвенатором, либо же обыкновенный шум.
В подтверждение моих слов - пара "скачков" на расстоянии приблизительно 5-15 от проблемного нуклеотида:
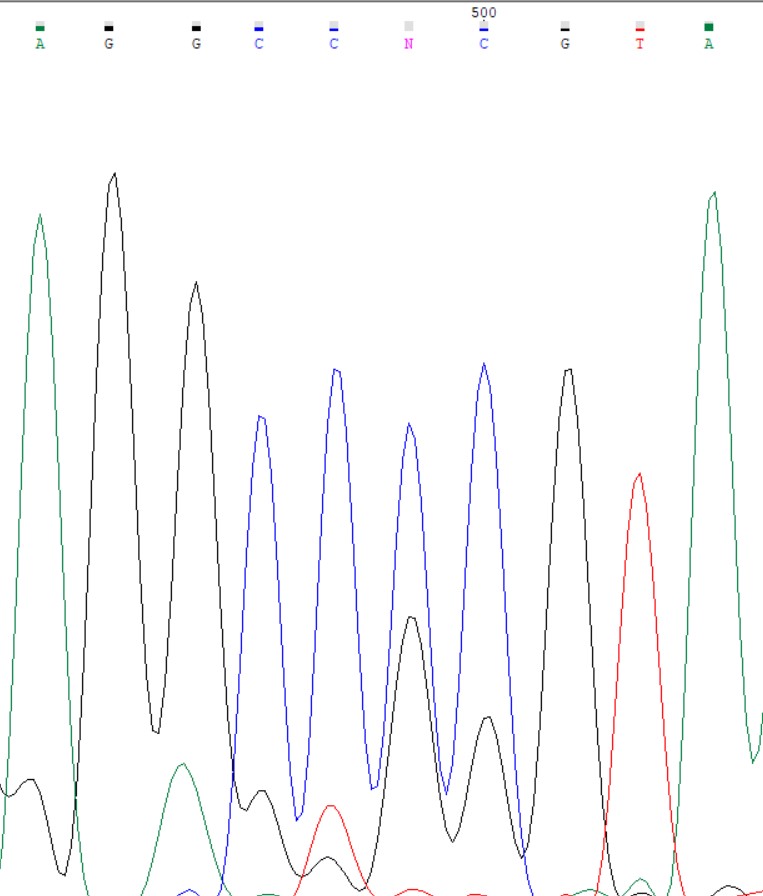

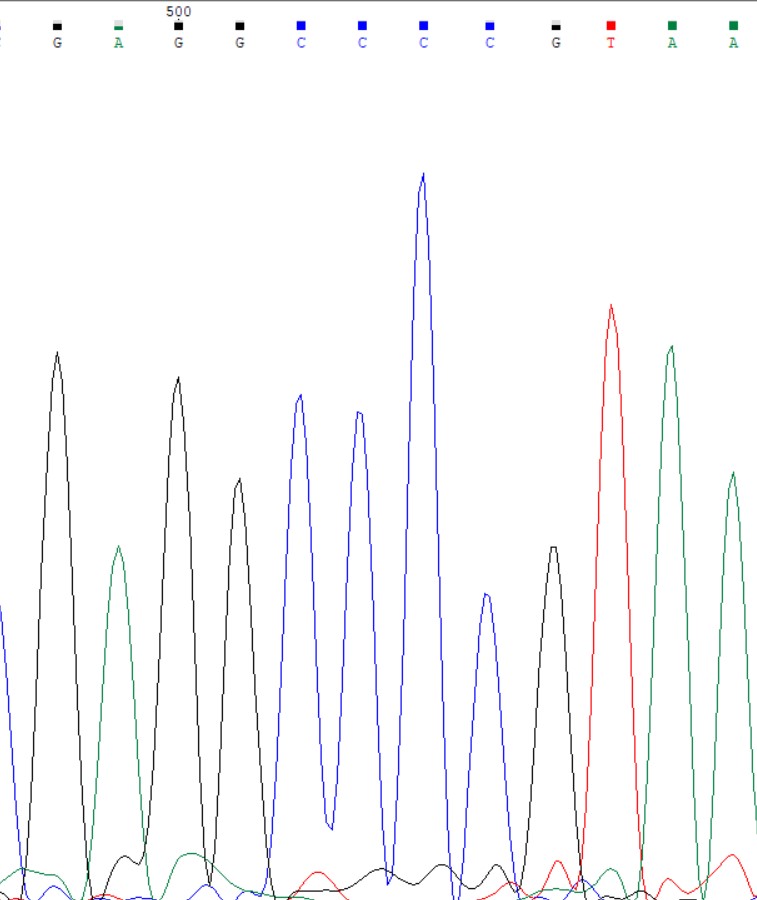
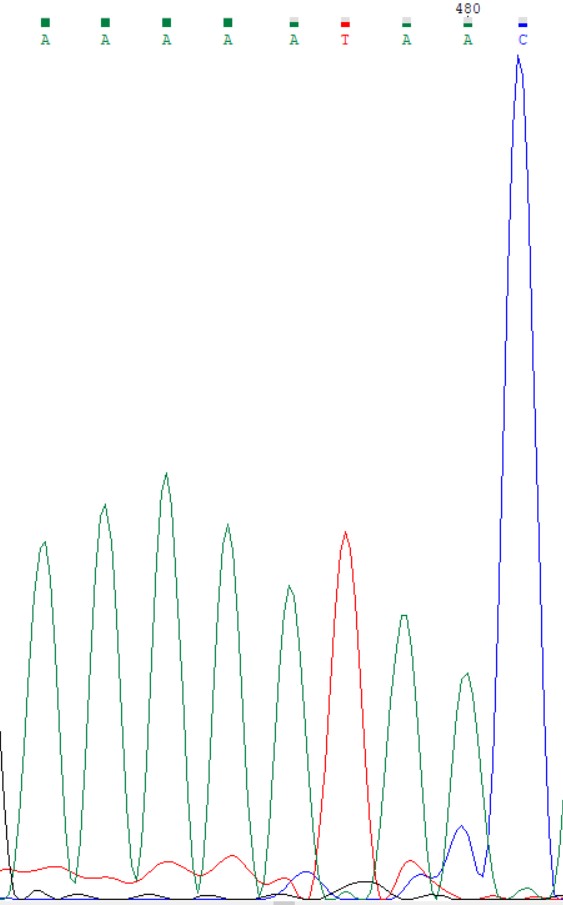
Прямая последовательность(слева) имеет в 499 положении неопределённый нуклеотид. Видимо, это из-за "вспышки" уровня гуанина и он посчитал вероятность цитозина (который как раз таки там и должен быть судя по обратной последовательности) низкой и предпочел оставить нуклеотид неопределённым.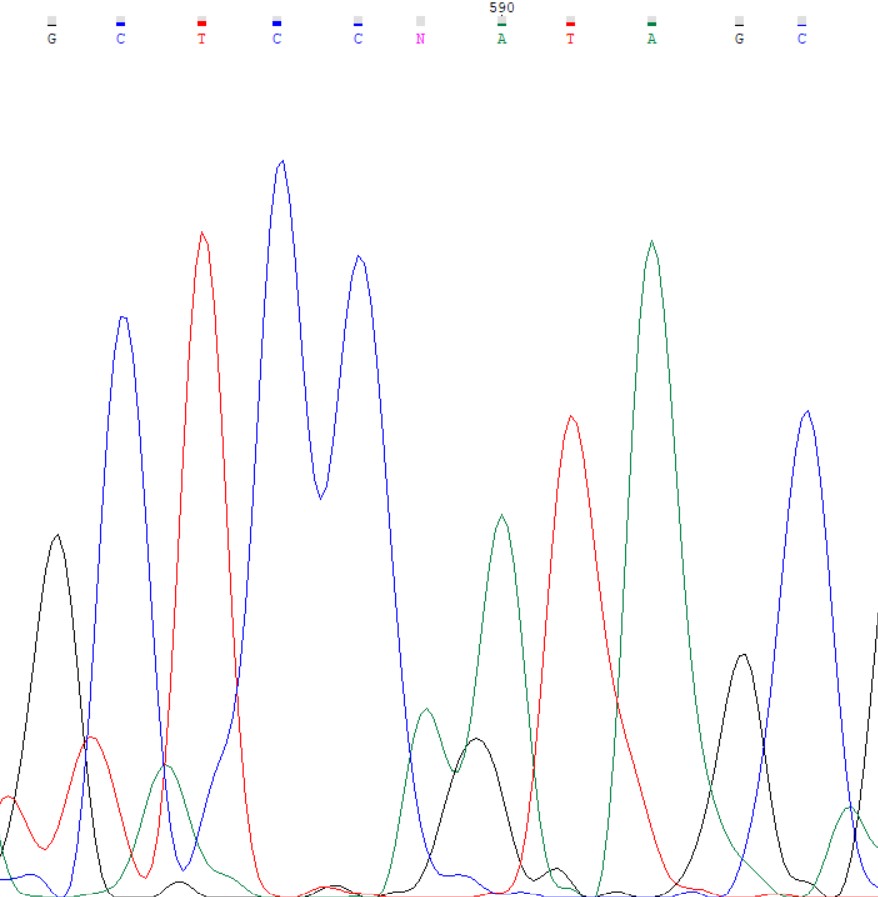

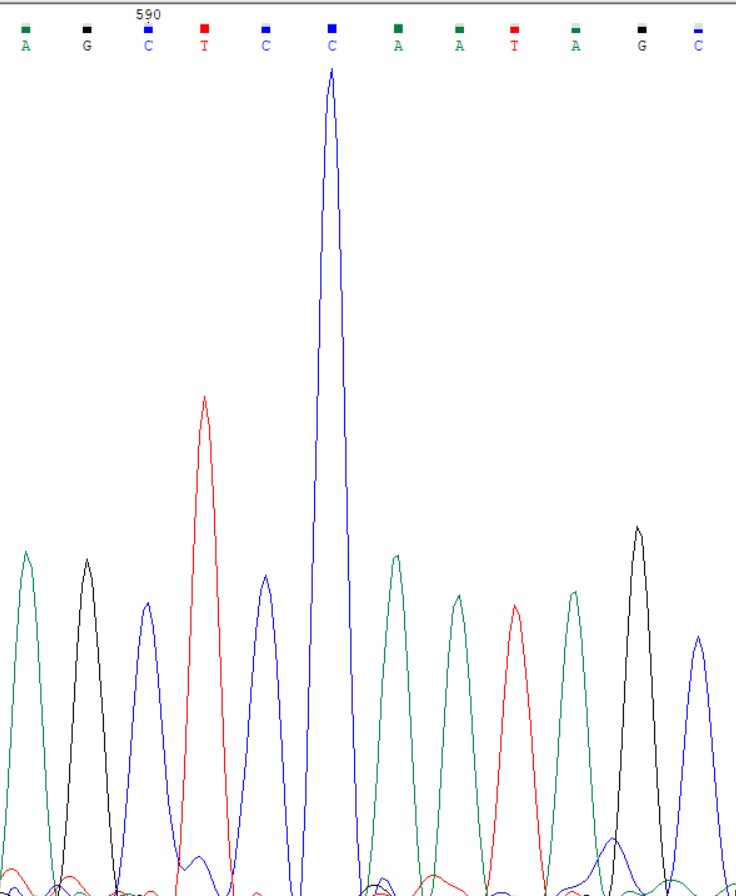
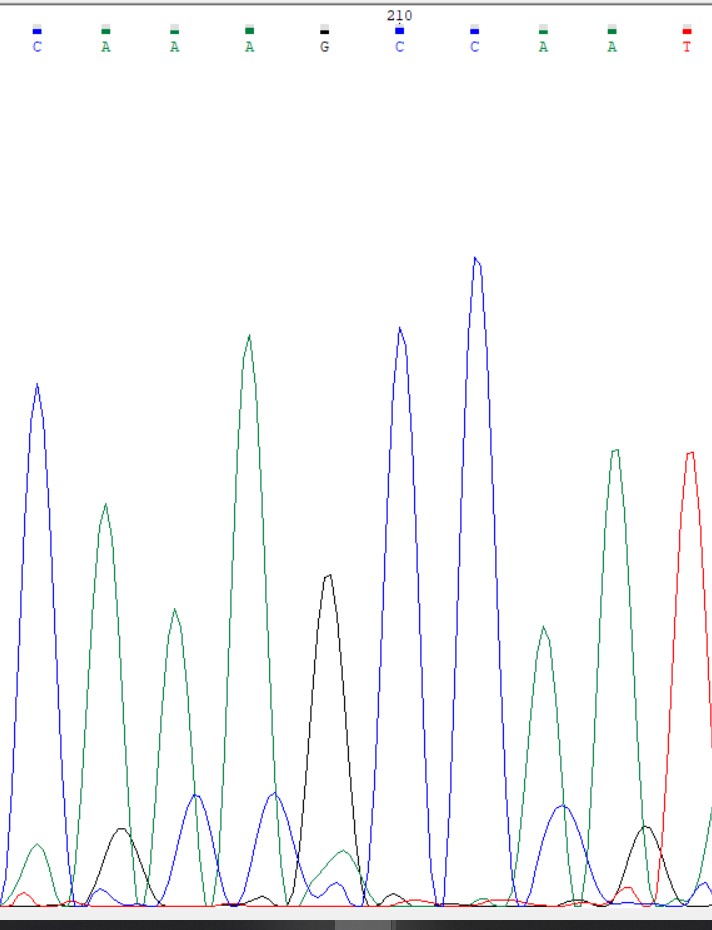
В прямой(слева) последовательности в 589 положении находится проблемный нуклеотид. Это связано с низким уровнем флюоресценции аденина(нам видна его вспышка, но, видимо, она не достигла того порогового уровня, который программа приняла бы как достаточно высокий, чтобы утверждать, что там именно аденин). Благодаря обратной последовательности мы можем доопределить, что это действительно аденин.
Качество хроматограммы определю по участку, который нам удалось распознать (поделив его длину на длину меньшей последовательности, т.е. - обратной(948). В результате получаем 504/948≈0.531
Но! Для более точной оценки качества хроматограммы, как мне кажется, стоит брать участок, который хорошо читается программой, т.е: 504/(948-197-33)≈0.7.
Так, по достаточно субъективной оценке, качество хроматограммы было примерно равно 70%.
На глаз могу предположить, что уровень сигнала превышает уровень шума примерно в 10-15 раз на основном (среднем) участке обоих последовательностей. А на всей последовательности это отношение примерно равно 5-10. Очень заметно, что уровень шума, относительно сигнала, достигает максимума в началах и концах последовательностей.
Интересный факт: Как я заметил на своих хроматограммах, для гуанина и аденина характерны большие "вспышки" флюоресценции, редко (почти не) встречающиеся у тимина и цитозина.
Если что, при анализе этих скачков я рассматривал обратную цепь в том виде, в каком она была получена с помощью секвенатора(т.е. не комплементарную)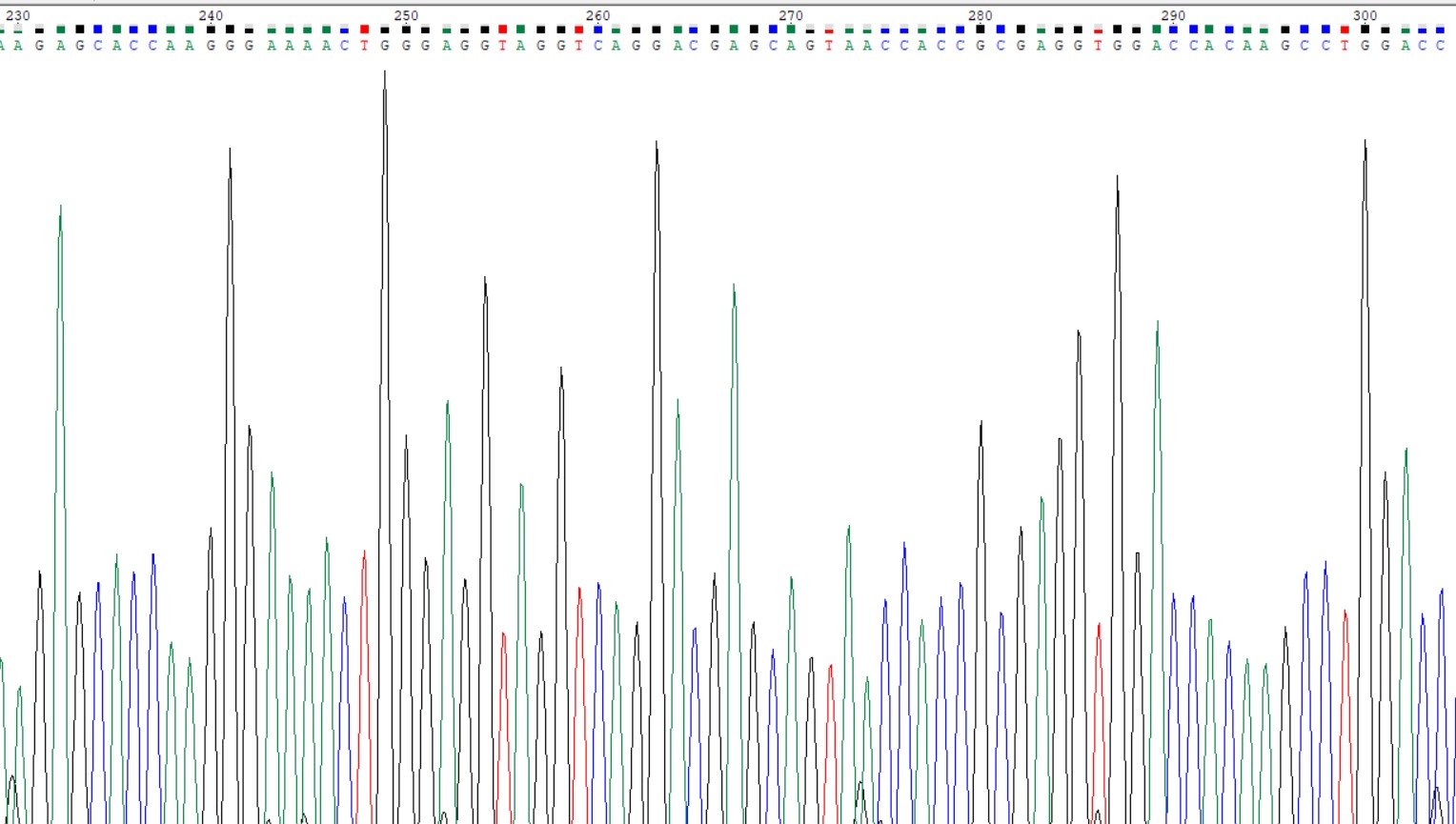

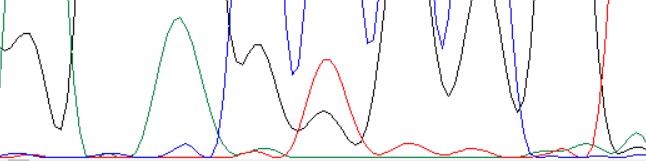
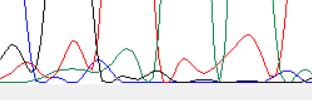
Что тут сказать... Всю хроматограмму совсем невозможно прочитать(даже какой-то её участок). Слева и справа от приведённого на картинке фрагмента только шум, в котором невозможно распознать что-либо. В самом фрагменте "всплеск" флюоресценции, который как и всю хроматограмму, невозможно прочесть. Саму хроматограмму можно скачать по ссылке.
Первый проблемный нуклеотид



Слева- прямая последовательность, справа - обратная. Проблемный нуклеотид находился в прямой последовательности, а точнее - в 202 положении. Как я понимаю, секвенатор посчитал уровень сигнала слишком низким, чтобы зафиксировать в этой точке гуанин. Но благодаря обратной последовательности можно убедиться, что там действительно гуанин (в обратной последовательности, как можно заметить, он находится в 207 положении).
Второй проблемный нуклеотид



Теперь обратная последовательность слева. Проблема заключается в 214 положении. Ситуация похожа на то, что было с первым проблемным нуклеотидом. Опять не стал учитываться сигнал от гуанина. В прямой последовательности в 219 положении находится гуанин, и с помощью выравнивания полученного раннее, мы можем отредактировать нуклеотид.
Третий проблемный нуклеотид



Слева - прямая последовательность ДНК, а справа - обратная. Достаточно интересные "скачки" красного на обоих хроматограммах. Хотелось бы предположить, что это полиморфизм, но подобные скачки в диапазоне 20 нуклеотидов достаточно часто встречаемы у обоих последовательностей. Предположу, что это либо ошибка при считывания последовательностей секвенатором, либо же обыкновенный шум.
В подтверждение моих слов - пара "скачков" на расстоянии приблизительно 5-15 от проблемного нуклеотида:


Четвёртый проблемный нуклеотид



Прямая последовательность(слева) имеет в 499 положении неопределённый нуклеотид. Видимо, это из-за "вспышки" уровня гуанина и он посчитал вероятность цитозина (который как раз таки там и должен быть судя по обратной последовательности) низкой и предпочел оставить нуклеотид неопределённым.
Пятый проблемный нуклеотид



В прямой(слева) последовательности в 589 положении находится проблемный нуклеотид. Это связано с низким уровнем флюоресценции аденина(нам видна его вспышка, но, видимо, она не достигла того порогового уровня, который программа приняла бы как достаточно высокий, чтобы утверждать, что там именно аденин). Благодаря обратной последовательности мы можем доопределить, что это действительно аденин.
Характеристика хроматограмм:
Длина начального и конечного трудно читаемого участка у прямой последовательности - 33 и 228, а для обратной - 33 и 197 нуклеотидов соответственно.Качество хроматограммы определю по участку, который нам удалось распознать (поделив его длину на длину меньшей последовательности, т.е. - обратной(948). В результате получаем 504/948≈0.531
Но! Для более точной оценки качества хроматограммы, как мне кажется, стоит брать участок, который хорошо читается программой, т.е: 504/(948-197-33)≈0.7.
Так, по достаточно субъективной оценке, качество хроматограммы было примерно равно 70%.
На глаз могу предположить, что уровень сигнала превышает уровень шума примерно в 10-15 раз на основном (среднем) участке обоих последовательностей. А на всей последовательности это отношение примерно равно 5-10. Очень заметно, что уровень шума, относительно сигнала, достигает максимума в началах и концах последовательностей.
Интересный факт: Как я заметил на своих хроматограммах, для гуанина и аденина характерны большие "вспышки" флюоресценции, редко (почти не) встречающиеся у тимина и цитозина.
Если что, при анализе этих скачков я рассматривал обратную цепь в том виде, в каком она была получена с помощью секвенатора(т.е. не комплементарную)

2. Пример нечитаемого фрагмента хроматограммы.

Что тут сказать... Всю хроматограмму совсем невозможно прочитать(даже какой-то её участок). Слева и справа от приведённого на картинке фрагмента только шум, в котором невозможно распознать что-либо. В самом фрагменте "всплеск" флюоресценции, который как и всю хроматограмму, невозможно прочесть. Саму хроматограмму можно скачать по ссылке.