Поиск сайтов разрывной транскрипции sgmRNA в геноме коронавируса HCoV-EMC/2012
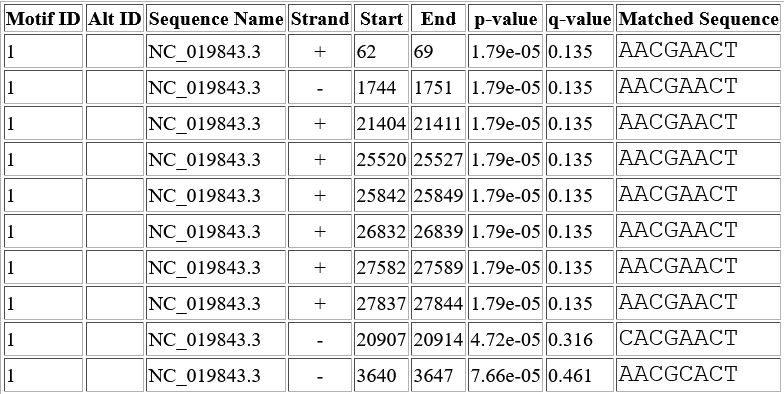
Для выполнения этого задания был взят PWM из 6.2. Я использовал программу FIMO.Получена следующая таблица:
Сравним с родственным короновирусом
Для ближайших родственников нет полного генома (поэтому из того же штамма, к сожалению, рассмотреть не удалось). Поэтому пришлось искать по subgenus(подрод)
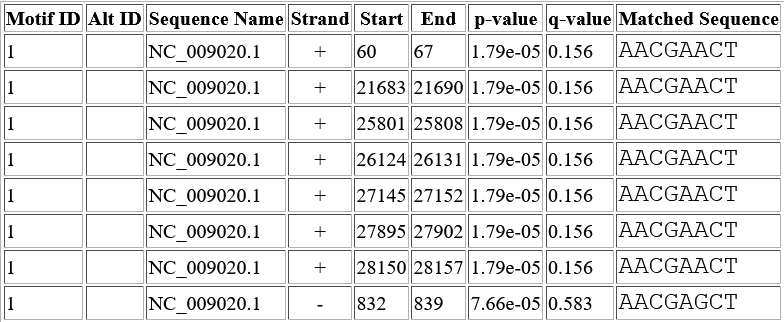
Результат для Pipistrellus bat coronavirus HKU5(Ссылка на NCBI)
Для HCoV-EMC/2012 из задания 6.2 было найдено 10 сигналов. Интересно, что один из генов расположен внутри гена полипротеина. Но он на другой цепи. Возможно это перекрывающиеся гены.
Что же касается Pipistrellus bat coronavirus HKU5, то у него совпало количество идентифицированных генов и количество находок.
LOGO для последовательностей Козак поздних генов коронавируса:

Рассмотрим, насколько сигнал специфичен для вида. Запустим FIMO для Tylonycteris bat coronavirus HKU4. Было найдено несколько нахдок с большим p-value, что означает, что данная последовательность специфична для данного вида.