HMM
Взял PF01687 СсылкаВыбранная доменная архитектура (HAD_2 и Flavokinase), представленна по Pfam в 307 последовательностях:

В uniprot сделал запрос: database:(type:pfam PF13419) database:(type:pfam PF01687):
Нашло 466 белка. Скачал таблицу с белками: Ссылка
Посмотрим на их длину:
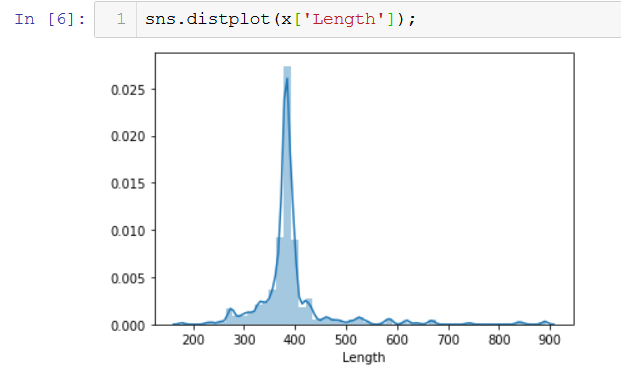
Теперь при помощи запроса: database:(type:pfam pf13419) database:(type:pfam pf01687) length:[350 TO 400] получим следующую таблицу: Ссылка
Новая гистограмма длин: (просто интересно стало)
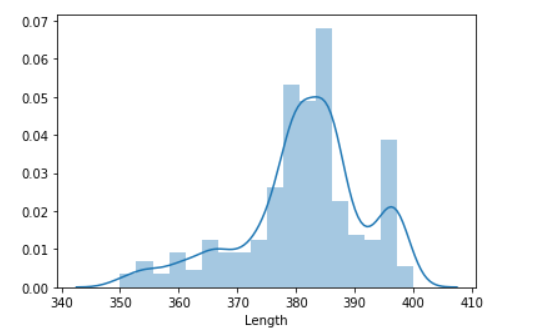
Их слишком много. Уменьшил границы до 380-384, и убедившись, что это неродственные организмы (посмотрел находки) скачал их. Теперь у меня 74 находки.
Теперь мы работаем именно с этими белками.
Написав скрипт, який викачуе послідовности Ссылка
Теперь мы объединим все последовательности и применим к ним выравнивание:
cat *.fa* > seq.fasta
muscle -in seq.fasta -out align.fasta
Как и полагается, вот ссылки ссылки:seq.fasta
align.fasta
Теперь обработаем наше выравнивание в JalView. Удалим блоки на N- и C- концах, а также удалю сильно отличающиеся последовательности.
after.fa
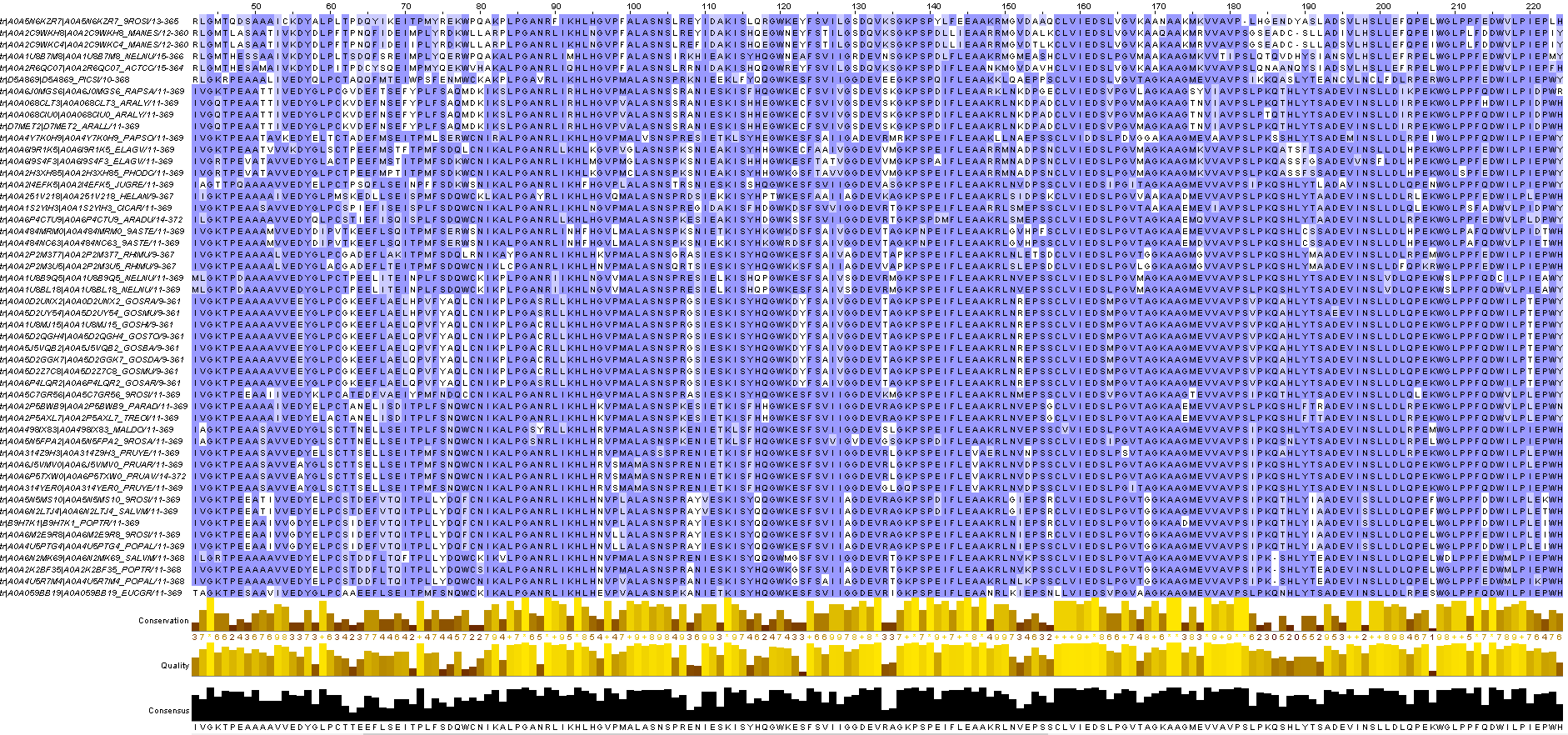
После геноцида осталось 58 последовательностей. Вот фрагмент из JalView:
Потом я запустил вот это:
hmm2build hmm.hmm after.fa
hmm2calibrate hmm.hmm
Вот что вышло из этой затеи: Ссылка на hmm.hmmПроверим наш hmm профиль)
Скачаю все белки с доменом PF01687 из UniProt (таких 40,687).
Вот файл Ссылка
Проверим по hmm-профилю командой:
hmm2search -E 0.01 hmm.hmm all.fa > result.txt
Полученный файл: Ссылка
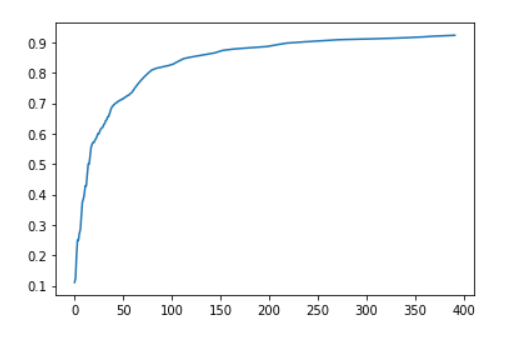
Был построен график весов выравниваний, для того, чтобы потом определить порог E-value включения белка в семейство.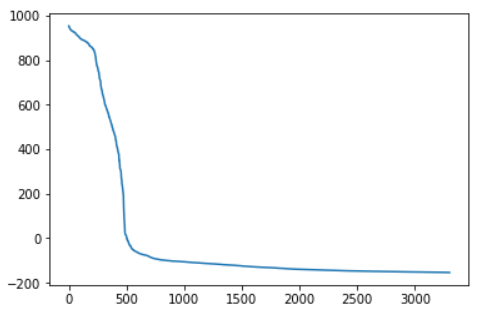
Выбрал порог, равный 378.9.
| uniprot+ | uniprot- | |
|---|---|---|
| hmm+ | 316 | 2986 |
| hmm- | 150 | 37701 |