На сайте http://www.ebi.ac.uk/ в строку поисковика писал "полное название бактерии" complete genome. Для каждой бактерии нашлась такая запись. Собственно по самой записи искал строку, содержащую информацию о 16S рРНК (Cntrl+F). В ней смотрел координаты и вырезал командой seqret -sask1 embl:"ID записи". Указываю координаты, если в записи стоит отметка complement, то использую опцию reverse.
| Название бактерий | Координаты 16S рРНК | Идентификатор генома в embl |
| Bacillus subtilis: | 9810 - 11364 | AL009126 |
| Bacillus anthracis: | 9315 - 10854 | CP002091 |
| Lactococcus lactis: | 511423-512971 | AM406671 |
| Streptococcus pneumoniae: | 16806-18218 | FM211187 |
| Clostridium tetani: | 41801-43309 | AE015927 |
| Clostridium botulinum: | 9246-10747 | FR773526 |
| Listeria monocytogenes: | 247258-248814 | CP003414 |
Выравнивание данных последовательностей:
Дерево, полученное программой dnaml (критерий максимального правдоподобия):
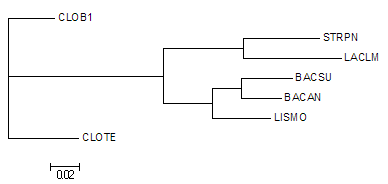
Топологически - полное совпадение с "белковым".
Еще я построил дерево программой dnapars:
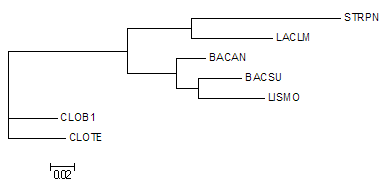
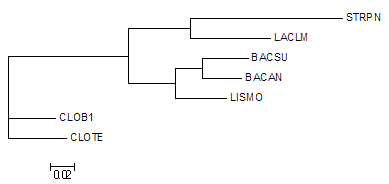
Получились 2 дерева, несколько различающиеся топологией. На первом - есть неправильная ветвь {bacsu;lismo}vs.{bacan,laclm,strpn,clob1,clote}, при этом отсутствует правильная ветвь {bacsu,bacan}vs.{lismo,laclm,strpn,clob1,clote}
В первую очередь я "вытащил" из файла proteo.fasta все белки для каждой конкретной бактерии (команда seqret proteo.fasta:*_STRPN извлечет все белки Streptococcus pneumoniae, полученный файл называл по мнемонике организма). Далее по каждому протеому создал базу данных (команда makeblastdb -in strpn -dbtype prot -out strpn_prot). Далее проводил BLAST-ом по БД (команда blastp -query clpx_bacsu -db strpn_prot -out strpn_out -evalue 0.001).
Дальше получил матрицу эволюционных расстояний с помощью fprotdist, а затем укорененное дерево программой fneighbor, алгоритм UPGMA.
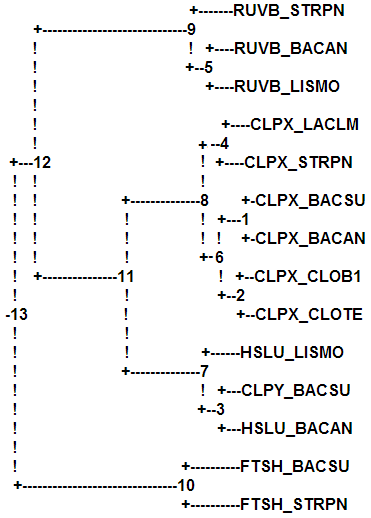
Паралоги: RUVB_STRPN, FTSH_STRPN, CLPX_STRPN; RUVB_BACAN, CLPX_BACAN, HSLU_BACAN и т.д. Ортологи, например: RUVB_BACAN, RUVB_LISMO; CLPX_CLOB1, CLPX_CLOTE; HSLU_LISMO, CLPY_BACSU.