Atlas of contacts|Атлас контактов
Last update on the 28th of February, 2017
Download all scripts as an archive.
Авторы
Дмитрий Мыларщиков, Семен Галкин.
Обзор
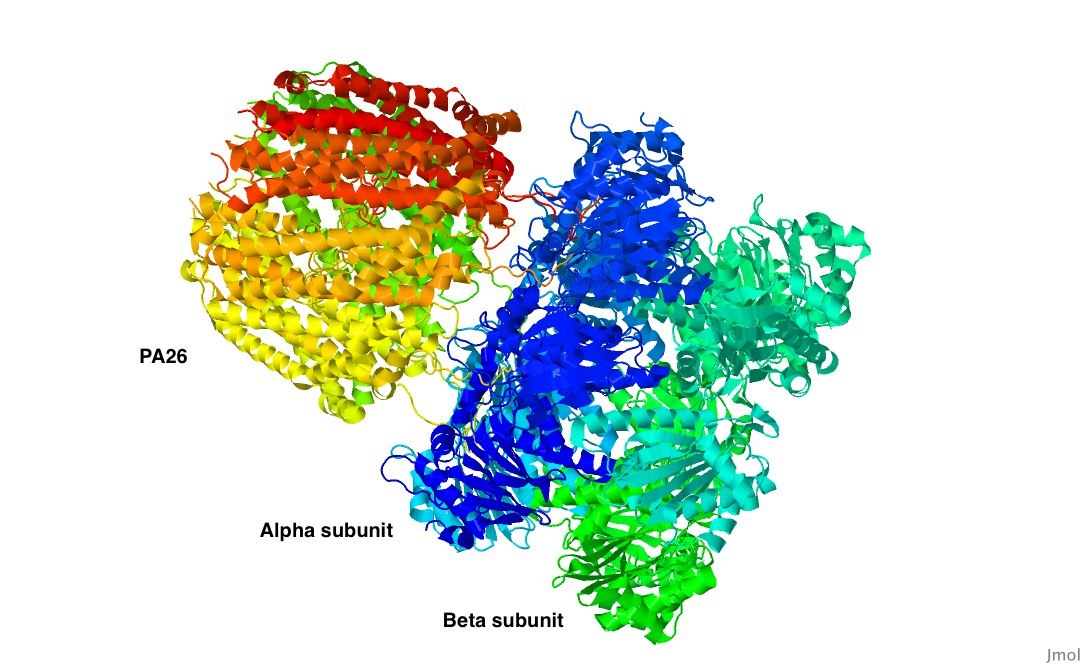
Исследуемый белок - 20s архейная протеасома в комплексе с активирующим её белком PA26 из Trypanosoma brucei[1]. Основные сведения об исследуемом объекте:
- PDB ID: 1YAR;
- Uniprot ID: P28061, P25156, Q9U8G2[2];
- Относится к классам гидролаз (альфа- и бета-субъединицы) и гидролаз-активирующих белков (PA26);
- Состоит из трёх субъединиц (рис. 1), 21 цепи;
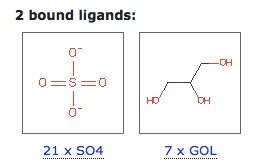
- Основные лиганды - глицерин (7 шт) и сульфат-ион (21 шт) (рис. 2)[3];
- Альфа-субъединица: длина 233 аминокислоты, цепи: A, B, C, D, E, F, G[4];
- Бета-субъединица: длина 217 аминокислот, цепи: H, I, J, K, L, M, N[5];
- PA26: длина 237 аминокислот, цепи: O, P, Q, R, S, T, U[6];
- Разрешение структуры в PDB – 1.9 Å.
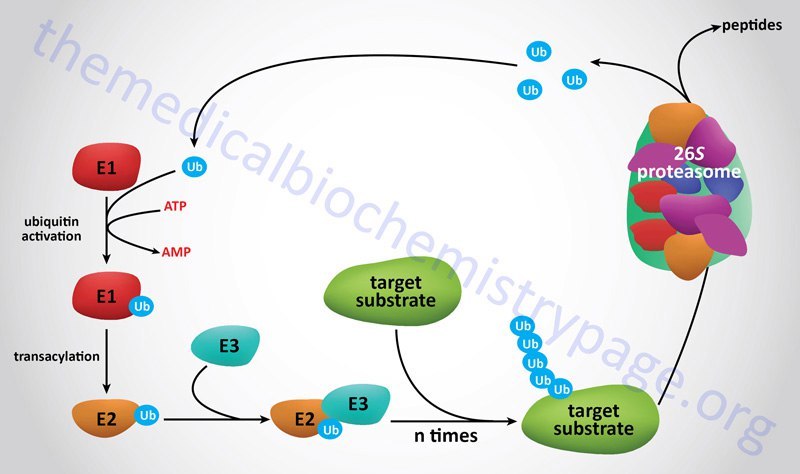
Протеасома - это белковый комплекс, который играет очень важную роль в жизни клетки, осуществляя контролируемую деградацию белка в клетке. В клетке этот белок представлен в виде гомодимера[7]. Белки, которым суждено попасть в протеасому, помечаются специальным белком убиквитином[8]. Такая система деградации белков называется убиквитин-зависимой, её краткая схема приведена на рисунке 3 (посредством белков e1,e2,e3 осуществляется полиубиквитинирование).
Белок-белковые контакты
Скрипт: ProteinКовалентные связи
Согласно результатам работы данного скрипта не обнаружено ни одной ковалентной связи между цепями и, очевидно, половинами протеасомы. Вследствие того, что нековалентные взаимодействия широко представлены, представляется возможным сказать, что протеасома является довольно гибкой структурой в отношении сборки и функционирования.
Водородные связи
Межмолекулярные водородные связи широко представлены в изучаемом объекте. Для того, чтобы их найти посредством Jmol, мы 1) применили команду "set hbondsRasmol FALSE", чтобы оказалось возможным найти водродные связи, образованные боковыми цепями аминокислот; 2) с помощью скрипта просмотрели различные комбинации субъединиц протеасомы (цепей и половин) на наличие между ними водородных связей.
Неудивительно, что пространственная структура каждой "полуединицы" стабилизирована большим количеством межмолекулярных водородных связей, в основом с участием боковых радикалов аминокислот. Объединение двух "полуединиц" обеспечено 141 водородной связью согласно алгоритму Jmol. По одному примеру для каждого взаимодействия приведены в апплете.
Солевые мостики
Солевые мостики возникают за счёт электростатического притяжения двух противоположно заряженных аминокислот, Glu- и Asp- против Arg+ и Lys+, если расстояние между центрами зарядов не больше 4 Å[10]. Принимая во внимание эти требования, мы разработали несколько скриптов, чтобы пронаблюдать предполагаемые солевые мостики. Было обнаружено 28 солевых мостиков между "полуединицами", большинство из которых не перекрывалось с водородными связями. Межцепочечные солевые мостики в пределах одной "полуединицы" практически не представлены: в каждом предполагаемом взаимодействии участвует в среднем по три атома от каждой цепи. Эти данные были получены посредством данного скрипта.
Напрашивается очевидный вывод: солевые мостики не вносят значительного вклада в стабилизацию четвертичной структуры протеасомы, нерегулярные проявления, возможно, возникли в результате нативного процесса сборки комплекса или несовершенства алгоритма поиска. Два предполагаемых солевых мостика показаны в апплете.
Гидрофобные взаимодействия
Сервис CluD[11] показал наличие 69 гидрофобных ядер (ссылка на скрипт), самое крупное из которых содержит 7446 атомов (рис. 4). В целом, ядра располагаются практически равномерно по всей протеасоме, что наводит на мысли о том, что гидрофобные структуры используются в основном для поддержания структуры (рис. 5), но если внимательнее приглядеться, можно заметить, что большая часть гидрофобных структур экспонирована в полость белка (рис 6). Возможно, такое строение протеасомы позволяет более эффективно расщеплять белки.
Лиганд-белковые контакты
Скрипт: LigandГлицерин
- IUPAC: Пропан-1,2,3-триол;
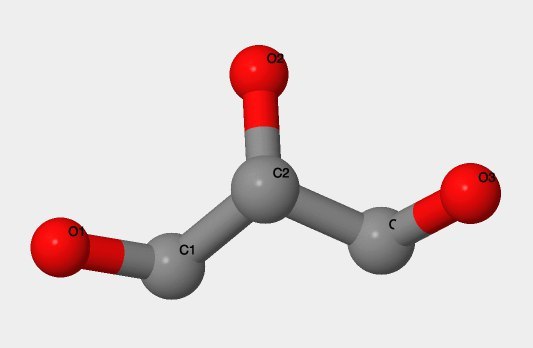
- Химическая формула: см. рис. 7;
- Брутто-формула: C3H8O3;
- Молярная масса: 92 г/моль;
- PubChem ID: 753.
Роль
Удалось найти исследование[12], обнаружившее, что добавление экзогенного глицерина усиливало активность протеасом. Однако кажется маловероятным, что глицерин оказывает непосредственное регулятивное воздействие на протеасому: скорее всего он играет роль поддержания структуры. Но данное утверждение является лишь предположением.
Связи
Глицерин не связывается ковалентно с белком, образование солевых мостиков при физиологических значениях pH невозможно. Но водородные связи с белком присутствуют и показаны в скриптах апплета.
Сульфат-ион
- IUPAC: сульфат-ион;
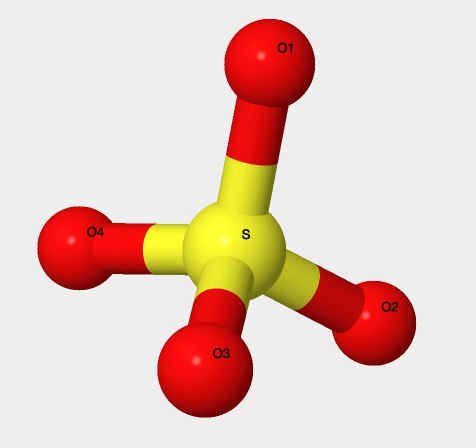
- Химическая структура: см. рис. 8;
- Брутто-формула: SO42-;
- Молярная масса: 96 г/моль;
- PubChem ID: 1117.
Роль
К сожалению, не удалось найти литературы, связанной с ролью сульфат-иона в протеасоме, но возможно предположить, что за счет образуемых водородных связей данная молекула способна стабилизировать структуру белка. Или же появление этих молекул в pdb файле связано с процессом получения данных о структуре (например, методика кристаллизации белка подразумевает использование сульфат-ионов).
Связи
Водородные связи с белком обнаружены и отображены средствами JSMol на сайте, солевых мостиков обнаружено не было (fлгорим поиска: множество атомов [GLU].OE1 и [ARG].CZ на расстоянии менее 4 Å от иона, обнаруженная связь не должна являться уже рассчитанной водородной).
Личный вклад авторов
Дмитрий работал над белок-белковыми контактами за исключением гидрофобных взаимодействий, разработал соответствующие скрипты, оформил отчёт и разместил его на своём сайте;
Семён работал над лиганд-белковыми контактами и гидрофобными взаимодействиями, разработал соответствующие скрипты, написал обзор и создал иллюстрации.
Источники
- Yi Yao et al, Structural and Functional Characterizations of the Proteasome-activating Protein PA26 from Trypanosoma brucei, The Journal Of Biological Chemistry, Vol. 274, No. 48, Issue of November 26, pp. 33921–33930, 1999;
- записи в базе данных Uniprot;
- страница структуры в PDB;
- альфа-субъединица в Uniprot;
- бета-субъединица в Uniprot;
- PA26 в Uniprot;
- статья в Википедии;
- статья в Википедии;
- источник;
- статья в Протеопедии;
- CluD;
- Deocaris CC et al, Glycerol stimulates innate chaperoning, proteasomal and stress-resistance functions: implications for geronto-manipulation, Biogerontology. 2008 Aug;9(4):269-82.
Authors
Dmitry Mylarshchikov, Simon Galkin.
Review
The studied object is 20S proteasome from Thermoplasma acidophilum and activating protein PA26 from Trypanosoma brucei[1]. Brief information about this protein complex:
- PDB ID: 1YAR;
- Uniprot ID: P28061, P25156, Q9U8G2[2];
- Belongs to hydrolase (alpha and beta subunits) hydrolase-activating proteins (PA26);
- Consists of three subunits (Fig. 1), 21 chains;
- Main ligands: glycerol (7 molecules) and sulfate ion (21 molecules) (Fig. 2)[3];
- Alpha subunit: 233 amino acids length, chains: A, B, C, D, E, F, G[4];
- Beta subunit: 217 amino acids length, chains: H, I, J, K, L, M, N[5];
- PA26: 237 amino acids length, chains: O, P, Q, R, S, T, U[6]:
- PDB structure resolution: 1.9 Å.


Proteasome is a protein complex that plays a huge role in cell’s life by exercising controlled protein degradation. In vivo protein exists in dimerized form[7]. Proteins that have to be cleaved are tagged with ubiquitin protein[8]. This system of protein cleavage is called "Ubiquitin-dependent protein degradation". Its brief scheme is presented in Fig. 3 (proteins e1,e2,e3 realize polyubiquitination).

Protein-protein contacts
Script: ProteinCovalent bonds
According to the results of a tricky script there are no covalent bonds between any chains and, obviously, semi-proteasomes. Thus, as non-covalent interactions are presented, it is possible to say that the proteasome is a flexible complex in terms of self-assembling and functionality.
Hydrogen bonds
Intermolecular hydrogen bonds are widely presented in studied object. In order to find them in Jmol, we 1) applied "set hbondsRasmol FALSE" attribute to make it possible to find hydrogen bonds with sidechains involved; 2) looked through various combinations of proteasome units (chains and semi-proteasomes).
There is no surprise that the structure of each semi-proteasome is stabilized with a huge ammount of hydrogen bonds, mainly sidechains involved. The connection of two moieties is provided with 141 hydrogen bonds according to Jmol algorithm. Single examples of both (interchain and interhalf) interactions are displayed in applet.
Salt bridges
Salt bridges occur between two opposit charged aminoacid sidechains, Glu- and Asp- versus Arg+ and Lys+, if the distance between the centers of the charge is less then 4 Å[10]. Taken into account these conditions, we've developed several scripts with an eye to observe putative salt bridges. There were discovered 28 putative salt bridges between semi-proteasomes, almost all of which are not interferred with hydrogen bonds. Regarding the interchain connections, there are fewer putative bridges. The average ammount of atoms involved in such type of connection is about 3 from each chain in each case. This information were extracted from the result of this script's work.
Overall, the obvious conclusion is derived: salt bridges do not make significant contribution in stabilizing the quaternary structure of proteasome, sporadic cases might be originated from native assembling process and/or imperfection of algorithm. Two putative cases are presented in the applet.
Hydrophobic interactions
Using CluD[11] service our group recognized that protein has 69 hydrophobic cores (script), the biggest one consists of 7446 atoms (Fig. 4). Generally, hydrophobic cores are almost uniformly distributed throughout the proteasome. It suggests that main hydrophobic cores function is a structure one (Fig. 5). But it is not hard to observe that plenty of cores are located inside the protein cavity (Fig. 6). This kind of protein construction may let proteasome to split proteins in more effective way.



Ligand-protein contacts
Script: LigandGlycerol
- IUPAC: Propane-1,2,3-triol;
- Structure: fig. 7;
- Brutto-formula: C3H8O3;
- Molar mass: 92 Da;
- PubChem ID: 753.
Role
Our group found an article saying that addition of exogenous glycerol may increase proteasomes activity[12]. But it seems quite unbelievable that glycerol may be a regulating molecule. So, we suggest that it could just support protein structure.
Bonds
Glycerol only forms hydrogen bonds with some protein amino acids (look it up in the applet). Salt bridges are impossible at physiological pH values.

Sulfate ion
- IUPAC: sulfate ion;
- Structure: fig. 8;
- brutto-formula: SO42-;
- Molar mass: 96 Da;
- PubChem ID: 1117.
Role
Unfortunately, we couldn’t find any articles saying about sulfate ion role in proteasome complex, but we can suggest that this ion may stabilize protein structure by creating hydrogen bonds with protein amino acids. Also, sulfate ions could appear in the structure due to the methods of protein crystallization or other processes of protein preparation.
Bonds
Hydrogen bonds were found and displayed in JSMol applet. Salt bridges were not found (search algorithm: atoms [GLU].OE1 and [ARG].CZ found within 4 Å the ion, putative bond must be not hydrogen one at the same time).

Personal contribution
Dmitry produced all protein-protein contacts work except for hydrophobic interactions, developed appropriate scripts, developed and hosted the report on his website;
Simon produced protein-ligand contacts, hydrophobic interactions, developed appropriate scripts, composed the overview and made all pictures.
References
- Yi Yao et al, Structural and Functional Characterizations of the Proteasome-activating Protein PA26 from Trypanosoma brucei, The Journal Of Biological Chemistry, Vol. 274, No. 48, Issue of November 26, pp. 33921–33930, 1999;
- records in Uniprot;
- structure page in PDB;
- alpha subunit in Uniprot;
- beta subunit in Uniprot;
- PA26 in Uniprot;
- article on Wikipedia;
- article Wikipedia;
- credits;
- article on Proteopedia;
- CluD;
- Deocaris CC et al, Glycerol stimulates innate chaperoning, proteasomal and stress-resistance functions: implications for geronto-manipulation, Biogerontology. 2008 Aug;9(4):269-82.