1. Вступление
Halobacterium salinarum – аэробный галлофильный микроорганизм, встречающийся в гиперсоленых местообитаниях. По современным филогенетическим данным систематика данного организма такова (таблица 1) [1]:
| Филлум: | Euryarchaeota |
|---|---|
| Класс: | Halobacteria |
| Отряд: | Halobacteriales |
| Семейство: | Halobacteriaceae |
| Род: | Halobacterium |
| Вид: | Halobacterium salinarum |
H. salinarum приспособился к обитанию в микроаэрофильных условиях. В связи с этим развился механизм бактериородопсинового фотосинтеза [2]. Экспрессируется белок бактериородопсин, который при попадании квантов света изменяет свою конфигурацию и функционирует как протонный насос. Образовавшийся протонный градиент может быть ис-пользован для работы АТФ-синтазы.
Из-за обитания чрезвычайно соленых условиях у H. salinarum существуют приспособления для выживания. Так, например, для снижения осмотического стресса экспрессируются несколько активных переносчиков для закачивания калия в клетку [3]. Для предотвращения высаливания белков, архея кодирует преимущественно кислые белки (средняя изоэлектрическая точка равна 5,03) [4].
В засоленных местообитаниях слабая защита от УФ-излучения. Для защиты развита сложная система репарации, а так же присутствует пигмент бактериоруберин, обуславливающий красный цвет микроорганизма и способный действовать как антиоксидант [5].
H. salinarum невероятный организм по своим приспособлениям к экстремальным метам обитания и интересный для изучения.
2. Материалы и методы
Генетические данные по микроорганизму получены из базы данных. {1}
Для анализа таблиц и построения гистограмм использовались Google sheets {2}.
Анализ данных проводился на языке программирования Python и Bash.
3. Результаты
Построена гистограмма{1} длин белков археи (рис. 1).
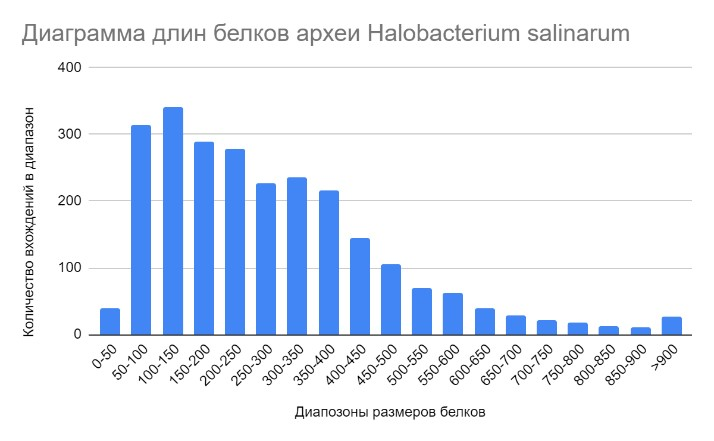
При сортировке таблицы были получены следующие значения для последовательностей, встречающихся в геноме археи (таб-лица 2). Преобладающее число – белок кодирующие последовательности. Интересно, что количество различных тРНК меньше, что согласуется с Воббл-гипотезой Крика [6] о том, что нуклеотид на 1 позиции в антикодоне тРНК можент не соответствовать строгим Уотсон-Криковским взаимодействиям.
| gene | 2582 |
|---|---|
| CDS | 2529 |
| tRNA | 48 |
| rRNA | 3 |
| ncRNA | 2 |
На основе анализа хромосомы и плазмид {2} археи была по-строена Таблица 3.
| seq_type | chromosome | CDS | tRNA | rRNA | ncRNA |
|---|---|---|---|---|---|
| chromosome | - | 2283 | 48 | 3 | 2 |
| plasmid | pHSAL1 | 151 | 0 | 0 | 0 |
| plasmid | pHSAL2 | 95 | 0 | 0 | 0 |
Из полученных данных следует, что большинство белок кодирующих генов находится в хромосоме. При этом в плазмиде отсутствуют тРНК, рРНК и некодирующие РНК, что говорит о том, что гены плазмиды специализированных под конкретных адаптации. Поэтому мы видим в плазмидах только белок-кодирующие последовательности.
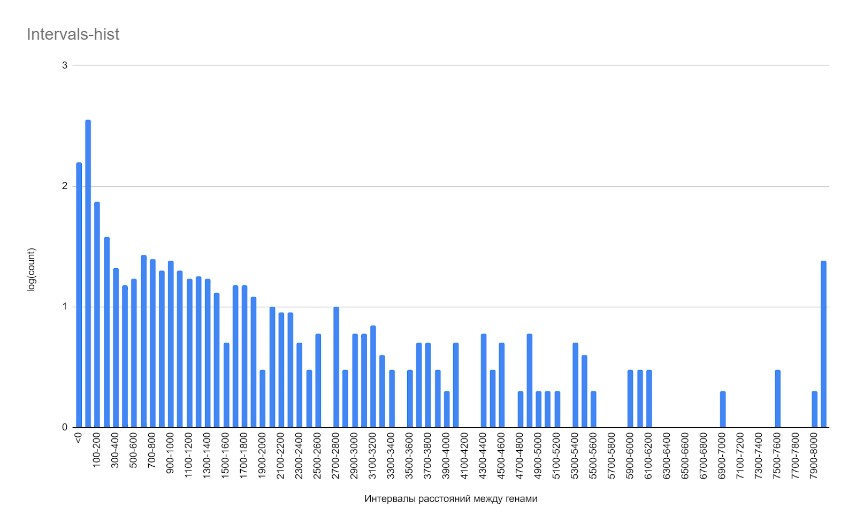
Гистограмма (рис.2) отражает межгенные расстояния в геноме археи. Наиболее распространенное межгенное расстояние находится в интервале 0-200. Расстояния более 5000 нуклеотидов встречаются редко, поэтому можно сказать, что в геноме археи короткие межгенные последовательности, что говорит о тенденции к компактизации генома. Так же наблюдается значительное количество межгенных расстояний длина которых меньше 0, что говорит о том, что есть гены, рамки считывания которых перекрываются. Это интересно, так как одни и те же участки генома находятся в разных генах, что может говорить о том, что в случае совпадения рамок считывания эти участи могут нести схожие функции, в продуктах этих генов.
Архея – галофильный организм, поэтому ожидалась высокая доля GC. Полученные данные подтверждают это. Доля GC в хромосоме археи составляет 67% что значительно больше ожидаемых 50% – среднего значения, если все нуклеотиды встречаются с одинаковой частотой. Высокий GC состав стаби-лизирует геном в экстремально соленых условиях.
4. Литература
[1] https://en.wikipedia.org/wiki/Halobacterium_salinarum
[2] Oesterhelt, D; Stoeckenius, W (1973). "Functions of a new photoreceptor membrane". Proceedings of the National Academy of Sciences of the United States of America. 70 (10): 2853–7. Bibcode:1973PNAS...70.2853O. doi:10.1073/pnas.70.10.2853. PMC 427124. PMID 4517939.
[3] Ng, W. V.; Kennedy, S. P.; et al. (2000). "Genome sequence of Halobacterium species NRC-1". Proceedings of the National Academy of Sciences. 97 (22): 12176–81. Bibcode:2000PNAS...9712176N. doi:10.1073/pnas.190337797. PMC 17314. PMID 11016950.
[4] Kozlowski, LP (26 October 2016). "Proteome-pI: proteome isoelectric point database". Nucleic Acids Research. 45 (D1): D1112–D1116. doi:10.1093/nar/gkw978. PMC 5210655. PMID 27789699.
[5] Ide, H., Takeshi, S., Hiroaki, T., Studies on the antioxidation activity of bacte-rioruberin, Urakami Found Mem, 1998, 6:127–33.
[6] Crick, F.H.C. (August 1966). "Codon—anticodon pairing: The wobble hy-pothesis" (PDF). Journal of Molecular Biology. 19 (2): 548–555. CiteSeerX 10.1.1.693.2333. doi:10.1016/S0022-2836(66)80022-0. PMID 5969078. Ar-chived (PDF) from the original on 4 March 2016. Retrieved 31 October 2015.