Задание 1. Поиск консервативных мотивов в выравнивании
В данном практикуме выполнялся поиск консервативных мотивов в выравнивании домена PF00217 (ATP:guanido phosphotransferase, C-terminal catalytic domain). В выравнивании seed имеется 53 последовательностей, в Swiss-Prot 136 находок с этим доменом. Было получено выравнивание последовательностей домена в формате fasta. При пороге идентичности 94 был выбран мотив NEED[HQ].R. При поиске по выравниванию данный паттерн находится в 46 последовательностях (из 53). Находок паттерна в других участках выравнивания нет. Таким образом мы нашли достаточно консервативный и специфичный паттерн. Также важно заметить, что паттерн содержит заряженные аминокислоты, а так же гистидин. Такое содержание аминокислот намекает на функцию связанную с позиционированием субстрата внутри белка.
Кодировка паттерна из формата Jalview была переведена в формат prosite N-E-E-D-[HQ]-x-R Был запущен поиск паттерна по базе данных Swiss-Prot при помощи сервиса ScanProsite. Было найдено 139 совпадений для 136 белков. Важно заметить, что белок содержащий 2 совпадения имеют длину в 2 раза больше чем остальные (KARG_ANTJA длина белка 715 ак, содержит 2 вхождения паттерна. Белок вероятно димер, так как обычная длина белков данного семейства порядка 350 ак). Белок содержащий 3 совпадения - тример (KCRF_STRPU). Найденный паттерн имеет хорошую консервативность для данного семейства белков и является необходимой частью их структуры, так как был найден во всех аннотированных белках данного семейства.
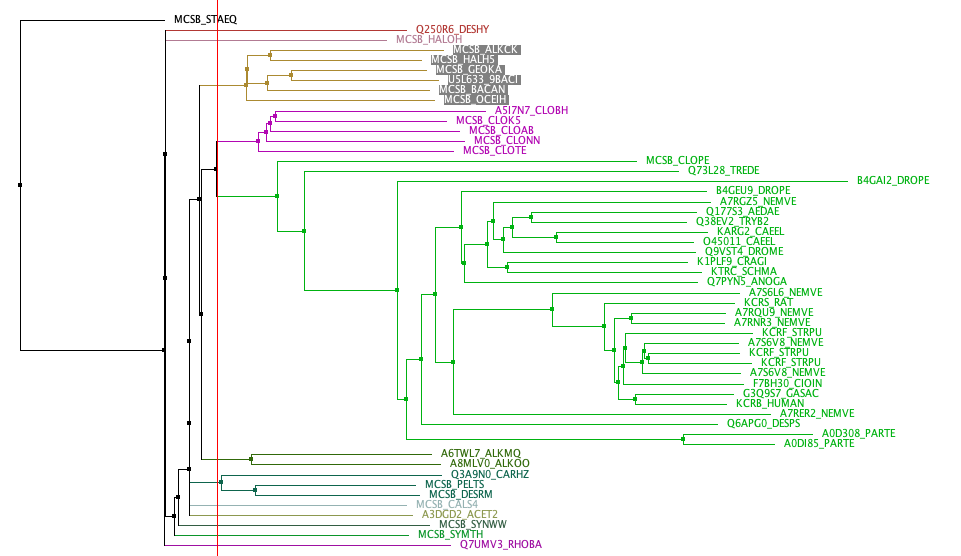
2. Поиск мотива, специфичного для одной клады филогенетического дерева
Было построено филогенетическое дерево, метод NJ (рис. 1). Выбранная клада содержит 6 последовательностей. Найдем какой-нибудь специфичный для нее паттерн. В качестве паттерна была предложена последовательность S[EG].E..S. Для данной последовательности был выполнен поиск по выравниванию. Нашлось 8 совпадений: 6 в выделенной каладе, 1 нахдока в MCSB_SYMTH на тех же координатах что и в целевой группе (возможно артефакт построения дерева), 1 находка в случайном месте выравнивания. Можно считать что данный мотив специфичен для выбранной клады.
3. PSI-BLAST
Был выбран идентификатор P47908, данный белок является фактором способствующим гибернации рибосом. Основная его функция -- уменьшение белкового синтеза для сохранения энегии в условиях клеточного стресса. Для данного белка был запущен алгоритм PSI-BLAST по базе данных swiss-prot. Результаты представлены в таблице 1. На итерации 3 не было найдено новых последовательностей, значит наступила стабилизация результата. Разница между максимальным и минимальным e-value сократилась. Все найденные белки имеют в названии слово рибосомальный и имеют связь с покоем (гибернацией) рибосом. В результате поиска удалось найти гомологи искомого белка.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
|---|---|---|---|---|---|
| 1 | 24 | P30334.1 | 0.004 | P47908.2 | 3e-143 |
| 2 | 28 | P9WMA8.1 | 3e-06 | P47908.2 | 5e-88 |
| 3 | 28 | P24694.1 | 4e-21 | P47908.2 | 4e-82 |
4. Поиск мотивов de novo с помощью MEME
Для поиска мотивов были получены последовательности из задания 1 в формате fasta (предварительно из последовательностей были удалены символы "-"). Запуск программы meme осуществлялся следующей командой:
meme pr10_1.fasta -o meme_res -minw 4 -maxw 10 -nmotifs 2
Было найдено 2 мотива со значимыми e-value (Выдача программы). Среди мотивов был найден ранее показанный мотив из выравнивания. Он был найден в 50 из 53 последовательностей (рис 2). Программа MEME отработала хорошо, мотивы из выравнивания были независимо найдены de novo.

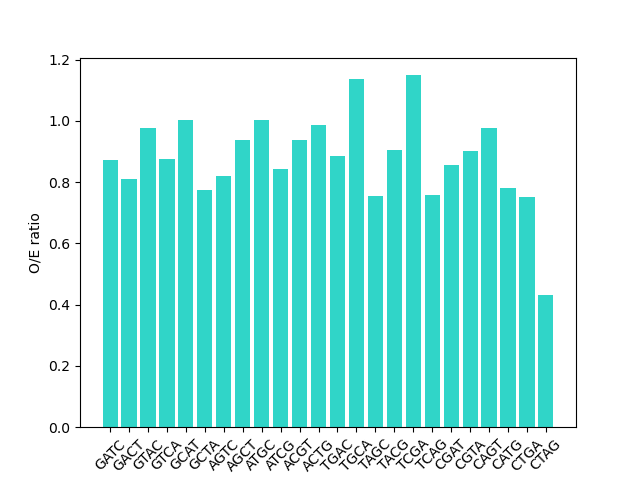
5. Оценка представленности сайта GATC в геноме археи
Для выполнения задания использовался геном археи Halobacterium salinarum. Поиск всех вариаций сайтов из букв GATC выполнялся программой cbcalc.
cbcalc -s sites.txt -M -o results.tsv GCF_004799605.1_ASM479960v1_genomic.fna
Результаты визуализированы при помощи гистограммы (рис 3). Искомый сайт (GATC) имеет скор O/E в районе 0.9. Таким образом он может быть сайтом метилирования.