Создание выборки из последовательностей
В данном практикуме выполнялся поиск сингала начала транскрипции (последовательность Shine-Dalgarno или SD) в геноме археи Halobacterium salinarum. Последовательность находится на расстоянии около -10 нуклеотидов от старт-кодона. Для поиска сигнала была собрана выборка из 100 промоторных последовательностей [-40:-1] белок-кодирующих генов. В качестве контроля были взяты участки из кодирующий последовательности того же белка [60-100]. Для удобства последовательности выбирались на "+" цепи.
MEME
Для поиска паттерна в собранных последовательностях использовалась программа MEME. Так как по литературным данным длина последовательности Shine-Dalgarno составляет 6 нуклеотидов. Поэтому минимальная длина паттерна была установлена 5, максимальная -- 8.
meme sd_seq.fasta -dna -nmotifs 1 -minw 5 -maxw 8
Некоторое подобие "мотива" было найдено в 2 из 100 последовательностях. Результат работы MEME Таким образом мы можем говорить о том что нашими методами мы либо не можем найти последовательность Shine-Dalgarno, либо, так как исследуемый организм архея, данная последовательность отсутствует.
Затем программа была запущена с параметрами минимальная длина - 22, максимальная - 28. Данный поиск выполнялся как попытка найти сайт связывания рибосомы. Полученный мотив (рис. 1) имеет 2 достаточно обогащенных участка. Далее интерес преставляет участок 17-26. Его длина 9. Запустим снова программу meme, но с параметром длины 9-11. В порядка половине последовательностей был найден мотив (рис 2).

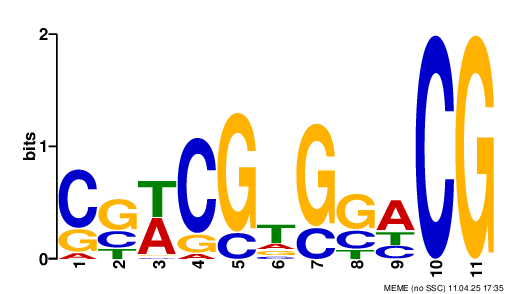
FIMO
Для проверки найденного сигнала была использована программа Fimo.
fimo --oc fimo_prot meme_out/meme.txt prot_seq.fasta
fimo --oc fimo_sd meme_out/meme.txt sd_seq.fasta
Выдача содержала отчет о поиске паттерна в последовательностях. Для промоторной области было найдено 43 значимых (p-value 0.0001) совпадения, для региона из белка 28 значимых находое. Данные результаты говорят о том, что найденный паттерн вероятно не является значимым сигналом в геноме.