Описание параметров BLAST
- Enter Query Sequence - в этом блоке можно ввести последовательность, с которой будет проводиться выравнивания и по которой будут искаться гомологичные последовательности в базе данных. Присутствует возможность ввести её напрямую, в виде файла, указать определенный участок последовательности, с которым будет работать алгоритм (Query subrange)
- Database - этот параметр задаёт базу данных, среди последовательностей которой будет проводиться поиск
- Organism - этот параметр задаёт организм или организмы, среди белков которых будет проводиться поиск. Также есть возможноть исключть из поиска белки каких-то организмов
- Exclude - позволяет исключить некоторые белки из поиска
- Algorithm - этот параметр задаёт алгоритм, по которому будет проводиться поиск
- Max target sequences - этот параметр задает максимальное количество последовательностей, которые будут выданы в результате поиска
- Short queries - данный параметр позволит программе самостоятельно изменять остальные параметры для улучшения поиска в случае коротких последовательностей
- Expect threshold - этот параметр задает граничное значение e-value, последовательности со значением выше данного не будут выдаваться в поиске
- Word size - этот параметр определяет размер слова, на которые будет разбита исходная последовательность и соответствия с которым будут искаться в базе данных (представляющей собой "алфавитный указатель" таких слов в других последовательностях) для уменьшения количества последовательностей, с которыми будут проводитья выравнивания (для ускорения работы алгоритма)
- Max matches in a query range - этот параметр задаёт ограничение числа находок в одной последовательности из банка
- Matrix - задаёт матрицу, значения которой будут использоваться для построения выравнивания
- Gap Costs - этот параметр определяет штрафы за гэпы
- Compositional adjustments - этот параметр позволяет ввести более сло
- Filters and Masking - позволяет задать дополнительные фильтры и маски
Поиск гомологов белка AHPC_AMPXN
При помощи BLAST был проведен поиск последовательностей, предположительно гомологичных белку AHPC_AMPXN в базе данных swissprot. Были заданы следующие параметры поиска:
Database: UniprotKB/Swiss-Prot(swissprot)
Max target sequences: 20000
Остальные параметры были заданы по умолчанию. Результаты поиска представлены в таблице
Среди найденых белков были выбраны 6 с различными названиями и e-value, но с высоким процентом покрытия (>85) и 1 предположительно негомологичный белок со значением e-value 1.8 и покрытием 85%
При помощи программы Jalview было построено множественное выравнивание этих последовательностей.

Затем я удалил сильно отличающуюся последовательность (ту самую, которая предположительно была негомологична) и заново выровнял последовательности.

Выделенный отрезок выравнивания из 11 позиций начинается и заканчивается абсолютно консервативными позициями, не содержит гэпов и имеет высокую плотность консервативных позиций (7 абсолютно
консервативных позиций, из чего можно сделвть вывод, что данные последовательности гомологичны.
Карта сходства двух белков
Из предложенных белков было выбрано 2 белка из разных групп (L8H299_ACACA из организма Acanthamoeba castellanii str. Neff и A0A2H3JYG9_WOLCO из Wolfiporia cocos (strain MD-104) . Для них
при помощи BLAST была построена карта сходства
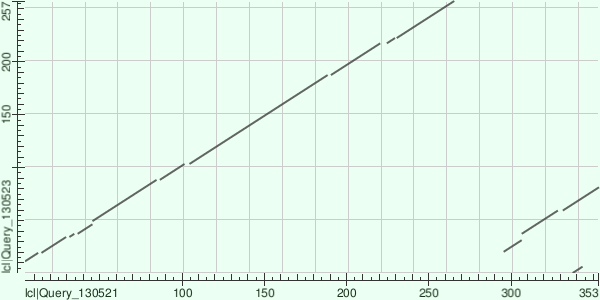
Как видно из графика, у последовательности, расположенной вертикально произошла делеция конца последовательности
У последовательности, расположенной горизонтально отрезок из начала дуплицировался и вставился в конец
Игры с BLAST
Для первого эксперимента я взял последовательность, которая не кодирует белок: "Diet mountain dew baby New York City
Never was there ever a girl so pretty". Если запусть BLAST с параметрами по умолчанию (кроме базы данных, которую по условия задания нужно брать swissprot и "Automatically adjust parameters for short input sequences",
где галочки быть не должно), не находится ни одной последовательности. Поэтому параметры поиска были изменены. "Expect threshold" был установлен 100, а "wordsize" - 2. Тогда BLAST
смог найти 49 результатов. Интересно, что у лучшего из них e-value 0.75, покрытие 78% и identity 33.33%. Для него было построено выравнивание с исходной последовательностью.

Далее было проведено неколько поисков с последовательностью белка AHPC_AMPXN и различными параметрами BLAST.
wordsize
Для начала все параметры были взяты по умолчанию и проведены запросы с различной длиной слов. С уменьшением длины слова увеличивается количество выдаваемых последователностей - 139 для wordsize 6, 185 для wordsize 3, 189 для wordsize 2.
Expected threshold
Тут всё очень предсказуемо и неинтересно - чем больше значение, тем больше результатов в выдаче
Matrix
BLOSUM90: min(e-value) = 9e-42, количество запросов = 100
BLOSUM80: min(e-value) = 5e-42, количество запросов = 100
BLOSUM62: min(e-value) = 1e-44, количество запросов = 102
BLOSUM50: min(e-value) = 5e-46, количество запросов = 119
BLOSUM45: min(e-value) = 4e-49, количество запросов = 117
Можно сделать вывод, что при уменьшении количества посделовательностей, по которой составлялась матрица BLOSUM, миимальное e-value уменьшается, а количество выдаваемых последовательной
несколько растёт