В данном практикуме мне досталась хромосома №10
Ниже представлена таблица со всеми использованными командами:
| Команда | Описание |
| hisat2-build chr10.fasta chr10_index | Индексирование референсной последовательности |
| fastqc chr10.fastq | Анализ качества чтений программой FastQC |
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr10.fastq chr10_trimmed.fastq TRAILING:20 MINLEN:50 | Очистка при помощи Trimmomatic. Были обрезаны с конца нуклеотиды с качеством ниже 20, а также убраны все последовательности длиной менее 50 нк |
| hisat2 -x chr10_index -U chr10_trimmed.fastq -S chr10_align.sam --no-spliced-alignment --no-softclip | Картирование чтений на геном (--no-softclip – выравнивание должно быть по всему риду, --no-spliced-alignment – картирование должно быть без разрывов) |
| samtools view -b chr10_align.sam -o chr10_align.bam | Перевод в .bam формат |
| sort chr10_align.bam chr10_sorted | Сортировка выравниваний по координате в референсе |
| samtools index chr10_sorted.bam | Индексирование |
| samtools flagstat chr10_sorted.bam | Получение информации о количестве чтений, картированых на геном |
| samtools mpileup -u -f chr10.fasta -o chr10_polymorf.bcf chr10_sorted.bam | Создание файла с полиморфизмами |
| bcftools call -cv chr10_polymorf.bcf -o chr10_polymorf.vcf | Перевод файла в формате bcf в формат vcf |
| convert2annovar.pl -format vcf4 chr10_polymorf.vcf > chr10_polymorf.avinput | Перевод файла в формате vcf в формат, используемый программой annovar |
| annotate_variation.pl -out chr10_refgene -build hg19 chr10_polymorf.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация файла c snp по refGene |
| annotate_variation.pl -filter -out chr10_dbsnp -build hg19 -dbtype snp138 chr10_polymorf.avinput /nfs/srv/databases/annovar/humandb.old/ | Aннотации файла с snp по базе данных dbsnp |
| annotate_variation.pl -filter -out chr10_1000genomes -build hg19 -dbtype 1000g2014oct_all chr10_polymorf.avinput /nfs/srv/databases/annovar/humandb.old/ | Aннотации файла с snp по базе данных 1000 genomes |
| annotate_variation.pl -regionanno -out chr10_gwass -build hg19 -dbtype gwasCatalog chr10_polymorf.avinput /nfs/srv/databases/annovar/humandb.old/ | Aннотации файла с snp по базе данных GWAS |
| annotate_variation.pl -filter -out chr10_clinvar -dbtype clinvar_20150629 -buildver hg19 chr10_polymorf.avinput /nfs/srv/databases/annovar/humandb.old/ | Aннотации файла с snp по базе данных Clinvar |
Триммирование
Исходно было 10666 чтений. После триммирования осталось 10526 (98.69%)Качество изначальных чтений бовольно хорошее (больше 20). Поэтому после обрезки качество сильно не изменилось. Было откинуто меньше 1,5% последовательностей. Поэтому, на мой взгляд, можно было обойтись и без триммировани.Качество до:
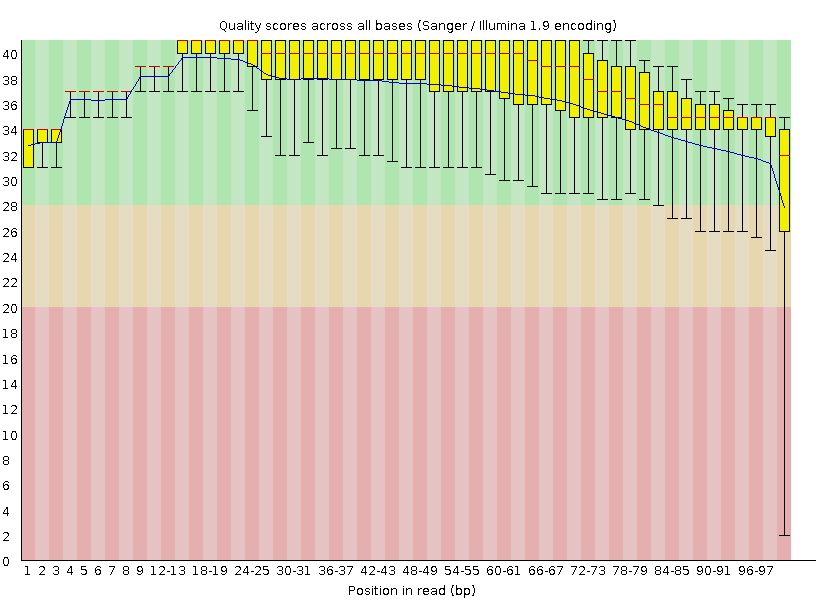
Качество после:
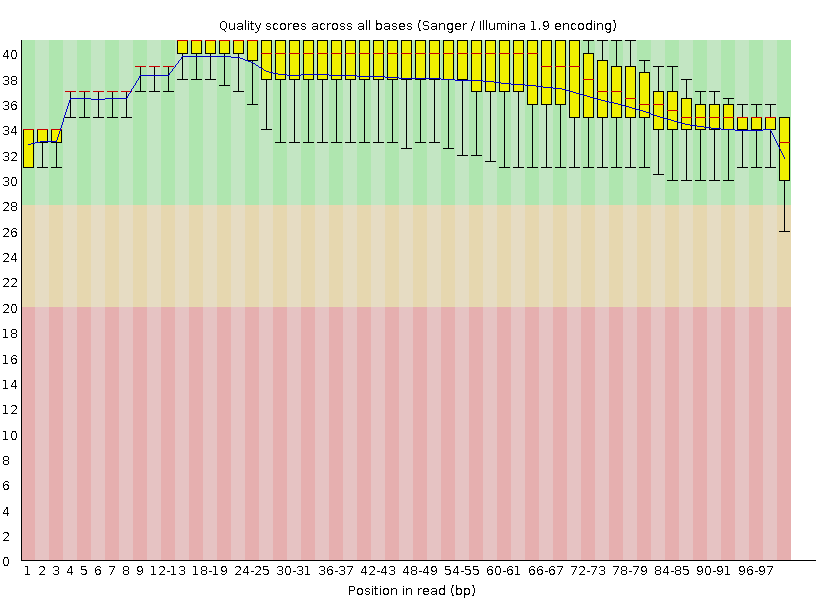
Картирование
Было откартировано 98.78% чтений. Результат работы программы hisat2:
10526 reads; of these:
10526 (100.00%) were unpaired; of these:
128 (1.22%) aligned 0 times
10398 (98.78%) aligned exactly 1 time
0 (0.00%) aligned >1 times
98.78% overall alignment rate
Можно сделать вывод, что это хорошее качество картирования
Описание 3 полиморфизмов
| Позиция в хромосоме | Тип полиморфизма | Референс | Чтения | Глубина покрытия | Качество чтения |
|---|---|---|---|---|---|
| 5804633 | Вставка | TTC | TTCTC | 119 | 217.468 |
| 5804865 | Замена | С | Т | 51 | 225.009 |
| 5805087 | Делеция | AACA | АА | 37 | 217.468 |
Анализ SNP
У меня получилось 57 SNPs (35 transitions and 22 transversions), и 9 инделей
| Качество | Глубина | |
|---|---|---|
| Медиана | 69.01 | 8 |
| Среднее | 97.47 | 21.62 |
Аннотация SNP
RefSeq
SNP были разделены на intronic (52), exonic (10) и UTR3 (4). SNP оказались в генах FAM208B, RTKN2 и CASP7. Мутаций, каоторые привели к изменению аминокислоты (nonsynonymous) оказалось 7, которые не привели (synonymous) - 3.
dbsnp
57 из 66 имеют rs
1000genomes
Средняя частота по выдаче 1000genomes - 61,24%
gwasgwasCatalog Name=Osteosarcoma chr10 5804531 5804531 G A het 225.009 70 gwasCatalog Name=Rheumatoid arthritis chr10 63958112 63958112 T C het 225.009 75 gwasCatalog Name=Vitiligo chr10 115481018 115481018 C T het 225.009 91
Как оказалось, найденные полиморфизмы связаны с остеосаркомой, ревматоидным артритом и витилиго
clinvar
Не было найдено ни одного snp в этой базе