Задание 1
Целью данного практикума была расшифровка результатов секвенирования.
- Исходные файлы (20_F.ab1 и 20_R.ab1), содержащие прямую и обратные цепи, были открыты в Chromas.
- Для обратной цепи был совершен переход к комплиментарной цепи (Reverse) и затем последовательности были вручную выровнены.
- Были определены нечитаемые концы. (В скобках указаны значения, автоматически определенные программой)
Прямая Обратная 3' 19 (50) 21 (48) 5' 52 (9) 43 (31) - Для каждого проблемного нуклеотида были было определено значение по комплиментарной цепи (что везде оказалось просто сделать).
- Было произведено выравнивание двух последовательностей программой needle alignment.needle, alignment.fasta
- На основе файла с выравниванием была получена консенсусная последовательность
Общий вид хроматограммы (за исключением концевых участков) очень хороший. Уровень сигнала в 4-7 раз выше уровня шума. Интересно, что почему-то иногда сигналы C
для прямой и G для обратной цепочек сильно превосходят уровень других сигналов. 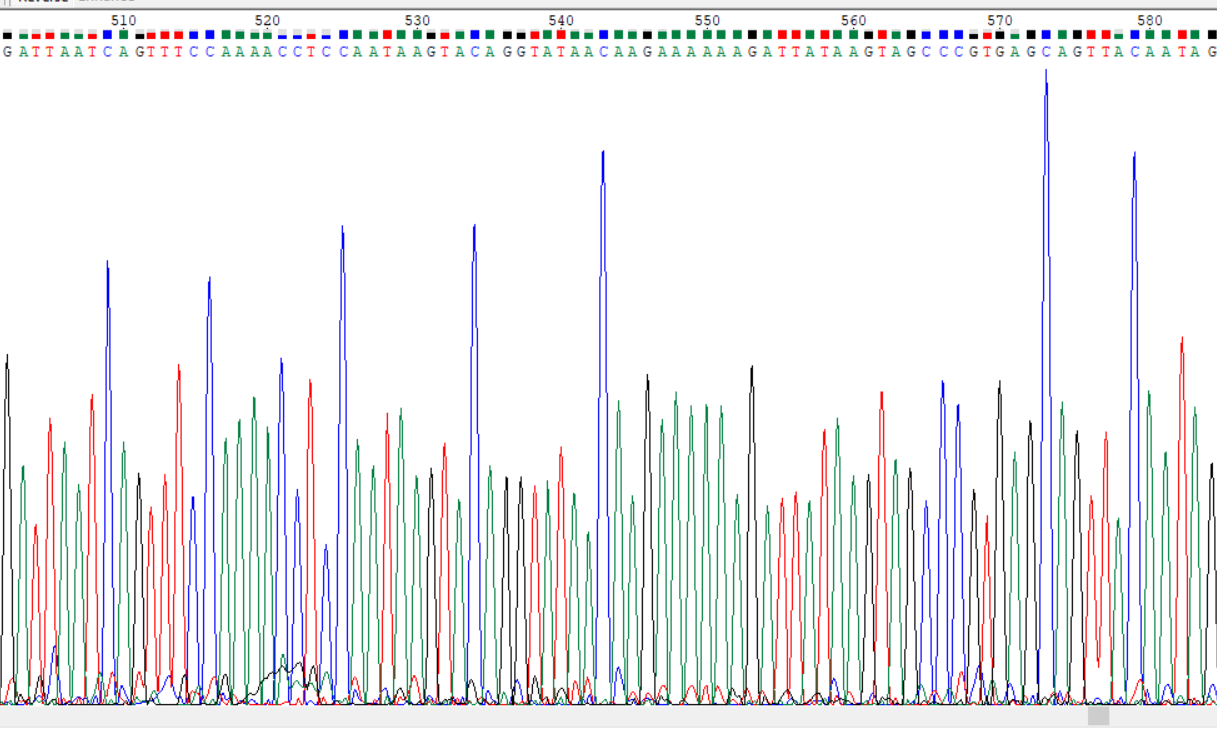
- Проблемные нуклеотиды
- Пример 1
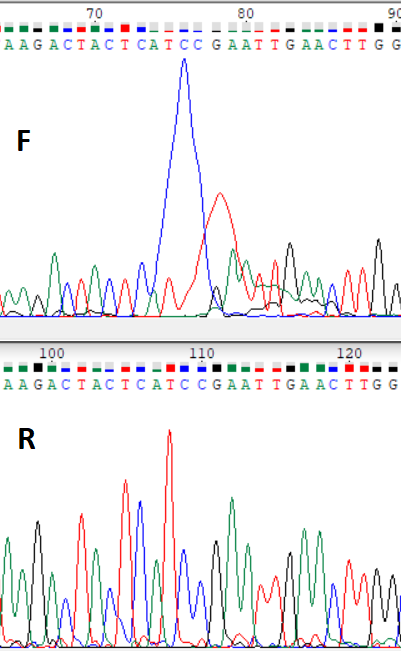 На верхней картинке "синяя" и "краснаая" краски разлились, последовательность легко восстанавливается по комплиментарной цепи
На верхней картинке "синяя" и "краснаая" краски разлились, последовательность легко восстанавливается по комплиментарной цепи - Пример 2
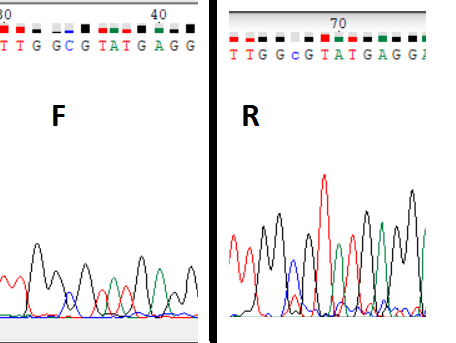 На обратной цепочке не распознался нуклеотид, так как пик не очень сильно превосходит уровень шума. Восстанавливается даже без комплиментарной цепочки.
На обратной цепочке не распознался нуклеотид, так как пик не очень сильно превосходит уровень шума. Восстанавливается даже без комплиментарной цепочки. - Пример 3
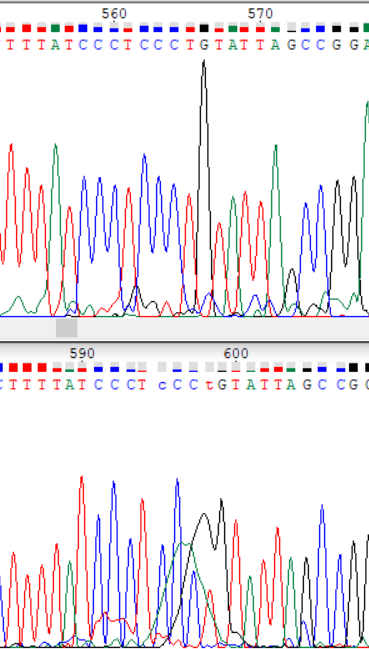 На обратной цепочке разлились "зеленая" и "черная" краски, легко восстанавливается по прямой цепочке
На обратной цепочке разлились "зеленая" и "черная" краски, легко восстанавливается по прямой цепочке - Пример 4
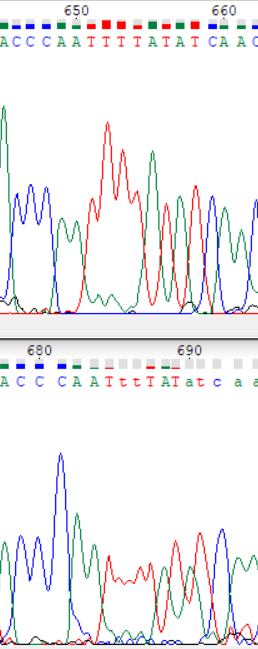 На обратной цепочке слились несколько пиков, легко восстанавливается по прямой цепочке
На обратной цепочке слились несколько пиков, легко восстанавливается по прямой цепочке
Задание 2
Так как предыдущая хроматограмма была высокого качества, я взял нечитаемую хроматограмму из папки bad.
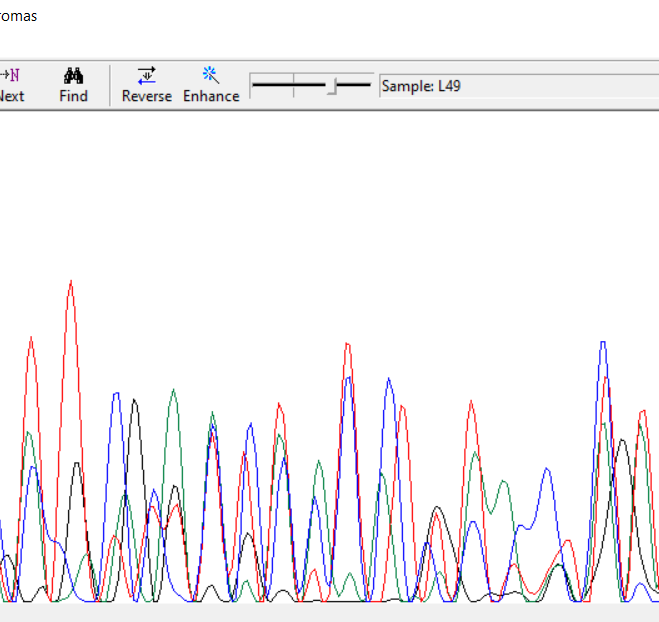 Хроматограмму прочитать невозможно, так как почти везде в одном месте расположено несколько различных близких силе сигнала пиков. Вероятно в препарате
находилось несколько различных ДНК-матриц.
Хроматограмму прочитать невозможно, так как почти везде в одном месте расположено несколько различных близких силе сигнала пиков. Вероятно в препарате
находилось несколько различных ДНК-матриц.