Валидация Задание 1.
В данном задании я изучал структру трипсина (PDB id 3unr). Это один из самых известных пищеварительных ферментов, относящийся к сериновым гидролазам. Целью статьи, в которой приводится данная структура, была попытка предсказать протонированность остатков аспартата, глутамата и гистедина при помощи анализа длины связей. Так, например, связи O=C и HO-C имеют разную длину, и если способ получения структурных данных позволяет их различать, то мы можем предсказывать протонированность. В статье оцениалось влияние разрешение на значимость подобных предсказаний, помимо трипсина рассматривалось ещё 3 других белка (лизоцим, субтилизин и cMyBp-C). Оценка качества структуры Оценка качества проводилась по отчету, представленому на сайте pdb. Разрешение достаточно хорошее (1.2 А), полнота 97%.
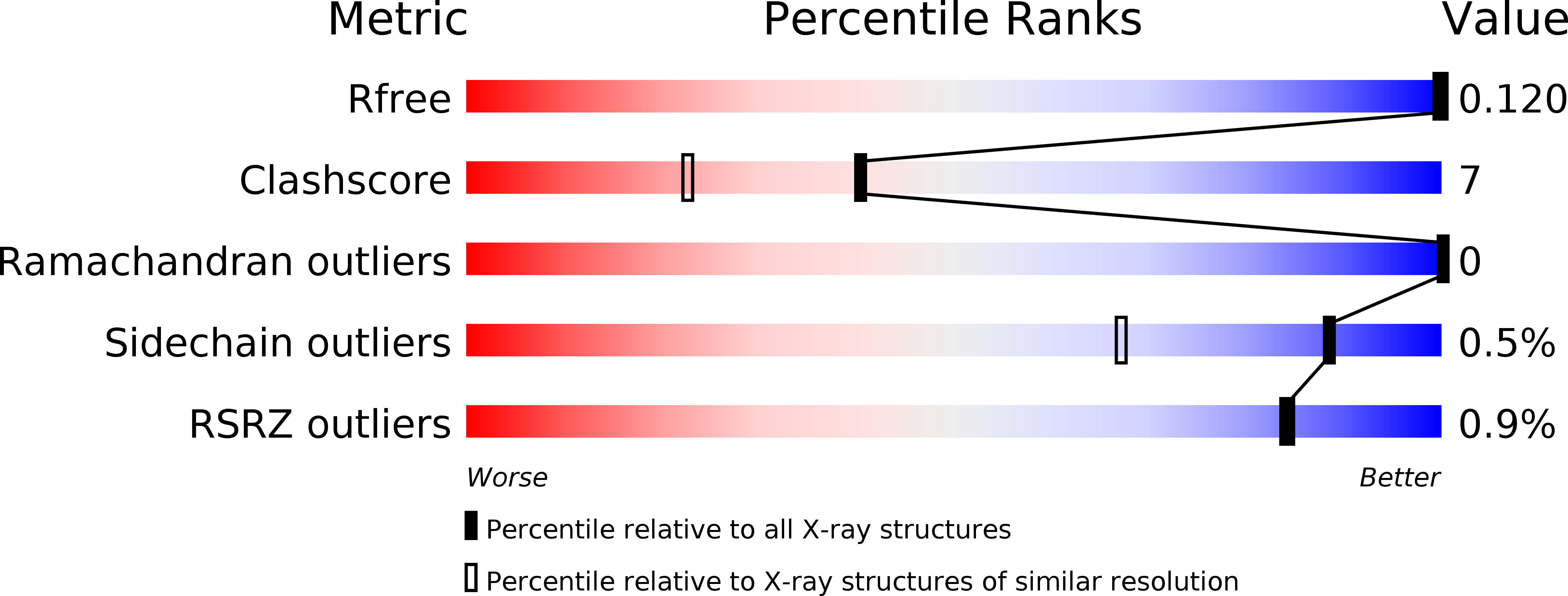
R-фактор имеет низкие значения (0.105), свободный R-фактор составил (0.121), он также низок и близок к R-фактору, что говоит об отсутствии переобучения и хорошем соответствии
модели экспериментальным данным.
Сlashscore составил 7 на 1000 атомов, что так же мне кажется очень хорошим значением.
Маргиналов по картам Рамачадрана вообще обнаружено не было, что также свидительствует о высоком качестве модели.
Доля маргинальных остатков в боковых цепях так же была крайне мала - 1% (всего 2 остатка из 226)
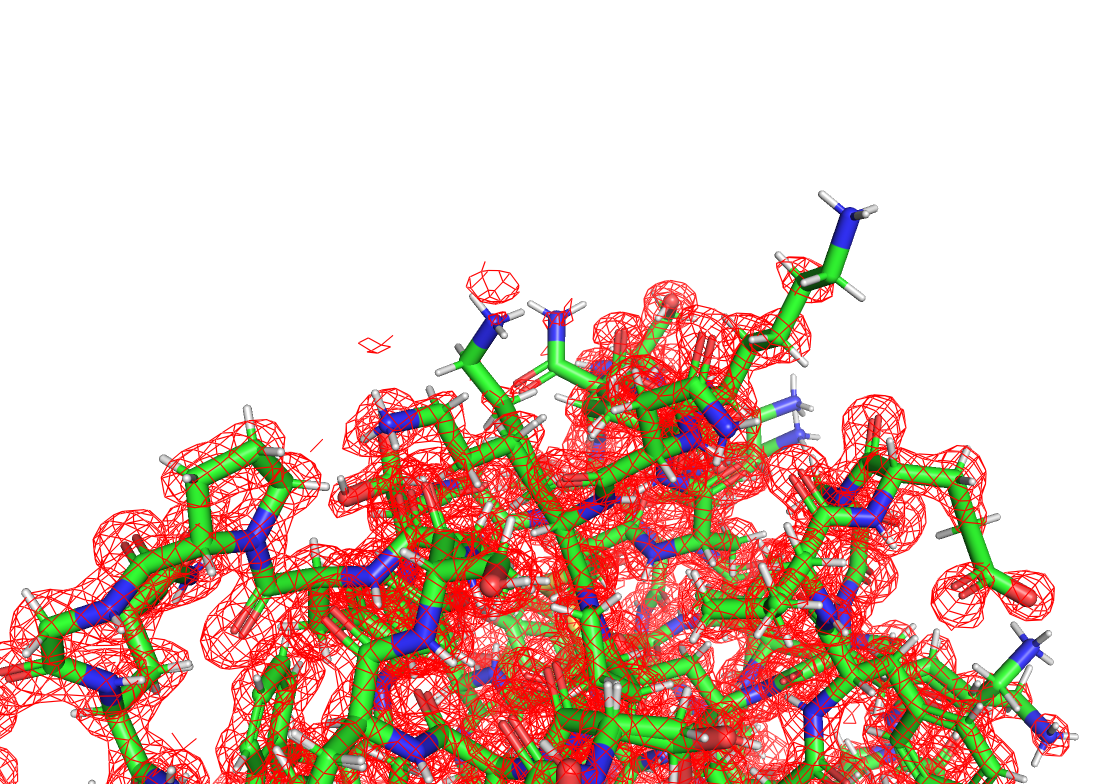
Почти все остатки покрыты электронной плотностью, за исключением некоторых торчащих наружу остатков лизина, что может объясняться их высокой подвижностью.
Задание 2.
В данном задании более подробно изучались некоторые маргинальные остатки, помимо отчета PDB использовался сервис MolProbity.
Его выдача не сильно отличалась от отчета PDB.
Длины и углы связей Маргиналов по длинам и углам связей обнаружено не было.
Clashes Все дефекты были связаны или с восстановленными водородами, или возникало перекрытие ван-дер-ваальсовых радиусов с кислородами воды, при этом для всех перекрытий с
кислородами воды существовал другой альтлок остатка, в котором перекрытия не было. На данном изображении представлен маргинальный альтлок лизина 224, как видно из изображения,
этот альтлок намного хуже подтверждается электронной плотностью, в целом данный остаток расположен на поверхности белка, поэтому может быть подвижен, что и может приводить к распознаванию
маргинальной конформации. 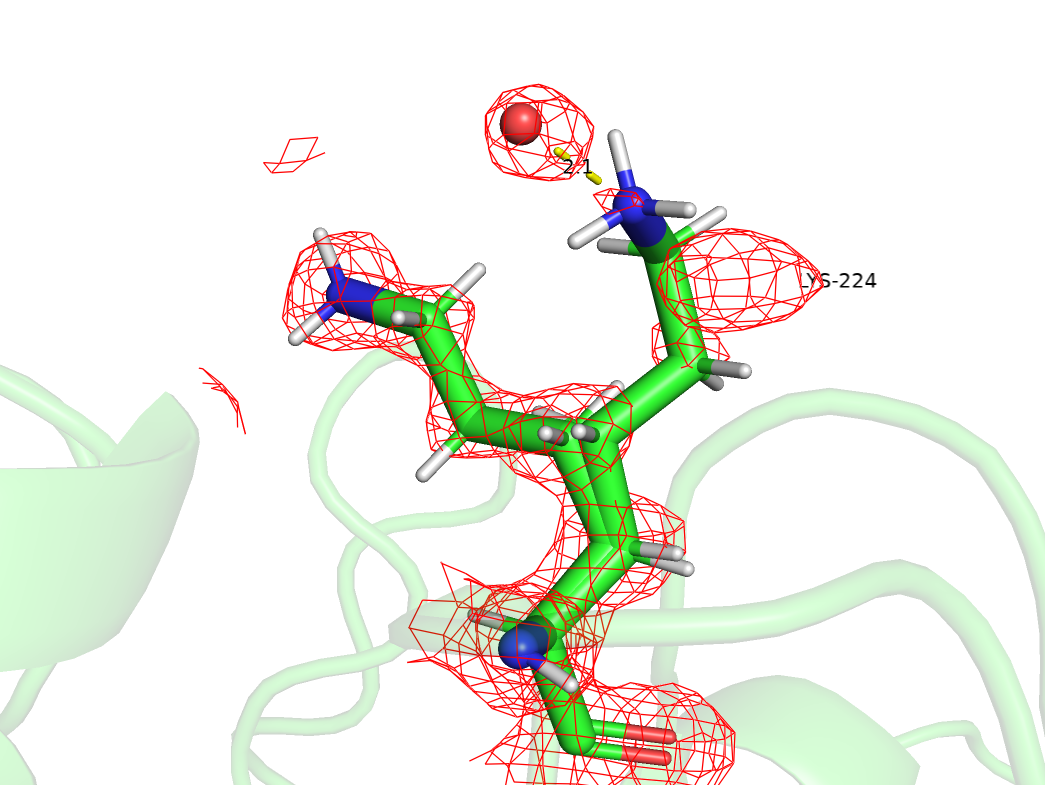 Также происходит перекрытие радиусов атомов между водородом одного из альтлоков валина 53 и двумя атомами (углеродом и водородом) лейцина 105, согласно отчету PDB перекрытие двух
водородов составило 0.74 А, а перекрытие водорода и углерода 0.69 А. По электронной плотности также видно, что она описывает "не конфликтующий" альтлок, тогда как "конфликтующий" выходит
за её границы.
Также происходит перекрытие радиусов атомов между водородом одного из альтлоков валина 53 и двумя атомами (углеродом и водородом) лейцина 105, согласно отчету PDB перекрытие двух
водородов составило 0.74 А, а перекрытие водорода и углерода 0.69 А. По электронной плотности также видно, что она описывает "не конфликтующий" альтлок, тогда как "конфликтующий" выходит
за её границы.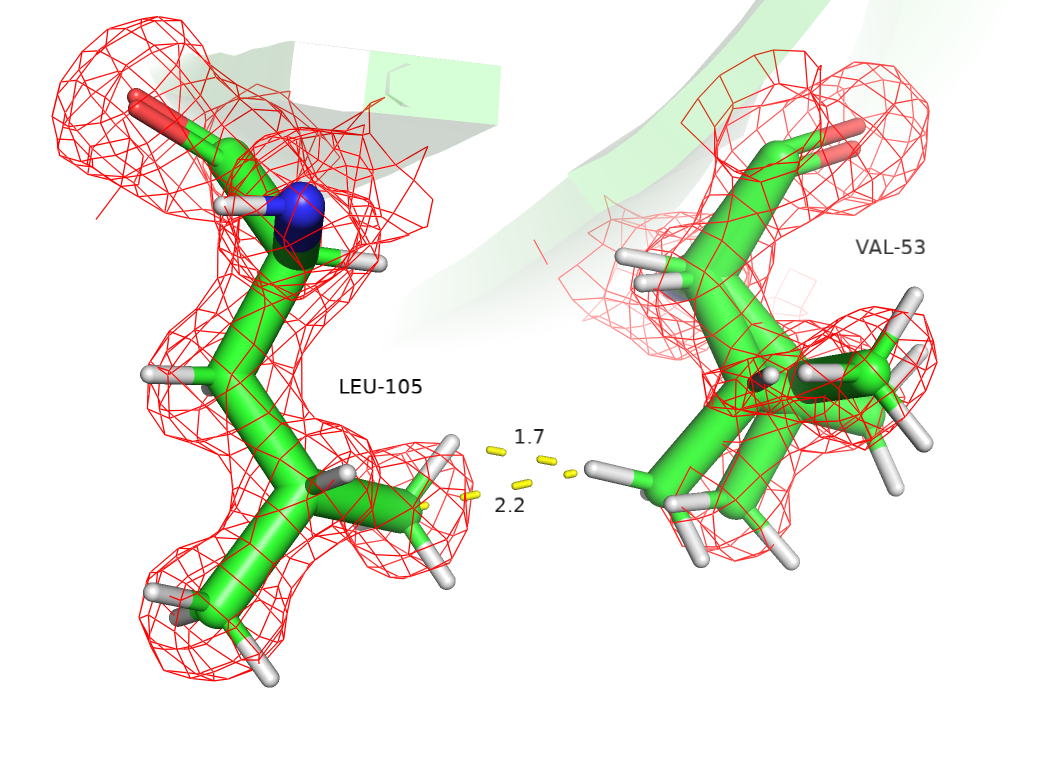 Торсионные углы. Для торсионных углов остова не было найдено ни одного маргинала, все остатки попадают в благоприятные области
карты Рамачадрана. Для боковых цепей в соответствии с отчетом pdb был найден 1 остаток (asn 115) с двумя маргинальными альтлоками, он также расположен на поверхности белка, из-за чего может быть подвижным, что вносит шум
в данные и приводит к появлению маргинальности. Для альтлока А торсионные углы составили 48.9 и -106.6, для альтлока В -163.2 и 61.4.
Торсионные углы. Для торсионных углов остова не было найдено ни одного маргинала, все остатки попадают в благоприятные области
карты Рамачадрана. Для боковых цепей в соответствии с отчетом pdb был найден 1 остаток (asn 115) с двумя маргинальными альтлоками, он также расположен на поверхности белка, из-за чего может быть подвижным, что вносит шум
в данные и приводит к появлению маргинальности. Для альтлока А торсионные углы составили 48.9 и -106.6, для альтлока В -163.2 и 61.4. 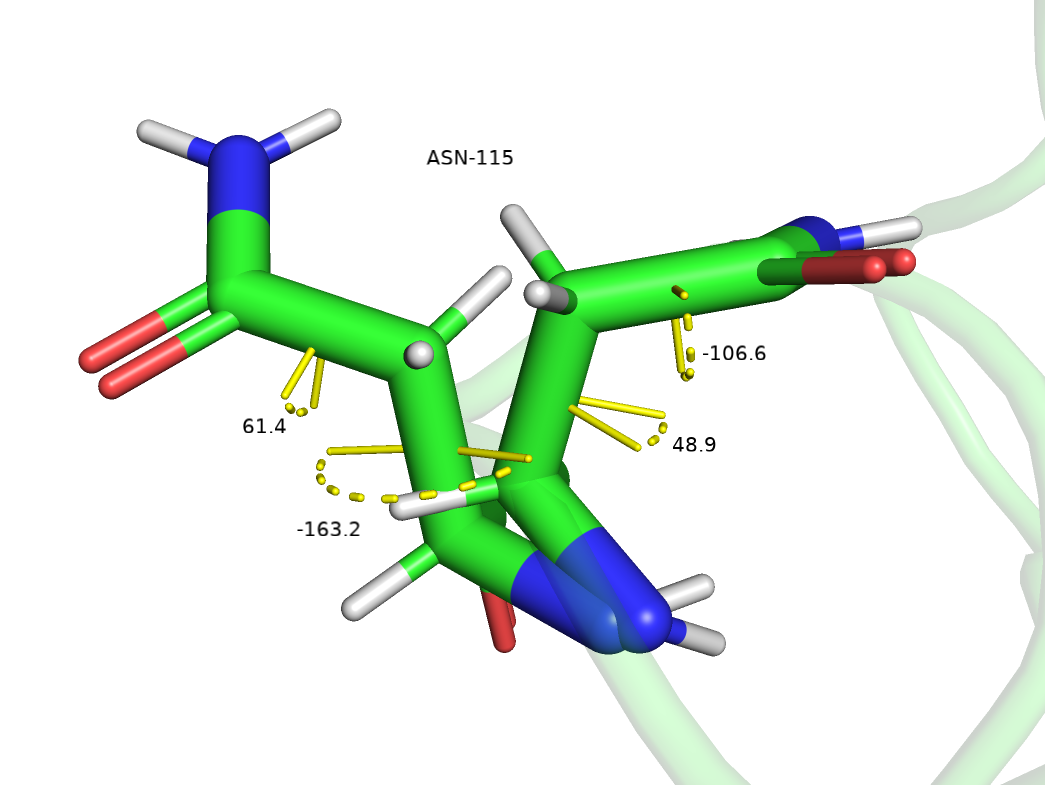 Лиганды
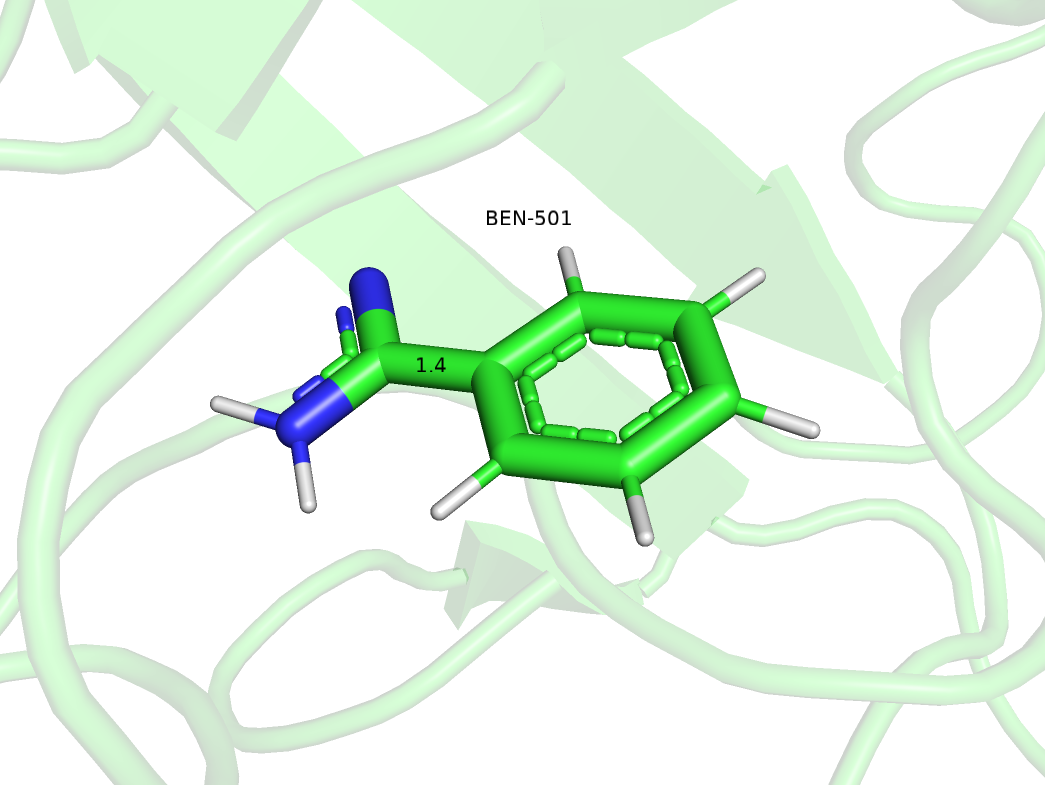
Для лигандов было больше маргинальных элементов. В бензамидине была обнаружена связь между углеродами нехарактерной длины: Observed - 1.43 А, Ideal 1.47 А.
Лиганды
Для лигандов было больше маргинальных элементов. В бензамидине была обнаружена связь между углеродами нехарактерной длины: Observed - 1.43 А, Ideal 1.47 А.
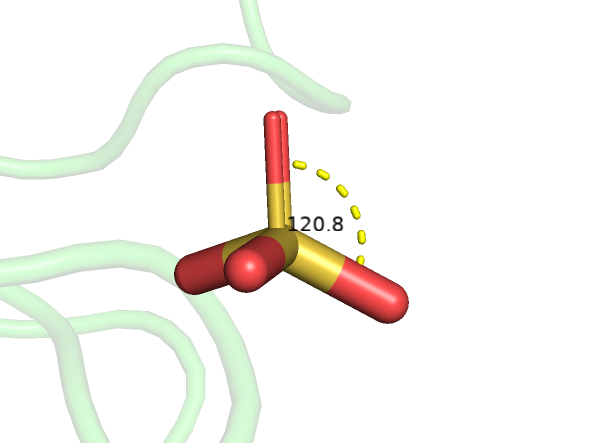
 Также были молекулы с нехарактерными углами между связями. Например, SO4, Углы: Observed 120.83, ideal 109.06.
Также были молекулы с нехарактерными углами между связями. Например, SO4, Углы: Observed 120.83, ideal 109.06.
Задание 3.
На мой взгляд данная структура отлично подходит для изучения особенностей исследуемого белка, она содержит крайне маленькое количество маргинальных остатков, и для подавляющего большинства из них описана альтернативная конформация, в которой данный остаток не является маргинальным. Также иногда маргинальность связана с тем, что остаток может быть подвижным в следствие расположения на поверхности и маленького количества стабилизирующих связей, все эти факторы не должны серьезно влиять на функционирование данного белка, структура также содержит электронные плотности, описывающие взаимодействующие с белком лиганды.
Задание 4.
Качество модели изменилось не сильно - оно и так было очень хорошим, и больших возможностей для улучшения не было. В результате несколько улучшилось
соответствие картам рамачадрана, однако несколько ухудшилось качество ротамеров. Также было удалено 36 молекул воды. Внесенные изменения в целом слабо затронули основной белок, в
основном изменения касались лигандов. Рассмотрим остаток, в который PDB REDO внес наибольшие изменения, теоретически улучшив структуру, это аргинин 117, расположенный на поверхности
белка. (На картинке зеленым показаны углероды структуры из PDB, фиолетовым - из "улучшенной" структуры).
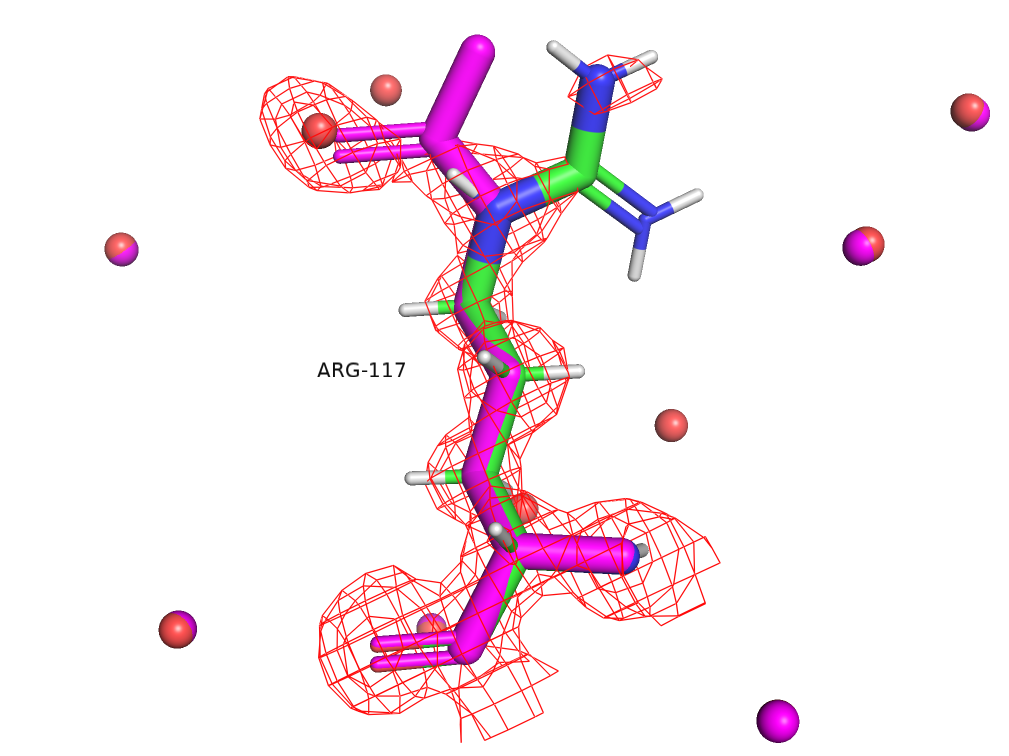 Как мы видим, программа "развернула" аргинин, убрав молекулу воды и вписав азот в её электронную плотность. На мой взгляд, это делает модель только
менее точной. Рассмотрим что изменилось для остатков, рассмотренных в задании 2. Для Лизина 224 не было внесено никаких изменений в положение самого лизина, однако было
изменено положение воды, однако почему-то в худшую сторону - она стала только ближе к азоту: расстояние стало 1.6 вместо бывших 1.9. (На картинке оранжевым изображено "новое" положение воды,
красным - "старое").
Как мы видим, программа "развернула" аргинин, убрав молекулу воды и вписав азот в её электронную плотность. На мой взгляд, это делает модель только
менее точной. Рассмотрим что изменилось для остатков, рассмотренных в задании 2. Для Лизина 224 не было внесено никаких изменений в положение самого лизина, однако было
изменено положение воды, однако почему-то в худшую сторону - она стала только ближе к азоту: расстояние стало 1.6 вместо бывших 1.9. (На картинке оранжевым изображено "новое" положение воды,
красным - "старое"). 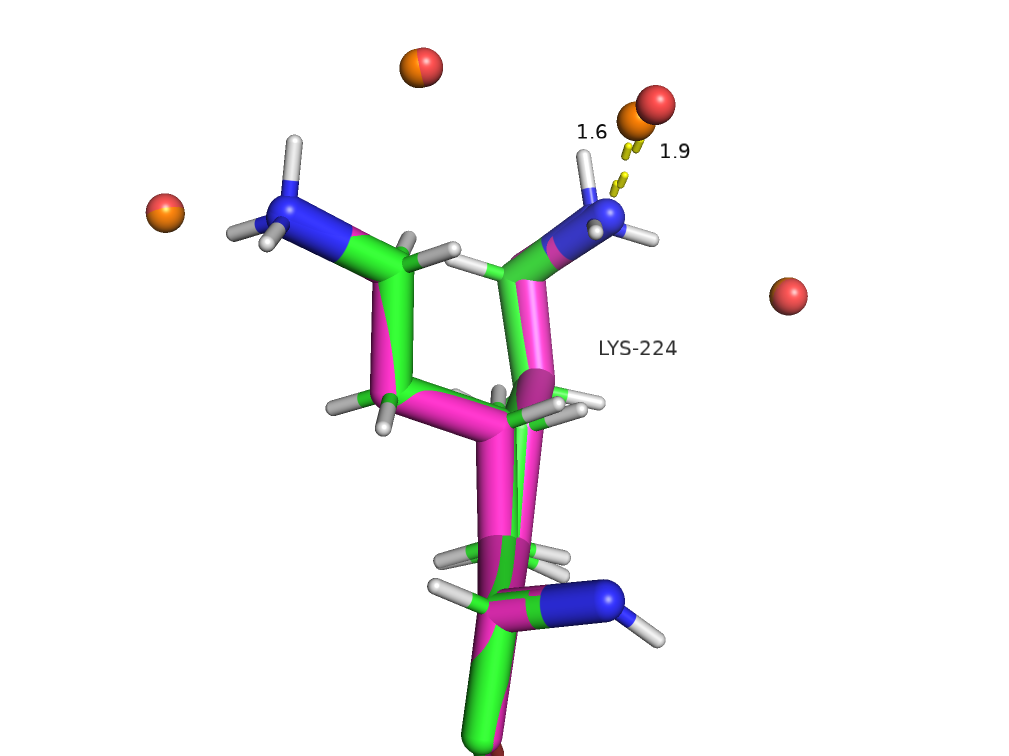 В положение лейцина-105 и валина-53, а также аспаргина-115 не было внесено никаких изменений. В бензамидине были подправлены
длины связей.
В положение лейцина-105 и валина-53, а также аспаргина-115 не было внесено никаких изменений. В бензамидине были подправлены
длины связей. 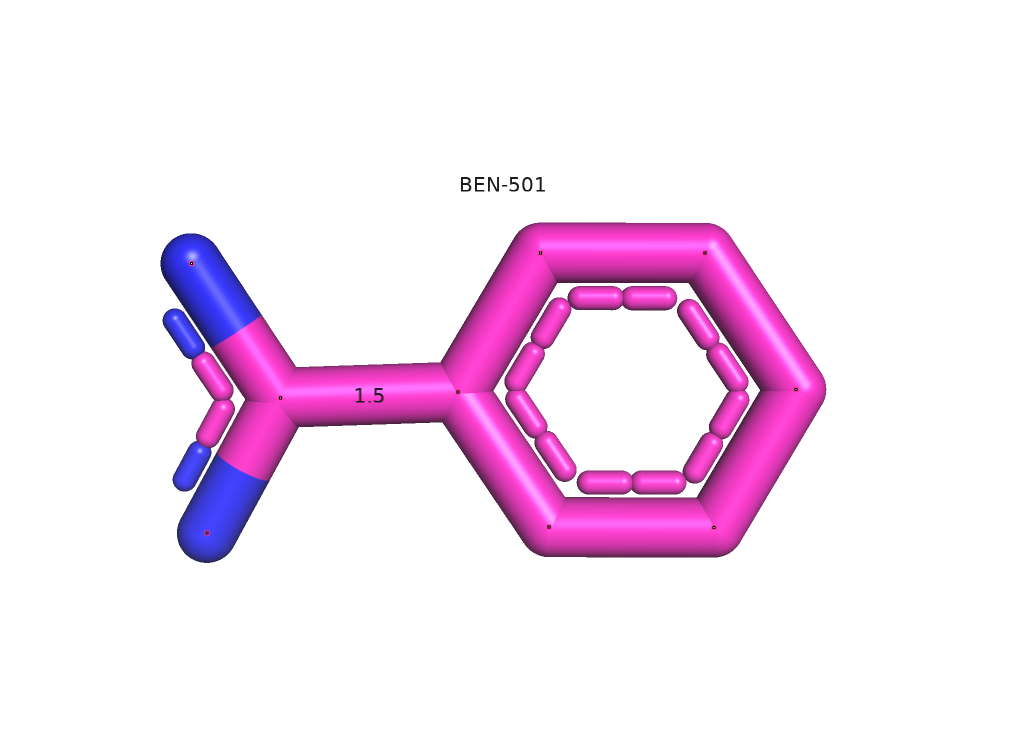 Для остатка SO4 угол между связями только ухудшился.
Для остатка SO4 угол между связями только ухудшился.