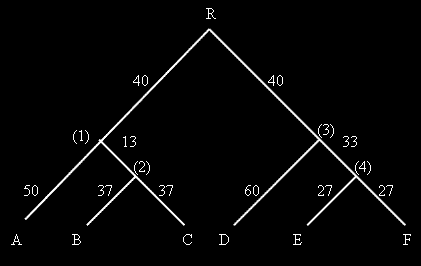
где (1) - CLCA_TIPA1 (2) - CLCA_SLON2 (3) - CLCA_ESLI3 (4) - CLCA_KOBR4 A - CLAA_ABLOM мутантные B - CLBB_MOLBA последовательности. C - CLCC_FORS4 D - CLDD_HHOKK E - CLEE_VAX57 F - CLFF_KONEC R - CLCA_ECOLI исходная последовательность.
Была использована полученная в одной из прошлых работ эволюционная модель, представленная ниже в виде дерева:

|
где (1) - CLCA_TIPA1 (2) - CLCA_SLON2 (3) - CLCA_ESLI3 (4) - CLCA_KOBR4 A - CLAA_ABLOM мутантные B - CLBB_MOLBA последовательности. C - CLCC_FORS4 D - CLDD_HHOKK E - CLEE_VAX57 F - CLFF_KONEC R - CLCA_ECOLI исходная последовательность. |
C помощью программы ednadist были определены эволюционные расстояния по Джуксу - Кантору между данными мутантными последовательностями эволюционной модели. Эволюционные расстояния представлены в виде таблицы:
| CLAA_ABLOM | CLBB_MOLBA | CLCC_FORS4 | CLDD_HHOKK | CLEE_VAX57 | CLFF_KONEC | |
| CLAA_ABLOM | 0,00 | 0,70 | 0,74 | 1,31 | 1,40 | 1,40 |
| CLBB_MOLBA | 0,70 | 0,00 | 0,53 | 1,48 | 1,48 | 1,45 |
| CLCC_FORS4 | 0,74 | 0,53 | 0,00 | 1,46 | 1,35 | 1,29 |
| CLDD_HHOKK | 1,31 | 1,48 | 1,46 | 0,00 | 0,88 | 0,84 |
| CLEE_VAX57 | 1,40 | 1,48 | 1,35 | 0,88 | 0,00 | 0,39 |
| CLFF_KONEC | 1,40 | 1,45 | 1,29 | 0,84 | 0,39 | 0,00 |
Далее, четыремя разными методами, были построены филогенетические деревья:
+---------------------CLAA_ABLOM
+-------------------3
! ! +---------------CLBB_MOLBA
! +-----2
--5 +---------------CLCC_FORS4
!
! +-------------------------CLDD_HHOKK
+---------------4
! +-----------CLEE_VAX57
+-------------1
+-----------CLFF_KONEC
|
|
+--------CLBB_MOLBA
+---3
! +-------CLCC_FORS4
!
! +-------------CLDD_HHOKK
--4-----------------2
! ! +------CLEE_VAX57
! +------1
! +-----CLFF_KONEC
!
+---------CLAA_ABLOM
|
|
+-----CLFF_KONEC
+-------4
+-------------------------3 +-------CLEE_VAX57
! !
! +----------------CLDD_HHOKK
!
! +--------CLBB_MOLBA
--2-----1
! +---------CLCC_FORS4
!
+----------CLAA_ABLOM
|
|
+--CLFF_KONEC
+--5
+-----4 +--CLEE_VAX57
! !
+--3 +-----CLDD_HHOKK
! !
! ! +--CLCC_FORS4
--1 +--------2
! +--CLBB_MOLBA
!
+--------------CLAA_ABLOM
|
|
После произведено сравнение полученных деревьев.
|
Исходное дерево модели | UPGMA | NJ | ML | Parsimony | ||
| 100111 | + | + | + | + | + | ||
| 000111 | + | + | + | + | + | ||
| 000011 | + | + | + | + | + |
Как бы это ни было удивительно, но все четыре дерева, полученные четыремя разными способами, имеют одинаковое разбиение по ветвям!
© Трушкин Никита,2005