BLOSUM
Матрица весов BLOSUM62 была создана по данным базы BLOCKS, содержащей эталонные выравнивания, для определения веса выравнивания цитоплазматических белков. Число 62 означает процент идентичности сравниваемых последовательностей – порог кластеризации, после которого последовательности считаются одинаковыми.
Матрица весов PHAT была разработана для мембранных белков с учетом их специфики.
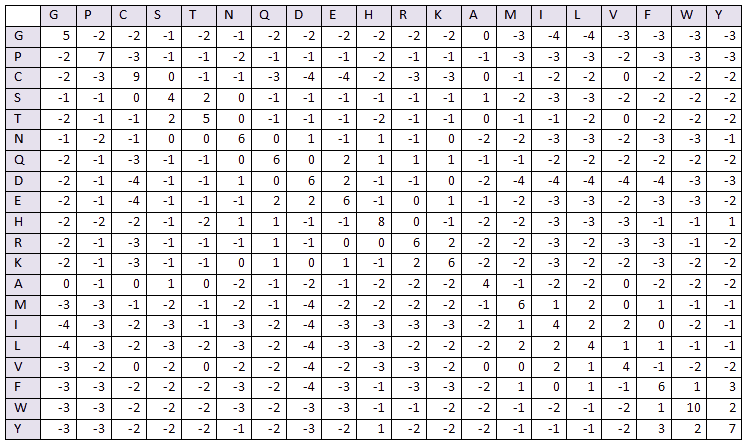
Я построила свою матрицу BLOSUM по данным базы BLOCKS на сегодняшний момент (табл. 1).
Таблица 1. Матрица BLOSUM, построенная вручную по данным базы BLOCKS для цитоплазматических белков
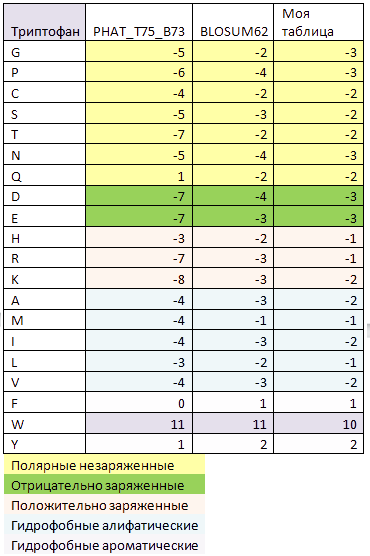
Проведем сравнение моей матрицы с матрицами BLOSUM62 и PHAT_T75_B73. Для этого возьмем отдельную аминокислоту – триптофан. Три значения представлены в таблице 2.
Таблица 2. Сравнение значений матриц весов замен для триптофана. Одинаковым цветом выделены аминокислоты, сходные по свойствам.
Таблица PHAT_T75_B73 построена с кластеризацией 75%, в отличие от матрицы BLOSUM, в которой процент кластеризации 62. Поэтому мы видим, что штрафы в матрице PHAT_T75_B73 гораздо больше. А моя матрица, как видно, более "мягкая". В ней все числа по модулю меньше, чем в других матрицах - меньше и штрафы, и "бонусы". Замена триптофана на самого себя почти не отличается, единственное что в моей матрице за это дается чуть меньший вес. За замену на близкую по свойствам аминокислоту дается чуть больший вес, чем, в других матрицах, а за замену на аминокислоту с другой функциональной группой дается меньший штраф, чем в BLOSUM62, и гораздо меньший, чем в PHAT_T75_B73 (все видно из таблицы). Что примечательно, триптофан настолько особенный по своей структуре и свойствам, что его замена на что-то другое очень сильно влияет на вес выравнивания.
Сравнение выравниваний мутантов, полученных вручную и с помощью программ needle и water.
В пакете EMBOSS команда needle использует алгоритм Нидлмана-Вунша для глобального выравнивания, а команда water – алгоритм Смита-Ватермана для локального выравнивания. Локальным оптимальным выравниванием называется такое оптимальное выравнивание фрагментов последовательностей, при котором любое удлинение или укорочение фрагментов приводит только к уменьшению веса. Локальному оптимальному выравниванию отвечает путь с наибольшим весом, независимо от того, где он начинается и где кончается. Данные, которые использовали программы в качестве стандартных:
Matrix (матрица весов): EBLOSUM62 Gap_penalty (штраф за гэп): 10.0 Extend_penalty (штраф за продолжение гэпа): 0.5
Мутант 1-1.
| Способ выравнивания | Вручную | needle | water |
| % идентичности | 6/20 = 30% | 6/64 ( 9.4%) | 6/16 (31.2%) |
| % сходства | 9/20 = 45% | 9/20 = 45% | 8/16 (50.0%) |
| вес по матрице BLOSUM62 | 13 | 13.0 | 15.0 |
Needle
Length: 64
P70994/1-64 1 MPYVTVKMLEGRTDEQKRNLVEKVTEAVKETTGASEEKIVVFIEEFTMRK 50
|..|.:..::.|.|.|...|
P70994 1 ---------EAWTHDFAKHHVHKGTMNHK--------------------- 20
P70994/1-64 51 DHYAVAGKRLSDME 64
P70994 20 -------------- 20
Water
Length: 16
P70994/1-64 10 EGRTDEQKRNLVEKVT 25
|..|.:..::.|.|.|
P70994 1 EAWTHDFAKHHVHKGT 16
Вес выравнивания вручную и программой needle не отличаются (13), программа water сделала выравнивание в 16 аминокислотных остатков, что позволило немного увеличить вес выравнивания за счет отсутствия штрафа за несовпадение (15).
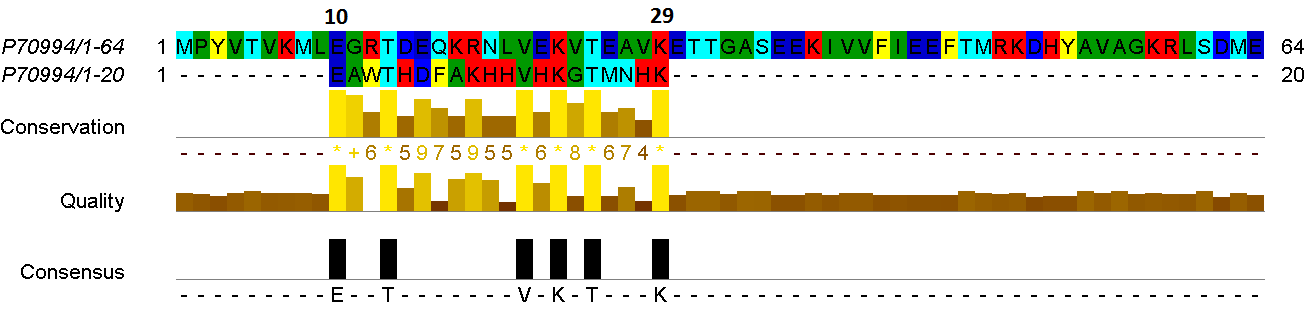
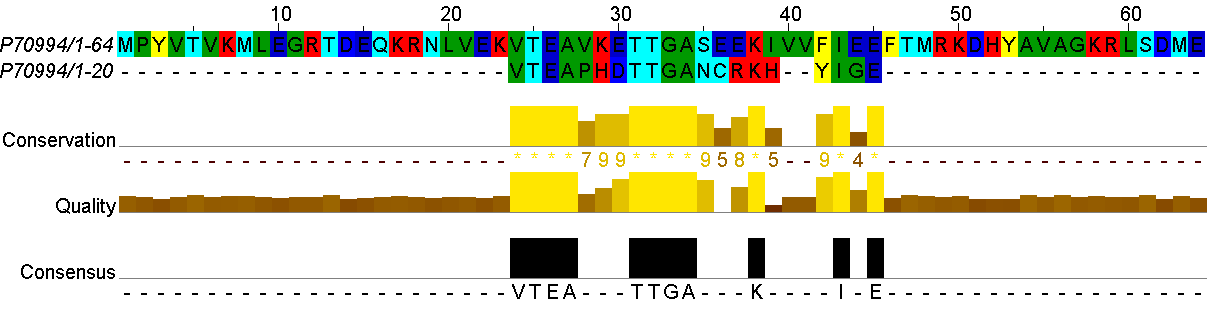
Мутант 1-2.
| Способ вырванивания | Вручную | needle | water |
| % идентичности | 11/22 = 50% | 11/64 (17.2%) | 9/15 (60.0%)) |
| % сходства | 16/22 = 72,72% | 14/64 (21.9%) | 14/64 (21.9%) |
| вес по матрице BLOSUM62 | 38 | 35.0 | 39.0 |
Needle
Length: 64
P70994/1-64 1 MPYVTVKMLEGRTDEQKRNLVEKVTEAVKETTGASEEKIVVFIEEFTMRK 50
||||..:||||:..| .:|.|
P70994 1 -----------------------VTEAPHDTTGANCRK--HYIGE----- 20
P70994/1-64 51 DHYAVAGKRLSDME 64
P70994 20 -------------- 20
Water
Length: 15
P70994/1-64 24 VTEAVKETTGASEEK 38
||||..:||||:..|
P70994 1 VTEAPHDTTGANCRK 15
Здесь значения различаются. Выравнивание вручную сделано с тем учетом, что два гэпа дадут большую выгоду, чем один (38) – я расположила аминокислоты по свойствам. Программа needle сделала один гэп, но рядом оказались аминокислоты более далекие по свойствам, что сделало выравнивание менее выгодным (35.5). А программа water решила ограничиться выравниванием длиной всего в 15 аминокислотных остатков, соответственно, меньше штрафа за несовпадение (39).
Мутант 1-3.
| Способ вырванивания | Вручную | needle | water |
| % идентичности | 12/20 = 60% | 12/64 (18.8%) | 12/20 (60.0%) |
| % сходства | 13/20 = 65% | 13/64 (20.3%) | 13/20 (65.0%) |
| вес по матрице BLOSUM62 | 48 | 48.0 | 48.0 |
Needle
Length: 64
P70994/1-64 1 MPYVTVKMLEGRTDEQKRNLVEKVTEAVKETTGASEEKIVVFIEEFTMRK 50
||||.|...
P70994 1 -----------------------------------------FIEEATPGH 9
P70994/1-64 51 DHYAVAGKRLSDME 64
.:.|.||||||
P70994 10 KNMAFAGKRLS--- 20
Water
Length: 20
P70994/1-64 42 FIEEFTMRKDHYAVAGKRLS 61
||||.|....:.|.||||||
P70994 1 FIEEATPGHKNMAFAGKRLS 20
Для этого выравнивания значения от полученных вручную не отличаются.
Сравнение выравниваний, полученных для полноразмерных мутантов вручную и построенных классическими алгоритмами Нидлмана-Вунша и Смита-Ватермана.
Использовала скрипт evolve_protein.pl для моделирования более близкой к реальности ситуации. Параметры мутантов для исследования указаны в таблице 3. Далее ручное выравнивание представлено картинкой, а программами needle и water - стандартными выдачами программ.
Таблица 3. Параметры эволюции мутантных белков.
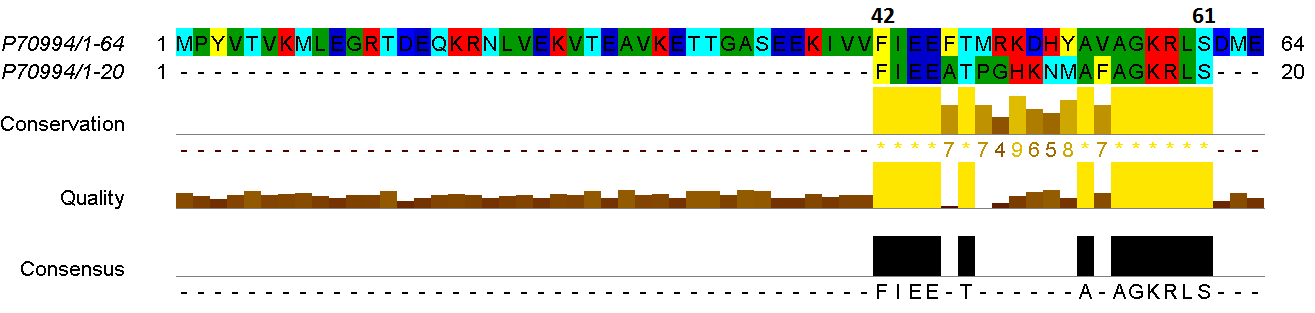
Мутант 3-1.
| % идентичности | 18/20 = 90% |
| % сходства | 18/20 = 90% |
Program: needle
Length: 64
Identity: 18/64 (28.1%)
Similarity: 19/64 (29.7%)
Gaps: 44/64 (68.8%)
Score: 87.0
P70994/1-64 1 MPYVTVKMLEGRTDEQKRNLVEKVTEAVKETTGASEEKIVVFIEEFTMRK 50
.||||:||||||||||||||
generations=1 1 ------------IDEQKQNLVEKVTEAVKETT------------------ 20
P70994/1-64 51 DHYAVAGKRLSDME 64
generations=1 20 -------------- 20
Program: water
Length: 19
Identity: 18/19 (94.7%)
Similarity: 19/19 (100.0%)
Gaps: 0/19 ( 0.0%)
Score: 88.0
P70994/1-64 14 DEQKRNLVEKVTEAVKETT 32
||||:||||||||||||||
generations=1 2 DEQKQNLVEKVTEAVKETT 20
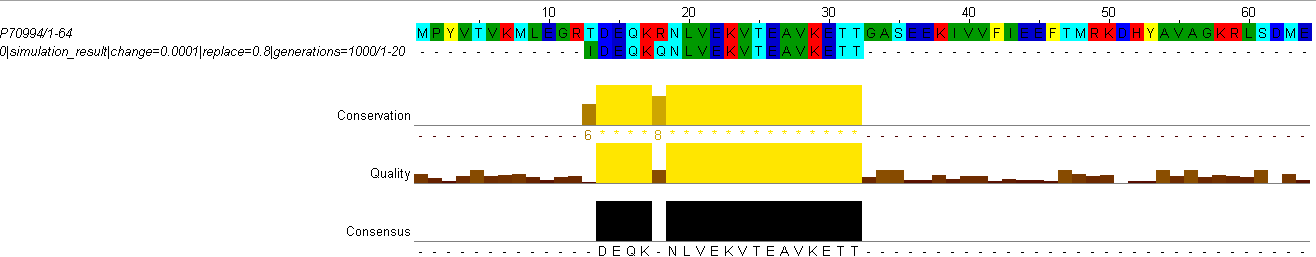
Мутант 3-2.
| % идентичности | 9/20 = 45% |
| % сходства | 9/20 = 45% |
Program: needle
Length: 64
Identity: 10/64 (15.6%)
Similarity: 12/64 (18.8%)
Gaps: 44/64 (68.8%)
Score: 24.5
P70994/1-64 1 MPYVTVKMLEGRTDEQKRNLVEKVTEAVKETTGASEEKIVVFIEEFTMRK 50
|.|:......||||||| |.:
generations=1 1 ----TCKLWCEDDVEQKRNLV--VCK------------------------ 20
P70994/1-64 51 DHYAVAGKRLSDME 64
generations=1 20 -------------- 20
Program: water
Length: 7
Identity: 7/7 (100.0%)
Similarity: 7/7 (100.0%)
Gaps: 0/7 ( 0.0%)
Score: 34.0
P70994/1-64 15 EQKRNLV 21
|||||||
generations=1 11 EQKRNLV 17
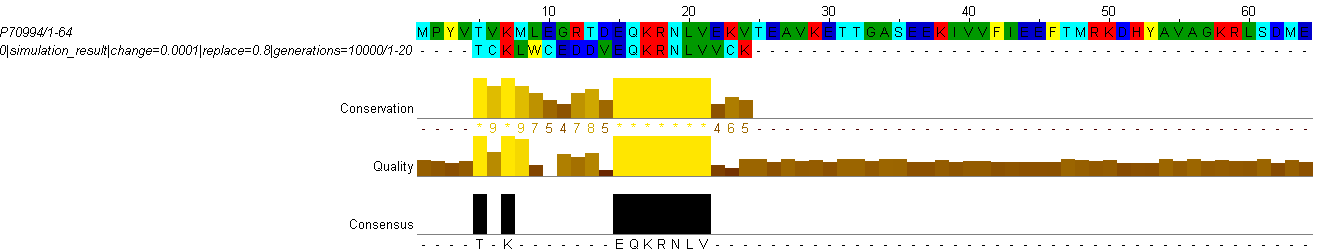
Мутант 3-3.
| % идентичности | 15/20 = 75 |
| % сходства | 16/20 = 80% |
Program: needle
Length: 65
Identity: 18/65 (27.7%)
Similarity: 18/65 (27.7%)
Gaps: 46/65 (70.8%)
Score: 81.0
P70994/1-64 1 MPYVTVKMLEGRTDE-QKRNLVEKVTEAVKETTGASEEKIVVFIEEFTMR 49
||||||||||||||| .|||
generations=1 1 MPYVTVKMLEGRTDEIFKRN------------------------------ 20
P70994/1-64 50 KDHYAVAGKRLSDME 64
generations=1 20 --------------- 20
Program: water
Length: 20
Identity: 18/20 (90.0%)
Similarity: 18/20 (90.0%)
Gaps: 1/20 ( 5.0%)
Score: 81.0
P70994/1-64 1 MPYVTVKMLEGRTDE-QKRN 19
||||||||||||||| .|||
generations=1 1 MPYVTVKMLEGRTDEIFKRN 20
Вывод: чем больше поколений, тем сильнее будут различаться последовательности. Различие можно уменьшить, уменьшая вероятность изменения остатка.
Время эволюции
Для 1000 поколений примерно 21 день, для 10000 поколений - 208 дней.
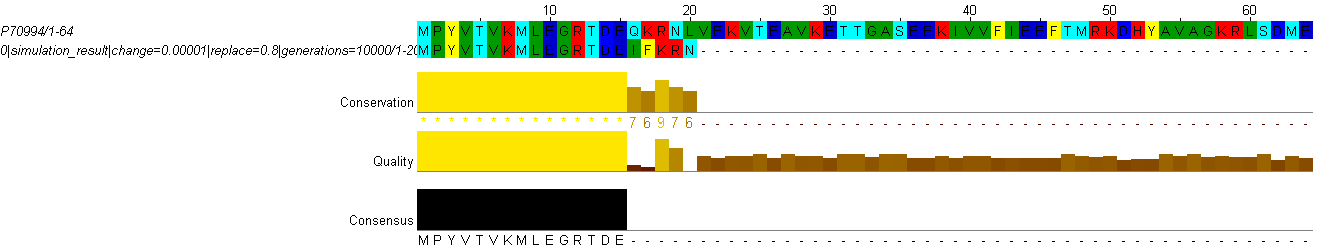
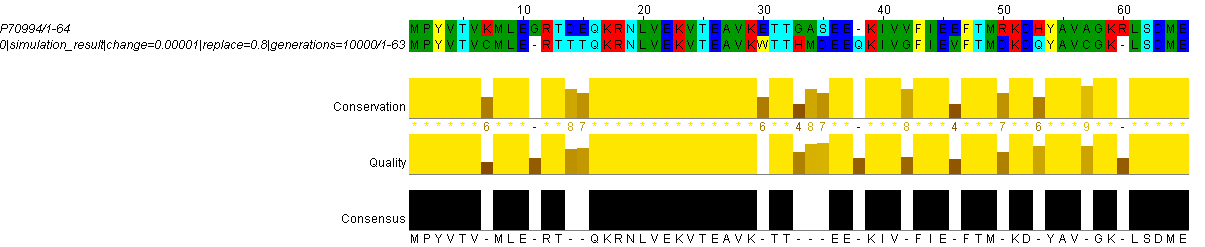
Я провела выравнивание полноразмерного мутанта с вероятностью изменения остатка 0,00001, вероятностью замены остатка 0,8, числом поколений – 10000. Стандартные параметры при использовании программ needle и water не были изменены, выравнивание вручную проводилось с помощью JalView.
Identity: 50/65 = 79.2%
Similarity: 50/65 = 76.9%
Program: needle
Length: 65
Identity: 50/65 (76.9%)
Similarity: 50/65 (76.9%)
Gaps: 3/65 ( 4.6%)
Score: 199.0
P70994/1-64 1 MPYVTVKMLEGRTDEQKRNLVEKVTEAVKETTGASEE-KIVVFIEEFTMR 49
||||||.||| ||..||||||||||||||.||...|| |||.|||.|||.
generations=1 1 MPYVTVCMLE-RTTTQKRNLVEKVTEAVKWTTHMDEEQKIVGFIEVFTMD 49
P70994/1-64 50 KDHYAVAGKRLSDME 64
||.|||.|| |||||
generations=1 50 KDQYAVCGK-LSDME 63
Program: water
Length: 65
Identity: 50/65 (76.9%)
Similarity: 50/65 (76.9%)
Gaps: 3/65 ( 4.6%)
Score: 199.0
P70994/1-64 1 MPYVTVKMLEGRTDEQKRNLVEKVTEAVKETTGASEE-KIVVFIEEFTMR 49
||||||.||| ||..||||||||||||||.||...|| |||.|||.|||.
generations=1 1 MPYVTVCMLE-RTTTQKRNLVEKVTEAVKWTTHMDEEQKIVGFIEVFTMD 49
P70994/1-64 50 KDHYAVAGKRLSDME 64
||.|||.|| |||||
generations=1 50 KDQYAVCGK-LSDME 63
Неудивительно, что выравнивания вручную и программами не отличаются по идентичности и сходству – у мутанта была небольшая абсолютная вероятность изменения остатка. Общий вид выравнивания также совпадает.