Реконструкция филогенетических деревьев
Пользуясь таксономическим сервисом NCBI, определила, к каким таксонам относятся отобранные мной бактерии (табл. 1).
Таблица 1. Таксономическая принадлежность выбранных мной бактерий.
| Класс | Порядок | Семейство | Род | Вид |
| Clostridia | Clostridiales | Clostridiaceae | Clostridium | Clostridium botulinum |
| Clostridia | Clostridiales | Clostridiaceae | Clostridium | Clostridium tetani |
| Bacilli | Lactobacillales | Enterococcaceae | Enterococcus | Enterococcus faecalis |
| Clostridia | Clostridiales | Clostridiales Family XI. Incertae Sedis | Finegoldia | Finegoldia magna |
| Bacilli | Lactobacillales | Lactobacillaceae | Lactobacillus | Lactobacillus acidophilus |
| Bacilli | Lactobacillales | Lactobacillaceae | Lactobacillus | Lactobacillus delbrueckii |
| Bacilli | Lactobacillales | Streptococcaceae | Lactococcus | Lactococcus lactis |
| Bacilli | Lactobacillales | Streptococcaceae | Streptococcus | Streptococcus pyogenes |
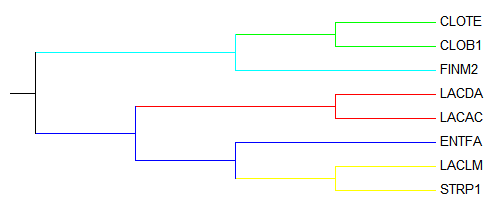
На дереве отобранных бактерий есть ветви, выделяющие таксоны (рисунок 1); ветви, соответствующие разным таксонам, выделены разными цветами, обозначенными в таблице 1.
Рисунок 1. Филогенетическое дерево выбранных организмов.
Из списка функций белков выбрала одну - фактор элонгации трансляции Ts, по белкам соответствующего семейства далее будет реконструировано филогенетическое дерево. Получила из Swiss-Prot последовательности белков с данной функцией из отобранных бактерий с помошью команды seqret.
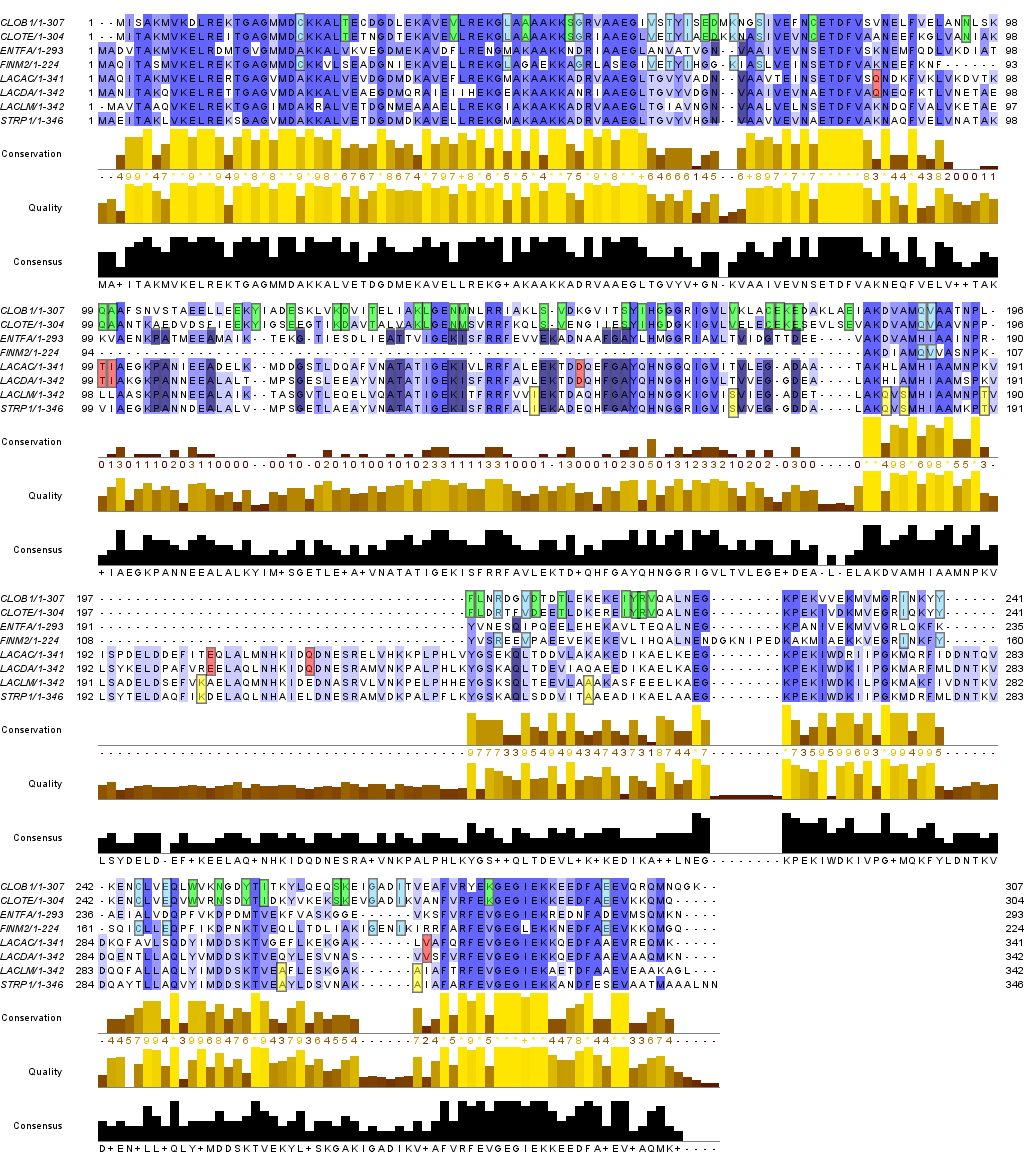
Поместила последовательности в один fasta-файл и отредактировала названия последовательностей, оставив только мнемонику видов. Создала выравнивание отобранных белков с помощью Web service в JalView (ссылка на проект в формате jar) и (ссылка на выравнивание в формате fasta). Ниже приведено изображение выравнивания (рисунок 1) в блочном формате (Format > Wrap) с блоками шириной 100 остатков, с раскраской по проценту идентичности.
В данном выравнивании есть диагностические позиции, которые выделены цветными прямоугольниками на рисунке 1. Цвета прямоугольников таксонов соответствуют цветам таксонов в таблице 1.
По выделенным диагностическим позициям видно, что много диагностических позиций, соответствующих порядкам.
Рисунок 2. Выравнивание белков, созданное в программе Jalview, и выделенные прямоугольниками диагностические позиции таксонов.
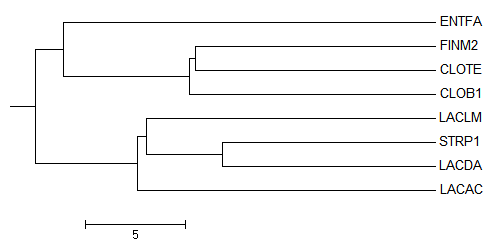
Реконструировала филогенетическое дерево четырьмя методами, доступными из JalView (меню Calculate > Calculate tree). Каждое дерево сохранила в Newick-формате в файл с соответствующим названием. В отчёте привела изображения деревьев, сделанные программой Mega (рисунки 3-6).
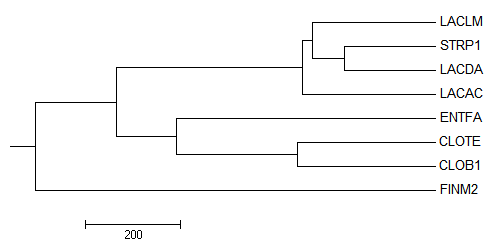
Рисунок 3. Филогенетическое дерево выбранных организмов с использованием параметров average distance using % identity.
В данном дереве есть две общая ветвь с правильным деревом - {CLOB1, CLOTE, FINM2} против {ENTFA, LACAC, LACDA, LACLM, STRP1} и {CLOB1, CLOTE} против {FINM2, ENTFA, LACAC, LACDA, LACLM, STRP1}, а все остальные ветви "уникальны":
- {CLOB1, CLOTE, FINM2, ENTFA} против {LACAC, LACDA, LACLM, STRP1}
- {LACAC, LACDA, STRP1} против {ENTFA, CLOB1, CLOTE, FINM2, LACLM}
- {LACDA, STRP1} против {ENTFA, CLOB1, CLOTE, FINM2, LACAC, LACLM}
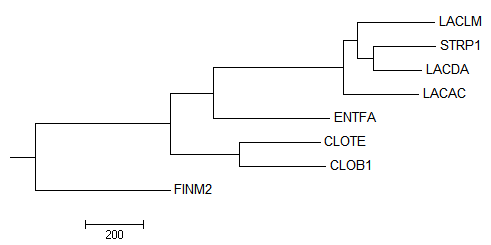
Рисунок 4. Филогенетическое дерево выбранных организмов с использованием параметров neighbour joining using % identity.
Те же ветви одинаковы с правильным деревом: {CLOB1, CLOTE, FINM2} против {ENTFA, LACAC, LACDA, LACLM, STRP1} и {CLOB1, CLOTE} против {FINM2, ENTFA, LACAC, LACDA, LACLM, STRP1}; отличаются:
- {CLOB1, CLOTE, FINM2, ENTFA} против {LACAC, LACDA, LACLM, STRP1}
- {LACDA, STRP1} против {ENTFA, CLOB1, CLOTE, FINM2, LACAC, LACLM}
- {LACAC, LACDA, STRP1} против {ENTFA, CLOB1, CLOTE, FINM2, LACLM}
Рисунок 5. Филогенетическое дерево выбранных организмов с использованием параметров average distance using BLOSUM62.
Здесь сохранилась только ветвь {CLOB1, CLOTE} против {FINM2, ENTFA, LACAC, LACDA, LACLM, STRP1}, а появились ветви:
- {CLOB1, CLOTE, ENTFA} против {LACAC, LACDA, LACLM, STRP1, FINM2}
- {LACAC, LACDA, LACLM, STRP1} против {CLOB1, CLOTE, FINM2, ENTFA}
- {LACDA, STRP1} против {ENTFA, CLOB1, CLOTE, FINM2, LACAC, LACLM}
- {LACLM, LACDA, STRP1} против {ENTFA, CLOB1, CLOTE, FINM2, LACAC}
Рисунок 6. Филогенетическое дерево выбранных организмов с использованием параметров neighbour joining using BLOSUM62.
Здесь также сохранилась только ветвь {CLOB1, CLOTE} против {FINM2, ENTFA, LACAC, LACDA, LACLM, STRP1}, появились ветви:
- {CLOB1, CLOTE, ENTFA} против {LACAC, LACDA, LACLM, STRP1, FINM2}
- {LACAC, LACDA, LACLM, STRP1} против {CLOB1, CLOTE, FINM2, ENTFA}
- {LACDA, STRP1} против {ENTFA, CLOB1, CLOTE, FINM2, LACAC, LACLM}
- {LACLM, LACDA, STRP1} против {ENTFA, CLOB1, CLOTE, FINM2, LACAC}
- {LACAC, LACDA, LACLM, STRP1, ENTFA} против {CLOB1, CLOTE, FINM2}
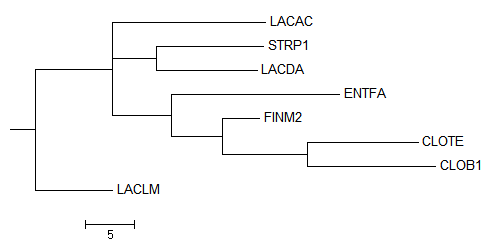
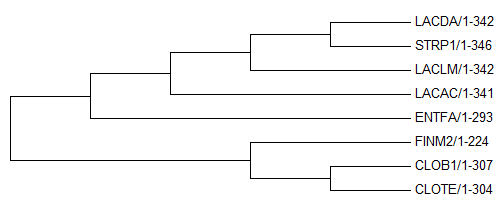
Импортировала выравнивание (файл в fasta-формате) в программу Mega, выбрав при импорте "Analyze". Реконструировала дерево методом "Maximum Parsimony". Укоренила дерево в наиболее правильную, с моей точки зрения, ветвь. Ниже приведено изображение дерева (рисунок 7).
Рисунок 7. Укорененное дерево выбранных организмов, сделанное в программе Mega.
Единственное, что возможно было сделать с деревом, полученным таким методом, чтобы сделать его похожим на правильное, это сделать укоренение таким образом, чтобы таксономически правильно были разделены порядки. Данное дерево отличается от правильного тем, что в нем содержит ветви:
- {LACDA, STRP1} против {ENTFA, CLOB1, CLOTE, FINM2, LACAC, LACLM},
- {LACLM, STRP1, LACDA} против {CLOB1, CLOTE, FINM2, LACAC, ENTFA},
- {LACLM, STRP1, LACDA, LACAC} против {CLOB1, CLOTE, FINM2, ENTFA}.