Практикум 9
Для выполнения задания я получил список идентификаторов Swiss-Prot с заданной мнемоникой организма, используя две команды:infoseq 'sw:*_ecoli (и *_bacsu)' -only -name -nohead -out ECOLI.txt (и BACSU.txt)
cut -f 1 -d '_' ECOLI.txt BACSU.txt | sort | uniq -d > common_mnems.txt
Затем я выбрал 3 произвольных мнемоники(GLNK, PURU и FOLD), которые использовал в последующих задачах.
Глобальное выравнивание гомологичных белков
needle sw:*_ecoli sw:*_bacsu *.needle -auto - именно эта команда использовалась для рассчетов глобального выравнивания.
Результаты можно увидеть в таблице:
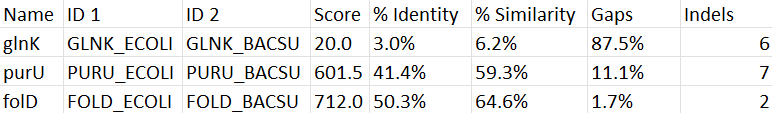
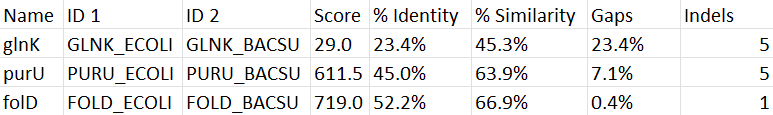
Локальное выравнивание гомологичных белков
water sw:*_ecoli sw:*_bacsu *.water -auto - по аналогии с предыдущей командой, только здесь мы используем water вместо needle
Выравнивание негомологичных белков
Методика такая же, сначала использовал needle, потом water.Результаты ниже:


Множественное выравнивание
Для выполнения данного задания я выбрал 1 из белков, рассматриваемых в 1 задании: PURU_*, где * - принадлежность к какому-либо организму.Мой выбор пал на следующих кандидатов:
PURU_ECOLI, PURU_BACSU, PURU_MYCBO, PURU_SHIFL, PURU_CORS1, PURU_HAEIN
Множественное выравнивание я делал 2-м указанным в подсказке способом - с помощью Jalview.
Результаты выполненного выравнивания здесь: