Учебный сайт Дюгая Ильи
A- и В- формы ДНК. Структура РНК
Для выполнения задания построил модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA: с помощью программы Рutty, используя протокол ssh, подключился к kodomo.cmm.msu.ru, перешел в директорию term3/block1/pr2. Ввел следующие команды, что бы указать путь к 3DNA:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
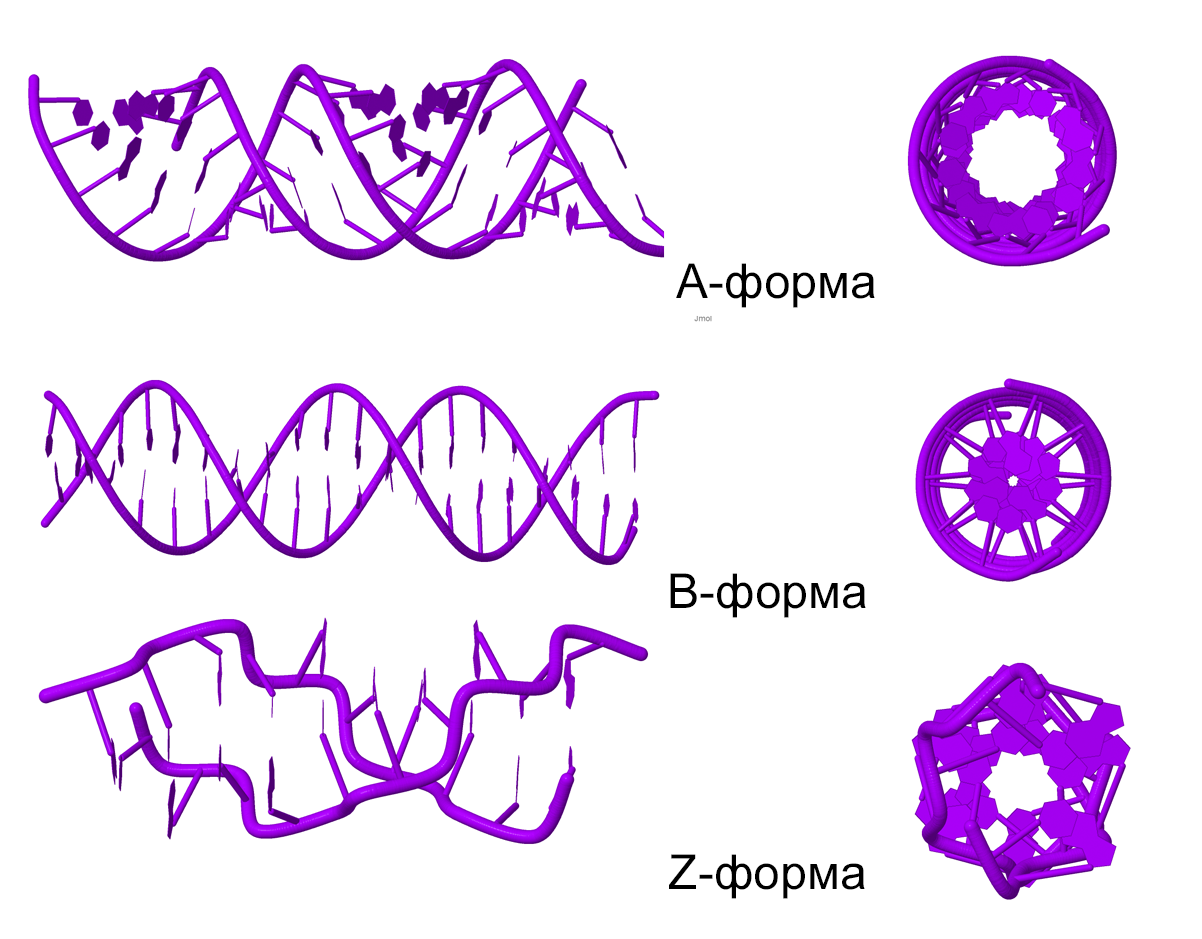
С помощью программы fiber пакета 3DNA построил A-, B- и Z-форму дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 повторений последовательности "gatc". Структуру дуплекса в А-форме сохранил в файле gatc-a.pdb, структуру дуплекса в В-форме - в файле gatc-b.pdb, структуру дуплекса в Z-форме - в файле gatc-z.pdb. Трехмерные модели изображены на рис. 1.
Рис. 1. Модели структур A-, B- и Z-формы ДНК, построенные с помощью инструментов пакета 3DNA.
Средства JMol для работы со структурами нуклеиновых кислот.
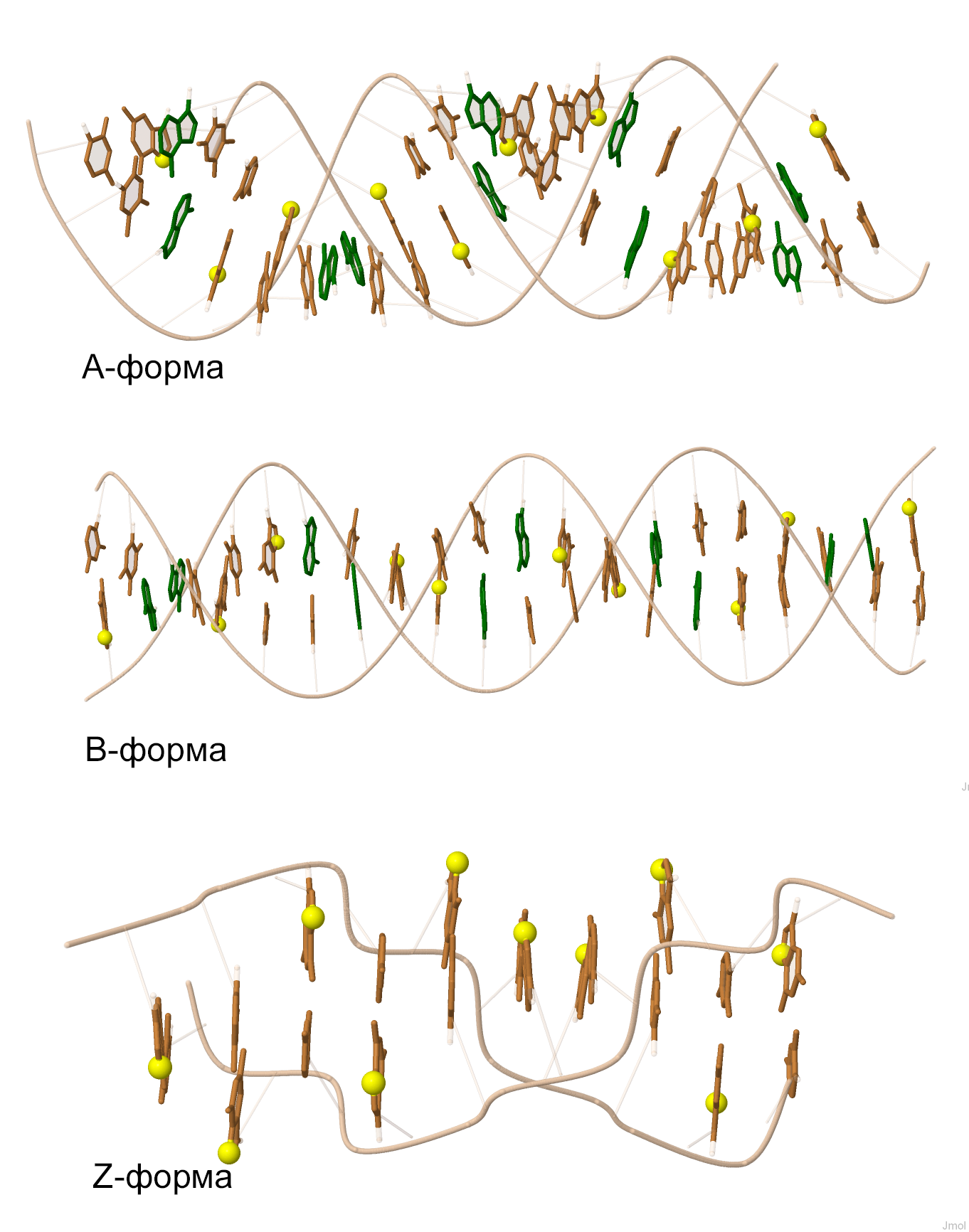
Научился выделять различные атомы и химические группировки, используя множества JMol. Выделил цветом или способом отображения сахарофосфатный остов ДНК, все нуклеотиды, все аденины, атом N7 во всех гуанинах (рис. 2).
Рис. 2. Модели структур A-, B- и Z-формы ДНК. Бежевым выделен сахарофосфатный остов, все нуклеотиды имеют форму палочек, зеленым цветом обозначены аденины, жёлтые шары - все атомы N7 в гуанинах.
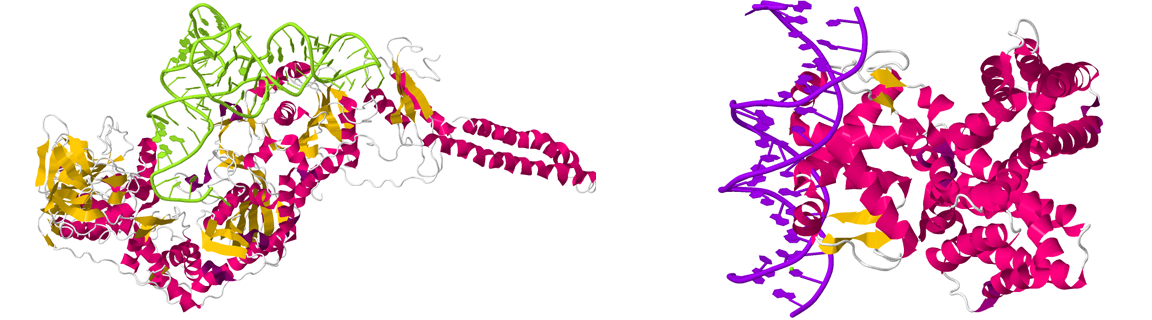
Получил файлы PDB по заданным в таблице идентификаторам (1eiy - фенилаланин-тРНК синтетаза; 1hw2 - FadR-ДНК комплекс), выбрал подходящую структуру (рис. 3).
Рис. 3. Фенилаланин-тРНК синтетаза (слева) и FadR-ДНК комплекс (справа). Окрашены в соответствии со вторичной структурой: светло-зеленый - РНК, фиолетовый - ДНК.
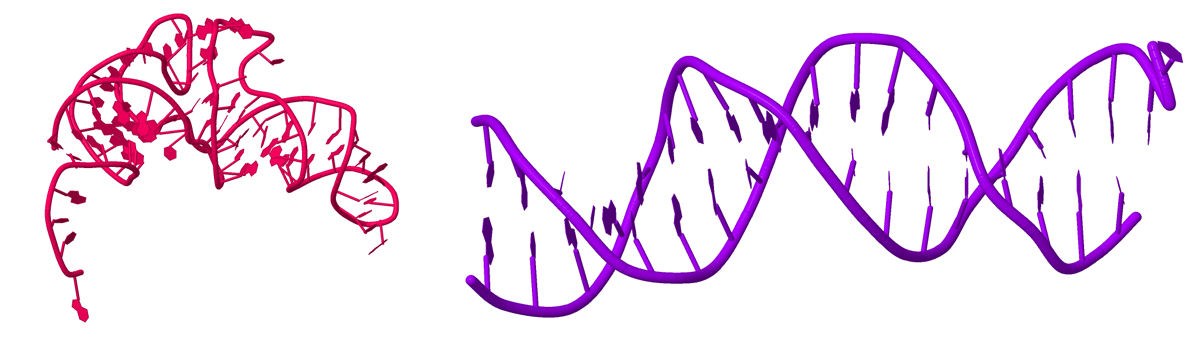
Открыл полученные из PDB файлы в JMol. Получил изображение только нуклеиновой кислоты (рис. 4). Внимательно рассмотрел и проверил заданные структуры ДНК и РНК на наличие разрывов.
Рис. 4. РНК из структуры фенилаланин-тРНК синтетазы (слева) и ДНК из структуры FadR-ДНК комплекса (справа).
Сохранил координаты атомов только ДНК и РНК в отдельных файлах для дальнейшей работы.
Сравнение структур 3-х форм ДНК с помощью JMol.
Открыл в JMol файл gatc-b.pdb, полученный при выполнении задания 1. Рассмотрел структуру и визуально определил большую и малую бороздку. Выбрал заданный мне гуанин в середине структуры (рис. 5). Определил, какие атомы основания явно обращены в сторону большой бороздки (выделены красным), и какие - в сторону малой (выделены синим, рис. 6).
Рис. 5, 6. Гуанин в середине структуры В-спирали ДНК. Синим выделены атомы, обращенные в сторону малой бороздки, красным - в сторону большой бороздки.
Сравнил основные спиральные параметры разных форм ДНК (таблица 1).
Таблица 1. Основные спиральные параметры для разных форм ДНК.
| A-форма | B-форма | Z-форма | |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (Â) | 28.03 | 33.75 | 43.50 |
| Количество оснований на один виток | 11 | 10 | 12 |
| Ширина большой бороздки (Â) | 16.81 | 17.91 | 18.30 |
| Ширина малой бороздки (Â) | 7.98 | 11.69 | 8.68 |
Все измерения проводились от фосфата гуанина.
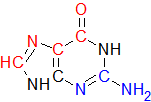
В упражнении 3 задания 2 требовалось измерить торсионные (двугранные) углы между атомами в одном нуклеотиде для А- и В-форм ДНК. Нам было предложено сравнить полученные результаты со значениями углов в презентации, любезно предоставленной Головиным А.В. Фрагмент презентации изображён на рис. 6.
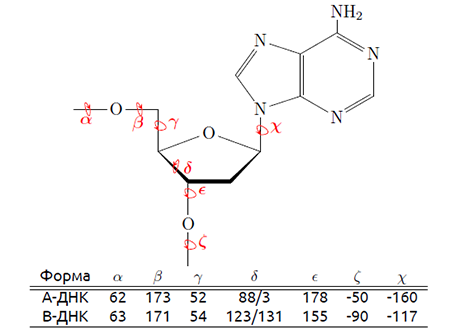
Измерил торсионные углы гуанина. Сравнил значения углов в А- и В-форме со значениями из презентации (таблица 2).
Таблица 2. Сравнение значений углов в А- и В-форме со значениями, приведенными в презентации.
| α | β | γ | δ | ε | ξ | χ | |
| А-форма (RasMol) | -64.11 | 174.80 | 41.70 | 79.09 | 157.76 | -65.12 | -157.20 |
| А-форма (презентация) | -62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| B-форма (RasMol) | -85.87 | 136.35 | 31.14 | 143.40 | 146.77 | -100.52 | -107.92 |
| B-форма (презентация) | -63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
По этим данным видно, что значения торсионных углов для В-формы, в частности, первые три угла, значительно отличаются от значений в презентации.
Таблица 3. Торсионные углы тРНК из структуры 1eiy.
| # | base | α (P - O5') |
β (O5' - C5') |
γ (C5' - C4') |
δ (C4' - C3') |
ε (C3' - O3') |
ζ (O3' - P) |
χ (C1' - N) |
| 1 | G | --- | -172.2 | 178.7 | 101.1 | -166.1 | -43.9 | 169.2 |
| 2 | C | -134.3 | -120.6 | 52.8 | 82.5 | -175.1 | -59.6 | -154.2 |
| 3 | G | -99.8 | 173.3 | 82.9 | 78.5 | -144.4 | -83.7 | -169.1 |
| 4 | G | -69.9 | 161.6 | 66.3 | 81.2 | -153.8 | -64.8 | -175.4 |
| 5 | A | -73.5 | -175 | 51 | 83.9 | -151 | -66.3 | -160.8 |
| 6 | U | -69 | 164.8 | 64.2 | 79.6 | -151.4 | -75.9 | -172 |
| 7 | U | -53.6 | -180 | 58 | 138.4 | --- | --- | -127.8 |
| 8 | C | --- | 172.6 | 40.7 | 84.7 | -152.7 | -77.3 | -172.3 |
| 9 | U | -48.3 | 177.4 | 37 | 85 | -149.6 | -79.8 | -165.1 |
| 10 | G | -41.5 | 174.5 | 30.8 | 87.2 | -154.6 | -73 | -154.9 |
| 11 | U | -62.9 | 167.1 | 50.5 | 81.4 | -160 | -66.5 | -167.7 |
| 12 | G | -59.6 | 173.1 | 53.8 | 83.4 | -167 | -60.4 | -173.3 |
| 13 | U | -85.6 | -167.6 | 58 | 80.1 | -136 | -68.8 | -156.1 |
| 14 | P | -53.7 | 170.2 | 44.2 | 79.7 | --- | --- | -153.2 |
| 15 | A | --- | 149 | 58.5 | 80.1 | --- | --- | -154.5 |
| 16 | A | --- | -174.6 | 60.3 | 75.3 | -146.6 | -62.2 | -173.4 |
| 17 | P | -82.9 | 179.9 | 69.4 | 84.4 | -151.5 | -69.3 | -172.9 |
| 18 | C | -90.8 | 168.7 | 74.2 | 81.3 | -141.7 | -96.3 | -176.3 |
| 19 | U | 41.1 | 162.3 | -47.4 | 99.2 | -152.5 | -68.5 | -155.8 |
| 20 | G | -57.3 | 162.1 | 58.1 | 83.3 | -156.7 | -80.8 | -170.1 |
| 21 | G | -53.1 | 168.4 | 51.4 | 83.8 | -151.2 | -77.8 | -160 |
| 22 | A | -71.8 | 156.4 | 70 | 84.2 | --- | --- | -158.3 |
| 23 | G | --- | 148.4 | 57 | 91.2 | -134.6 | -76.3 | 173.4 |
| 24 | C | -75.1 | 179 | 58.4 | 89.4 | -145.8 | -71.1 | -161.4 |
| 25 | U | -69.4 | 173 | 49.4 | 82.8 | -162 | -73.4 | -160.1 |
| 26 | C | 138 | -155.7 | -163.9 | 79.3 | -169.9 | -79.1 | -168.2 |
| 27 | A | 131.3 | 177.1 | -153.1 | 91.4 | -117.3 | -65.5 | -175.3 |
| 28 | G | -50 | 160.2 | 41.4 | 80.4 | --- | --- | -166.8 |
| 29 | G | --- | 176.6 | 59.6 | 157.4 | --- | --- | -79.7 |
| 1 | C | -91.7 | -160.7 | 61.3 | 81.3 | --- | --- | -160 |
| 2 | G | -78.2 | 170.1 | 61.7 | 81.8 | -166.5 | -54 | -164 |
| 3 | C | -70.1 | 168.4 | 54.8 | 77.5 | -156 | -69.4 | -159.6 |
| 4 | U | -59.2 | 171.4 | 47.2 | 82.2 | -154.8 | -78.8 | -162.4 |
| 5 | U | -60.4 | 173.4 | 46.4 | 82.3 | -155.3 | -64.4 | -161.8 |
| 6 | A | -62.7 | 171.9 | 48.5 | 89.2 | -158.3 | -77 | -156.3 |
| 7 | A | -65.7 | 175.3 | 58.8 | 80.3 | -148.2 | -74.4 | -166.2 |
| 8 | G | -41.4 | 157.8 | 48.1 | 79.9 | -154.7 | -74.5 | -172.5 |
| 9 | A | -56.7 | 176.1 | 38 | 77 | -146 | -75.8 | -160.7 |
| 10 | C | -81.5 | 175.4 | 62.8 | 82.4 | -150.5 | -75.6 | -157.3 |
| 11 | A | -73.1 | -173 | 51.5 | 83.1 | -157.4 | -67.8 | -161.9 |
| 12 | C | --- | 179.3 | 33.8 | 83.2 | -157.5 | -69.9 | -170 |
| 13 | A | --- | -150.7 | 69 | 155.7 | --- | --- | -85.3 |
| 14 | G | --- | 137.1 | -67.9 | 177.7 | --- | --- | -95.6 |
| 15 | U | -98.3 | -149.6 | 56.2 | 87.7 | --- | --- | -145.6 |
| 16 | C | -71.3 | -179.5 | 48.3 | 85.7 | -164.2 | -49.8 | -144.8 |
| 17 | A | -71.6 | -175.1 | 49.1 | 87.7 | -143.4 | -62.1 | -158.2 |
| 18 | G | -58.6 | -176.7 | 38.8 | 82 | -166.6 | -56.7 | -157.4 |
| 19 | A | -56.4 | 165.2 | 52.3 | 82.8 | -159.9 | -72.3 | -164.8 |
| 20 | C | -72 | 175 | 53.7 | 81.3 | -147.3 | -76.3 | -162.5 |
| 21 | C | -60.7 | 171.5 | 48.8 | 81.8 | -148 | -73.6 | -163.5 |
| 22 | G | -85.1 | -177.4 | 62.6 | 81.8 | -136.5 | -71.6 | -163.1 |
| 23 | C | -57.7 | 169 | 47.8 | 77.5 | -161.1 | -49.3 | -161.7 |
| 24 | G | -51.9 | 175.1 | 32.1 | 88.3 | -147.4 | -66.7 | -158.2 |
| 25 | A | -59.8 | 179.9 | 47.3 | 84.3 | -152 | -73.4 | -161.4 |
| 26 | G | --- | 161.3 | 178.2 | 87.3 | -148.6 | -78.5 | -176.7 |
| 27 | U | --- | -140.9 | 50.3 | 81.2 | --- | --- | -159.6 |
| 28 | C | --- | -175.3 | 153.2 | 138.4 | --- | --- | -143.1 |
| 29 | C | --- | 175.1 | 47.1 | 85.8 | --- | --- | -163.9 |
| # | среднее значение |
-56.5 | 75.9 | 45.3 | 88.4 | -152.4 | -70.6 | -153.2 |
С помощью Microsoft Excel были проанализированны торсионные углы тРНК структуры 1eiy, было получено среднее значение для каждого угла.
Определение структуры водородных связей.
Получено изображение тРНК из pdb, раскрашенное с выделением стеблей. (см. Рисунок 7).
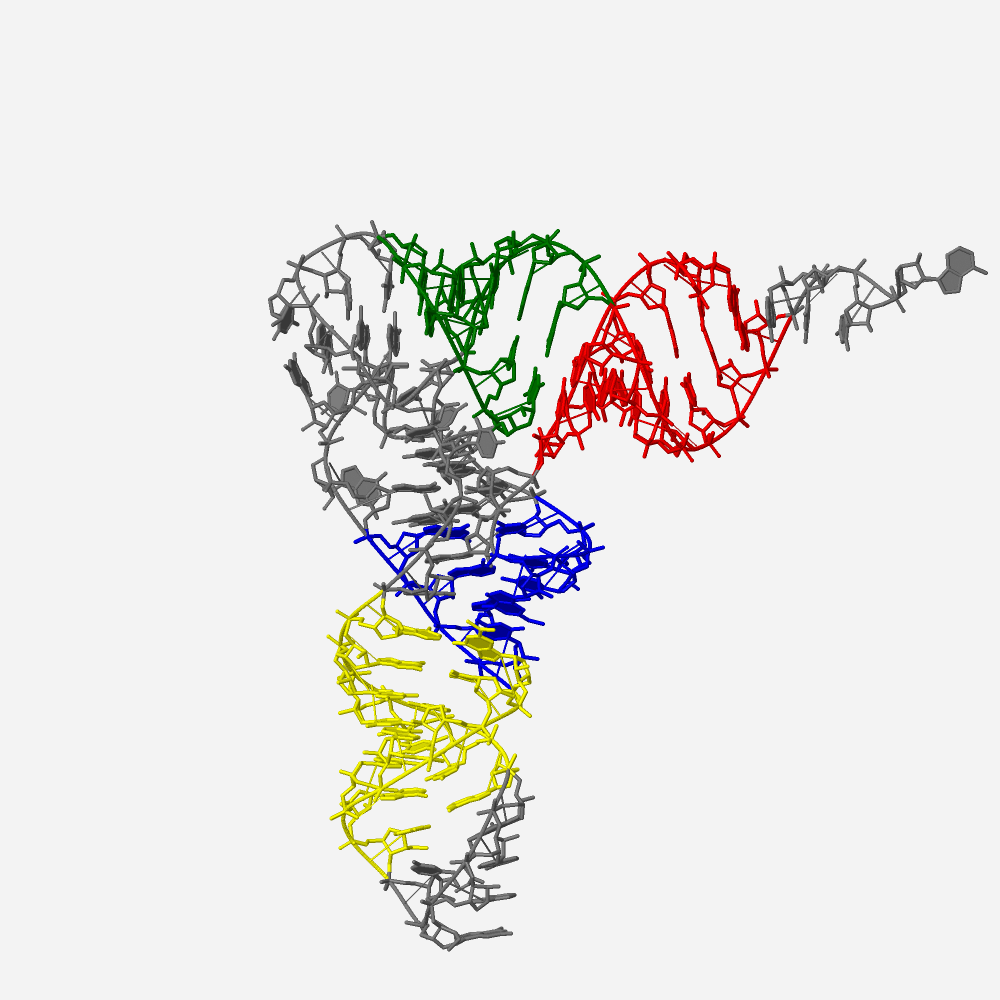
Рис. 7 Cтебли в тРНК, информация о которых получена при помощи програмного пакета 3DNA: красным цветом выделен акцепторный стебель, зеленым Т-стебель, синим D-стебель и желтым антикодоноый стебель. Нуклеотиды не участвующие в образовании стеблей показаны серым.
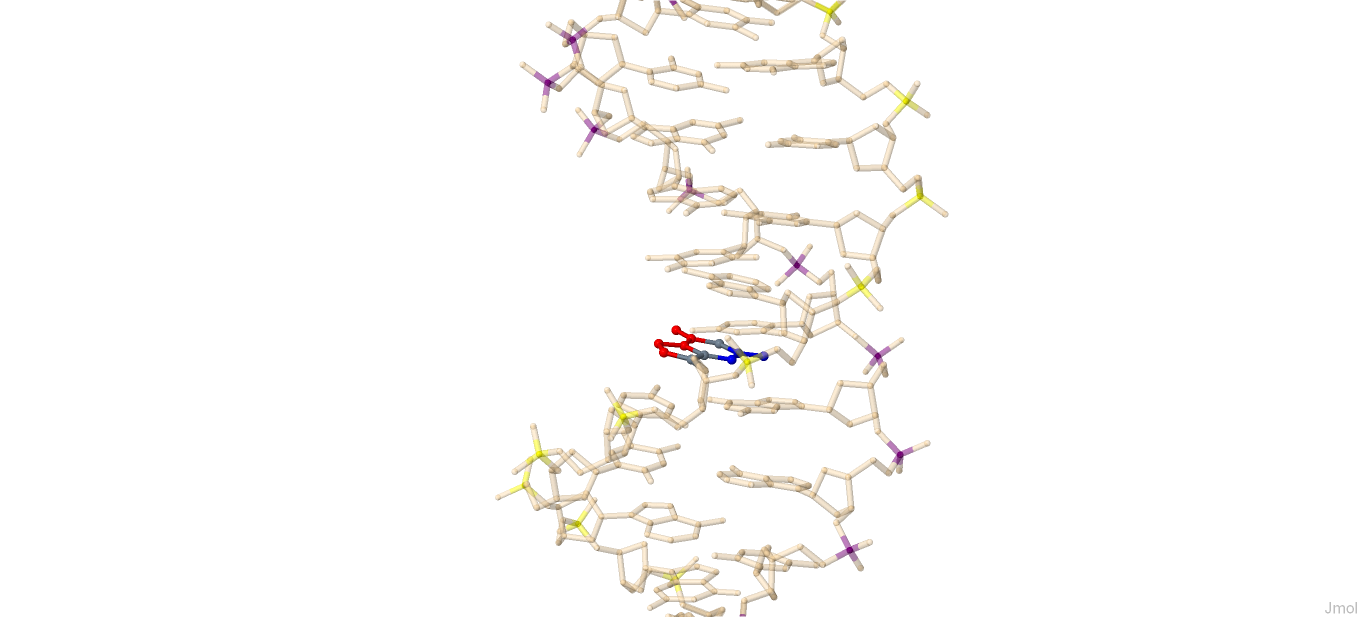
Неканоничные (или модифицированные) взаимодействия происходят между следующими парами нуклеотидов:
4 (0.018) A:...4_:[..G]G-*---U[..U]:..69_:A (0.015) | 7 (0.008) A:...7_:[..U]Ux----A[..A]:..66_:A (0.016) | 13 (0.013) A:..54_:[5MU]u-**-xa[1MA]:..58_:A (0.012) | 15 (0.015) A:..36_:[..A]Ax*---U[..U]:..33_:A (0.014) | 16 (0.008) A:..38_:[..A]A-*---c[OMC]:..32_:A (0.008) | 17 (0.004) A:..39_:[PSU]P-*---A[..A]:..31_:A (0.007) | 22 (0.015) A:..44_:[..A]Ax*---g[M2G]:..26_:A (0.013) | 27 (0.025) A:..14_:[..A]A-**-xU[..U]:...8_:A (0.016) |
Водородные взаимодействия, стабилизирующие структуру, происходят между следующими парами нуклеотидов: 54----58 и 55----18, они стабилизируют Т-петлю
Поиск стэкинг-взаимодействия.
Нашел стекинг взаимодействия с наибольшим и наименьшим перекрытием. (Рисунок 8)
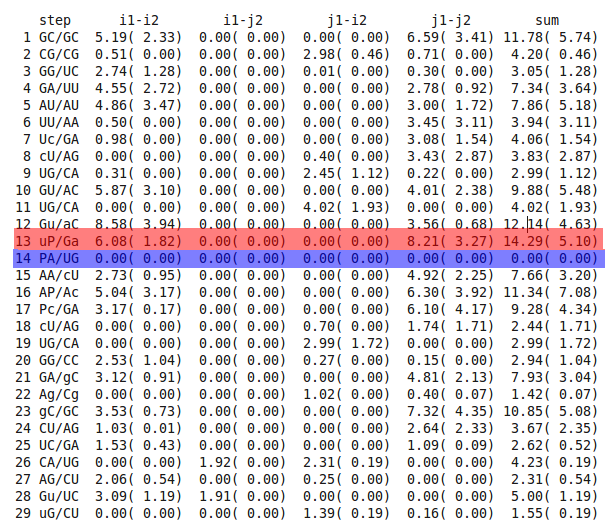
Рис. 18 Данные программы analyze для выбора анализируемых пар нуклеотидов с наибольшим и наименьшим перекрытием.
Для анализа стекинг взаимодействия отобраны структуры №13 и №14 с наибольшим и наименьшим перекрытием соответственно. Для получения графических изображений этих взаимодействий на Рисунке 17 и Рисунке 18 был написан следующий bash-скрипт берущий данные из выходного файла analyze
bash-скрипт для получения Рисунка 9 и Рисунка 10
ex_str -13 stacking.pdb step13.pdb
ex_str -14 stacking.pdb step14.pdb
stack2img -cdolt step13.pdb step13.ps
stack2img -cdolt step14.pdb step14.ps
convert step13.ps fig_17.png
convert step14.ps fig_18.png
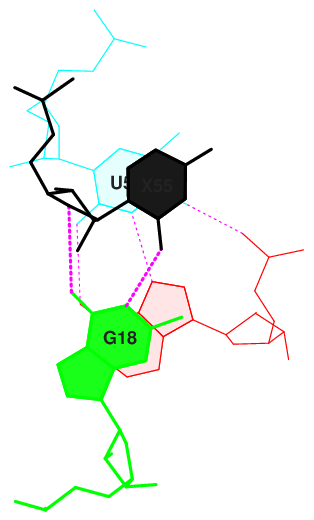
Рис. 9 Графическое изображение нуклеотидов с наибольшим перекрытием.

Рис. 10 Графическое изображение нуклеотидов с наименьшим перекрытием.
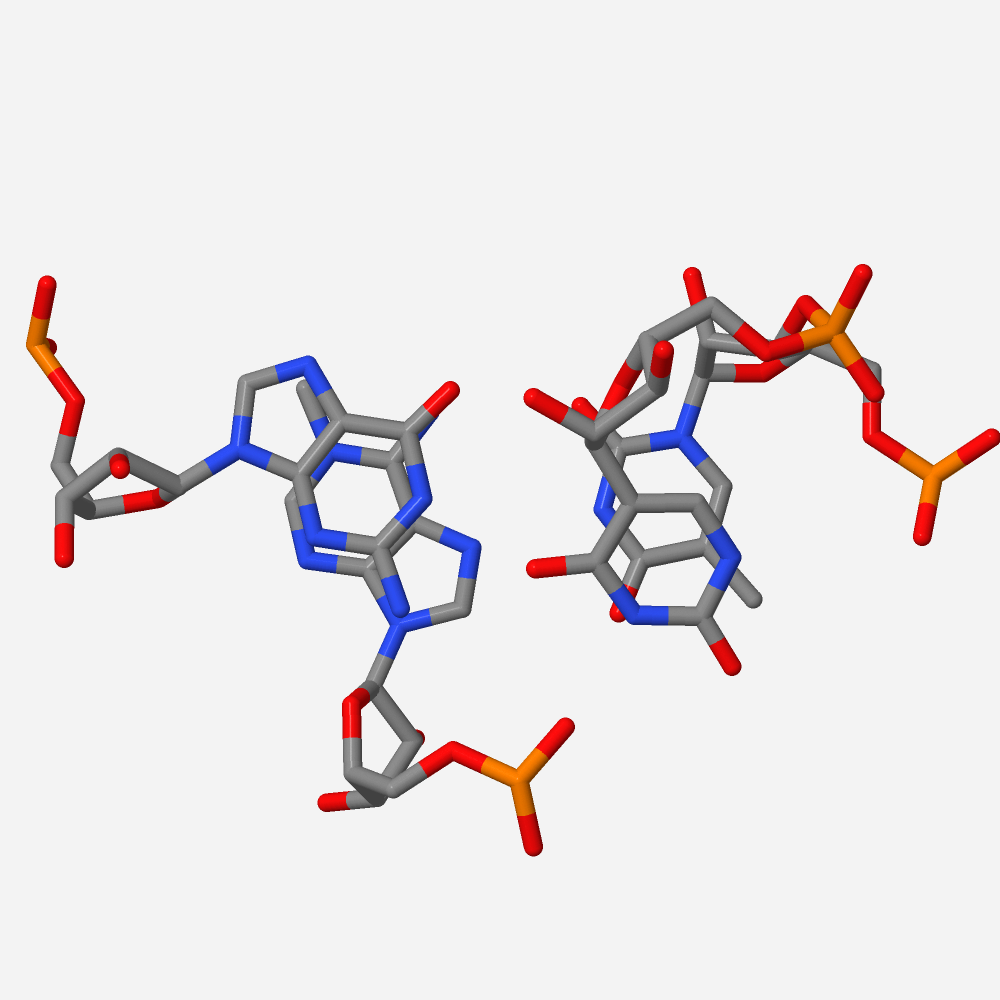
Рис. 11 Графическое изображение в программе Jmol нуклеотидов с наибольшим перекрытием.
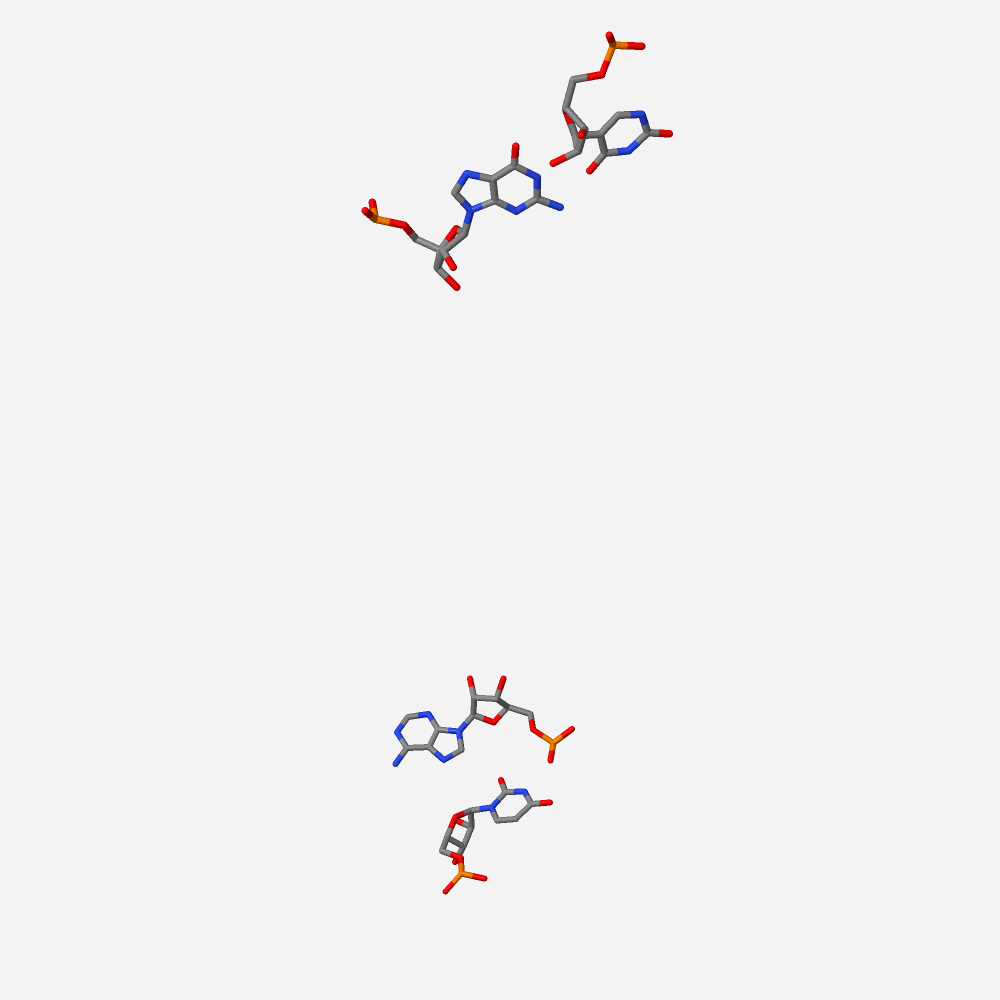
Рис. 12 Графическое изображение в программе Jmol нуклеотидов с наименьшим перекрытием.