Учебный сайт Дюгая Ильи
Построение дерева по нуклеотидным последовательностям
Я построил филогенетическое дерево выбранных бактерий на основе нуклеотидных последовательностей РНК малой субъединицы рибосомы (16S rRNA).
Последовательности РНК каждой бактерии были взял из файлов с расширением .frn (из базы полных геномов NCBI), записал в один файл. Затем построил выравнивание с помощью Muscle, открыл его в MEGA и реконструировал дерево методом Neighbor-Joining. Полученное дерево можно увидеть на рис. 1.
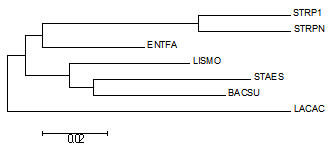
Рис. 1. Изображение филогенетического дерева выбранных видов бактерий отдела Firmicutes, построенного на основе выравнивания последовательностей РНК малой субъединицы рибосомы с помощью метода Neighbor-Joining.
На полученном дереве нет нетривиальной ветви {LACAC,ENTFA,STRP1,STRPN,STAES} vs {BACSU,LISMO} правильного дерева, что немного хуже результатов, получаемых при построении дерева по белковым последовательностям (при использовании выравниваний белков некоторые методы давали и полную реконструкцию правильного дерева).
Построение и анализ дерева, содержащего паралоги
Я построил дерево гомологов белка CLPX_BACSU, найденных среди белков выбранных бактерий.
Для этого я сначала скачал последовательность CLPX_BACSU из Swissprot, скачал из директории P:\y13\term4\Proteomes протеомы моих бактерий и объединил их в один файл. Затем с помощью blastp нашёл гомологов этого белка в протеомах бактерий аналогично тому, как это делалось в предыдущем семестре. Далее я составил список идентификаторов найденных гомологов и с помощью программы seqret скачал их последовательности, которые затем выровнял с помощью Muscle. Полученное выравнивание импортировал в MEGA и реконструировал дерево методом Neighbor-Joining. Получившееся дерево с отмеченными на нём ортологами, паралогами и эволюционными событиями можно найти на рис. 2.
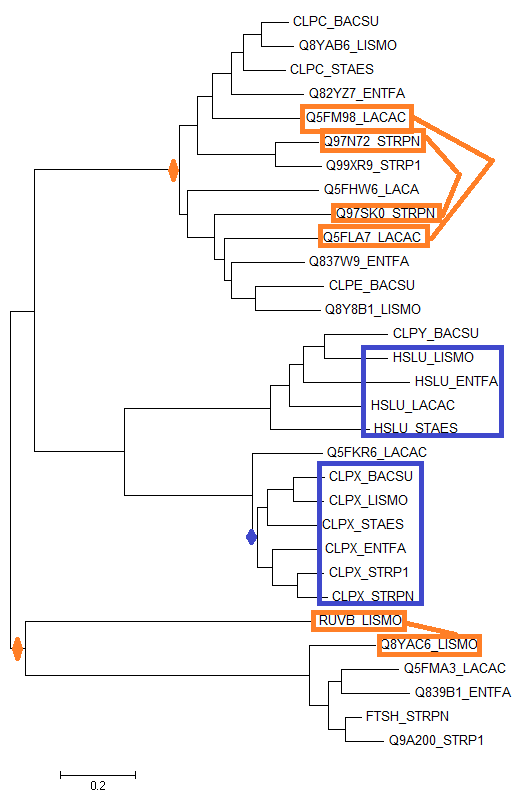
Рис. 1. Изображение филогенетического дерева гомологов белка CLPX_BACSU. Дерево получено с помощью метода Neighbor-Joining. Синими рамками выделены группы попарно ортологичных белков, оранжевыми - пары паралогов. Синими ромбиками отмечено разделение путей эволюции белков в результате видообразования, оранжевыми - дупликации гена.
Дата последнего обновления: 28.05.2014
Copyright © Дюгай Илья, 2013.