Учебный сайт Дюгая Ильи
Главная
Первый семестр
Второй семестр
Третий семестр
Четвертый семестр
Седьмой семестр
Ссылки
Об авторе
Визуализация электронной плотности
Выбор модели
В качестве модельной, была выбрана структура белка IRE1 H. sapiens (Inositol requiring enzyme), изучаемого мной в курсовой работе. Идентификатор PDB: 5HGI. Используя сервис EDS был получен файл, описывающий электронную плотность структуры, который можно получить по ссылке.
Разрешение структуры, указанное в PDB, составляет 2,58 Å. Дополнительную информацию о структуре можно найти в оригинальной статье.
Визуализация электронной плотности
Для визуализации электронной плотности белка 5HGI использовались следующие команды:
isomesh new_map, map, 1, name ca+c+n+o and !het, carve=2
isomesh new_map2, map, 2, name ca+c+n+o and !het, carve=2
isomesh new_map3, map, 3, name ca+c+n+o and !het, carve=2
Были использованны уровни подрезки 1, 2 и 3. Изображения на рисунках 1, 2 и 3 соответственно.
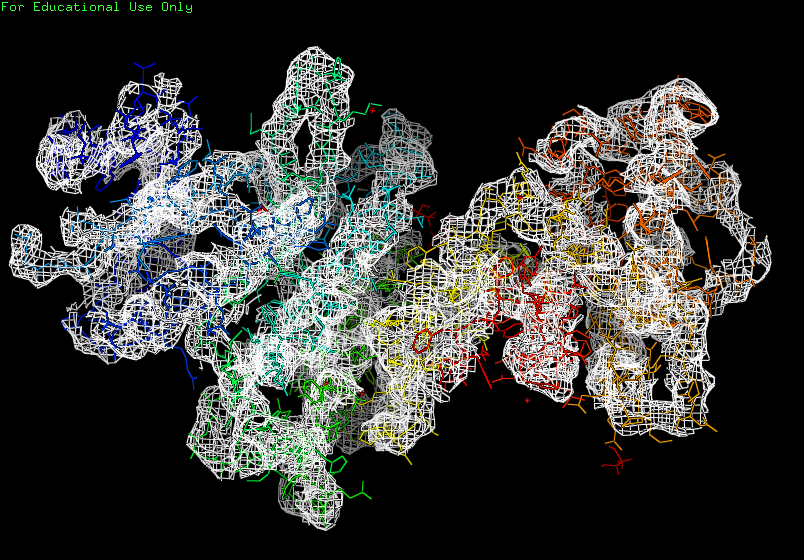
Рис. 1. Визуализация электронной плотности при σ=1.
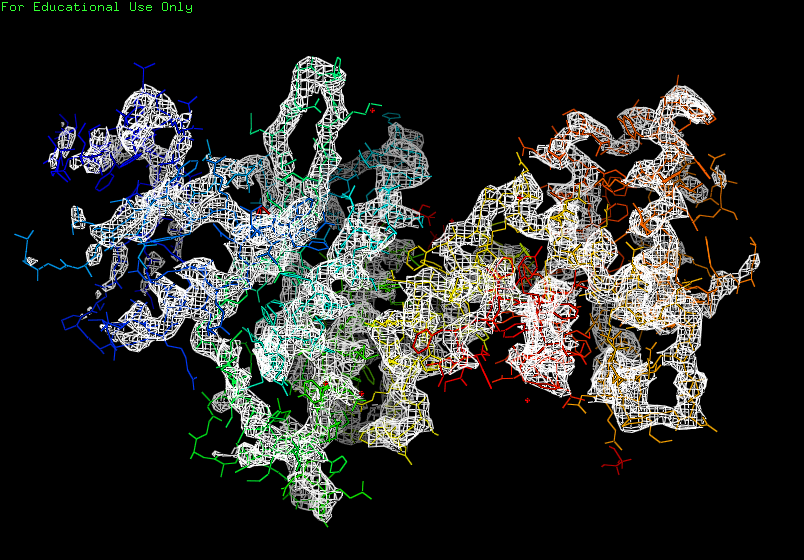
Рис. 2. Визуализация электронной плотности при σ=2.
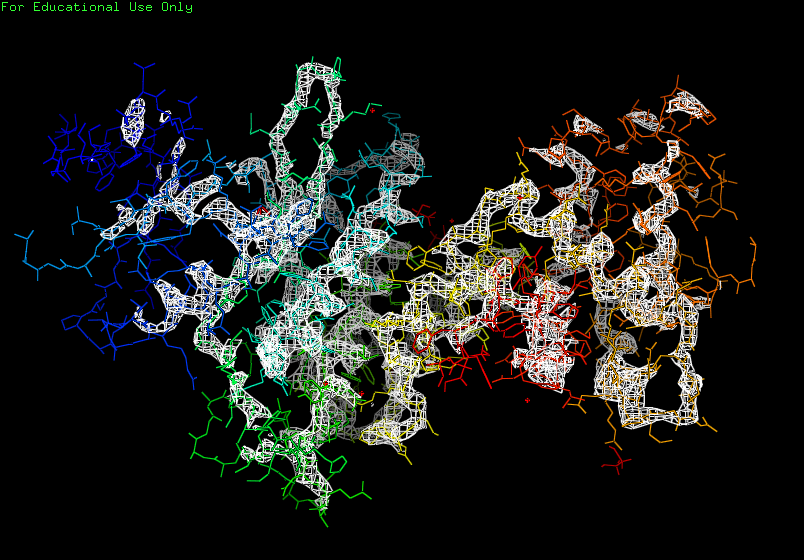
Рис. 3. Визуализация электронной плотности при σ=3.
На рисунке 4 представлена визуализация электронной плотности вокруг полипептидной цепи при σ=1.

Рис. 4. Визуализация электронной плотности вокруг полипептидной цепи при σ=1.
Для визуализации электронной плотности вокруг трех аминокислотных остатков был выбран участок с 824 по 826. Был использован уровень подрезки 0,5. Видно, что электронная плотность хорошо описывает структуру остатков.
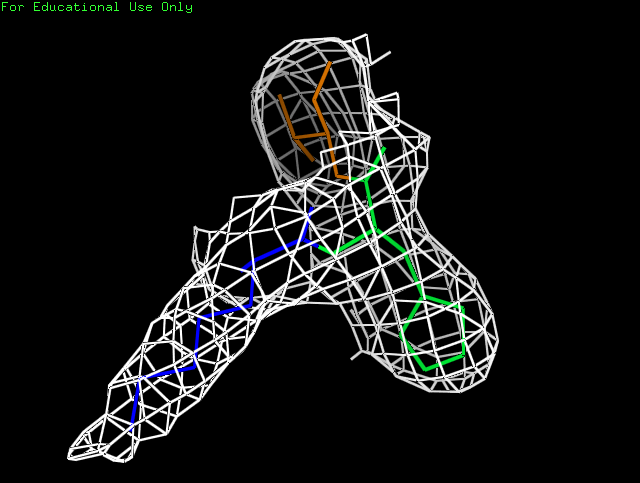
Рис. 5. Визуализация электронной плотности аминокислотных остатков при σ=3.
Copyright © Дюгай Илья, 2013.