Задание 1. Описание выданного контига и одного из генов на нем, для которого предсказан альтернативный сплайсинг
Исходный скеффолд с RefSeq ID: NW_013578456 принадлежит организму Lingula anatina.
Лингула (лат. Lingula) — род плеченогих из отряда лингулид класса лингулят, ведут роющий образ жизни. Это одни из древнейших плеченогих, доживший до наших дней. Известны с раннего ордовика (около 500 млн лет назад). Окаменелости ископаемых лингул найдены по всему миру, особенно много в Европе, Юго-Восточной Азии и Северной Америке.[1]

|
Kingdom: Animalia Phylum: Brachiopoda Class: Lingulata Order: Lingulida Family: Lingulidae Genus: Lingula |
Краткая информация о скеффолде:
- Размер: 446021 bp
- Число генов: 36
- Число кодирующих последовательностей: 40
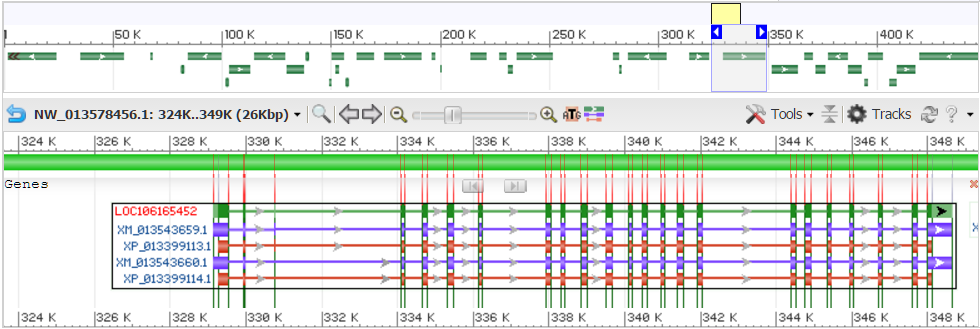
В данном скеффолде был найден ген, для которого предсказан альтернативный сплайсинг:
- Ген: LOC106165452
- Расположение: (329136..348633)
- Длина: 19,498
- Описание: кодирует EML2(echinoderm microtubule-associated protein-like 2)
На Рис. 1 можно увидеть различные варианты транскрибируемых с гена мРНК и соответсвующие им изоформы белков.
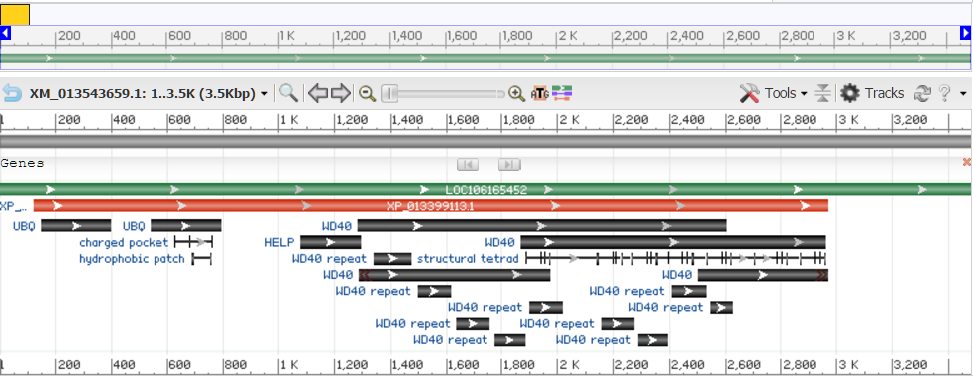
Также в геномном браузере были получены изображения мРНК и белка для одной из изоформ (X1)(всего их две). Они приведены на Рис. 2 и Рис. 3.

Задание 2. Предсказание генов и белок-кодирующих областей в выданном контиге
Выполнено с помощью сервера AUGUSTUS в режиме Prediction. Для сравнения необходимо было использовать в качестве модели наиболее близкий к изучаемому организм. Выбор оказался довольно сложным, так как не было представлено организмов надтипа Lophotrochozoa, поэтому я выбрала нематоду Caenorhabditis elegans (по современной классификации менее таксономически далекого от изучаемого объекта организма).
Параметры запуска: Prediction job ID: predSa7LzW89 AUGUSTUS parameter project identifier: caenorhabditis Genome file: Lingula_anatina.fasta User set UTR prediction: false Report genes on: both strands Alternative transcripts: medium (т.к. есть альтернативные транскрипты) Allowed gene structure: predict any number of (possibly partial) genes Ignore conflictes with other strand: false
На выходе был получен архив [tar.gz], содержащий шесть файлов различных расширений. (Табл. 1)
| Расширение файла | Содержание |
| *.aa | предсказание генов в виде fasta-последовательности белков |
| *.cdsexons | предсказанные экзоны в fasta-последовательности ДНК |
| *.codingseq | предсказание генов в виде fasta-последовательности кодирующих участков ДНК |
| *.gbrowse | трэк-файл предсказания для GBrowse |
| *.gff | предсказание генов в обычном [gff]-формате (General Feature Format) |
| *.gtf | предсказание генов в [gtf]-формате ([gff] 2-ой версии) |
Просмотреть файл с предсказанными генами [augustus.gff]
Предсказание AUGUSTUS плохо совпадает с аннотацией в GenBank. Так, при наличии 40 аннотированных генов в базе программа предсказывает 35 таковых по данным таблицы .gff. Неверно указаны координаты, не смогла выбранный найти ген с альтернативным сплайсингом. Возможно, причиной не очень качественного предсказания является выбор модельного объекта, слишком далекого по таксономии от изучаемого организма.