Задание 1. Предсказание вторичной структуры заданной тРНК.
Упражнение 1. Предсказание вторичной структуры тРНК путем поиска инвертированных повторов.
Программа einverted из пакета EMBOSS позволяет найти инвертированные участки в нуклеотидных последовательностях. Предсказание вторичной структуры тРНК проведено на примере PDB ID 1FFY. На вход программе необходимо подать фаста-последовательность РНК и задать ряд параметров: штраф за гэп, минимальный вес находок, бонус за пару, штраф за мисмэтч.
При подборе параметров, наилучшим результатом оказался следующий:
SEQUENCE: Score 73: 21/33 ( 63%) matches, 5 gaps
1 gggcttgtagctcaggtg-gtta--gagcgcacccctg 35
|||| || || | || | || | || ||||
73 cccggac-tcacctgaacttggtggctggagt-gggaa 38
Сравнение данных результатов с их описанием, полученным ранее с помощью find_pair, приведены в табл.1.
Упражнение 2. Предсказание вторичной структуры тРНК по алгоритму Зукера.
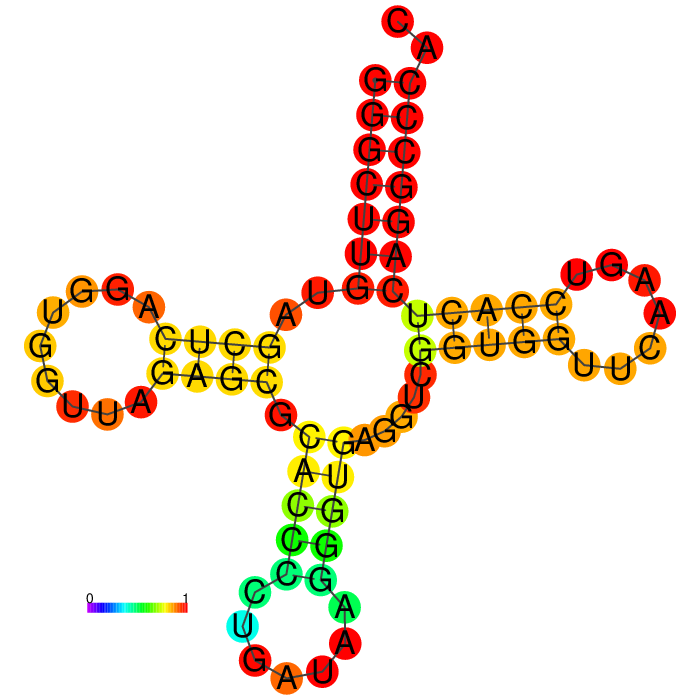
С помощью web-сервиса RNAfold был реализован алгоритм Зукера. Результаты предсказания также приведены в табл.1. Изображение полученной структуры приведено на рис.1.
| Участок структуры | Реальные позиции по результатам find_pair | Предсказано пар по einverted | Предсказано пар по алгоритму Зукера |
| Акцепторный стебель | 5'-1-7-3' 5'-66-72-3' 7 пар | 6/7 | 7/7 |
| T-стебель | 5'-49-53-3' 5'-61-65-3' 5 пар | 0/5 | 5/5 |
| D-стебель | 5'-10-13-3' 5'-22-25-3' 4 пары | 0/4 | 4/4 |
| Антикодоновый стебель | 5'-26-32-3' 5'-38-44-3' 7 пар | 6/7 | 5/7 |
| Общее число канонических пар нуклеотидов | 20 | 12 | 19 |
Из таблицы видно, что предсказания einverted далеки от реальных, а алгоритм Зукера может давать ошибки.
Задание 2 Поиск ДНК-белковых контактов в заданной структуре.
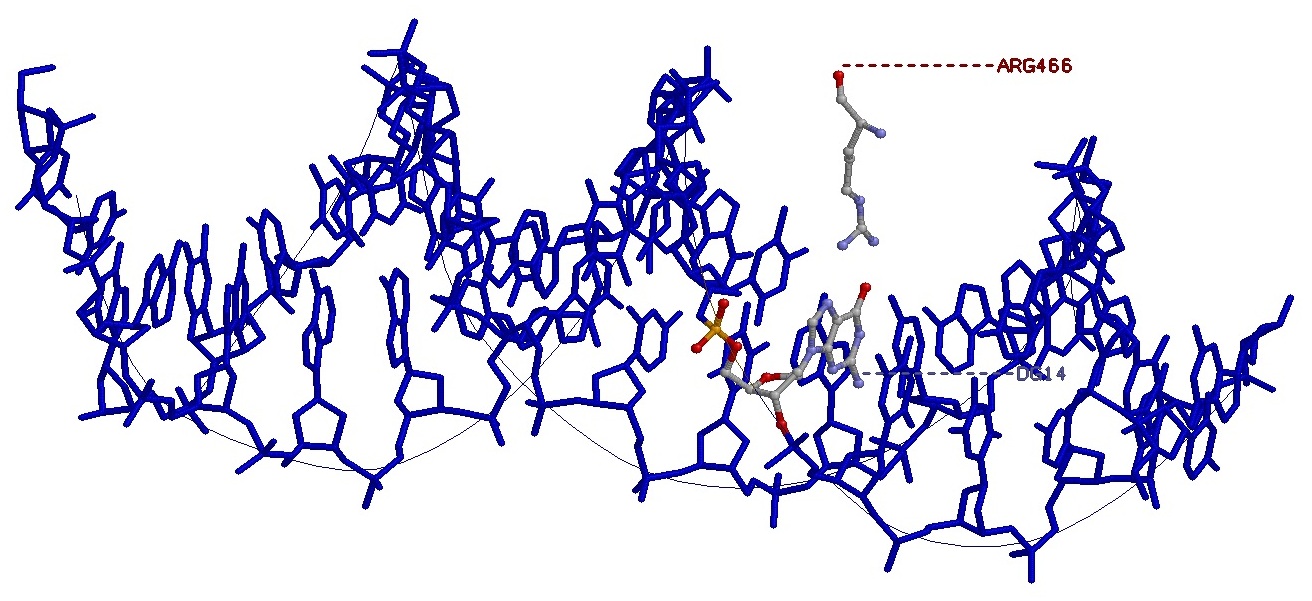
Исходный ДНК-белковый комплекс - PDB ID: 1R4O
Это задание выполнялось с помощью JMol. Вначале был создан скрипт, который в JMol последовательно дает изображение всей структуры, только ДНК в проволочной модели, той же модели но с выделенными шариками множеством атомов set1, затем set2 и set3. Обозначения:
set 1 - множество атомов кислорода 2'-дезоксирибозы
set 2- множество атомов кислорода в остатке фосфорной кислоты
set 3 - множество атомов азота в азотистых основаниях
Cсылка на скрипт: script1.txt
Описание ДНК-белковых контактов в заданной структуре. Пусть полярными считаются атомы кислорода и азота, а неполярными - атомы углерода, фосфора и серы. Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5A. Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5A.
Для обнаружения всех ДНК-белковых контактов был написан скрипт (смотреть).
Результаты представлены в табл.2:
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 1 | 2 | 3 |
| остатками фосфорной кислоты | 7 | 15 | 21 |
| остатками азотистых оснований со стороны большой бороздки | 2 | 1 | 3 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 0 | 0 |
Таким образом, можно сделать вывод о том, что в образовании ДНК-белковых контактов преимущественно участвуют неполярные атомы 2'-дезоксирибозы, полярные атомы фосфорной кислоты и неполярные атомы азотистых оснований со стороны большой бороздки. Атомы азотистых оснований со стороны малой бороздки вообще не принимают участия в образовании ДНК-белковых контактов.
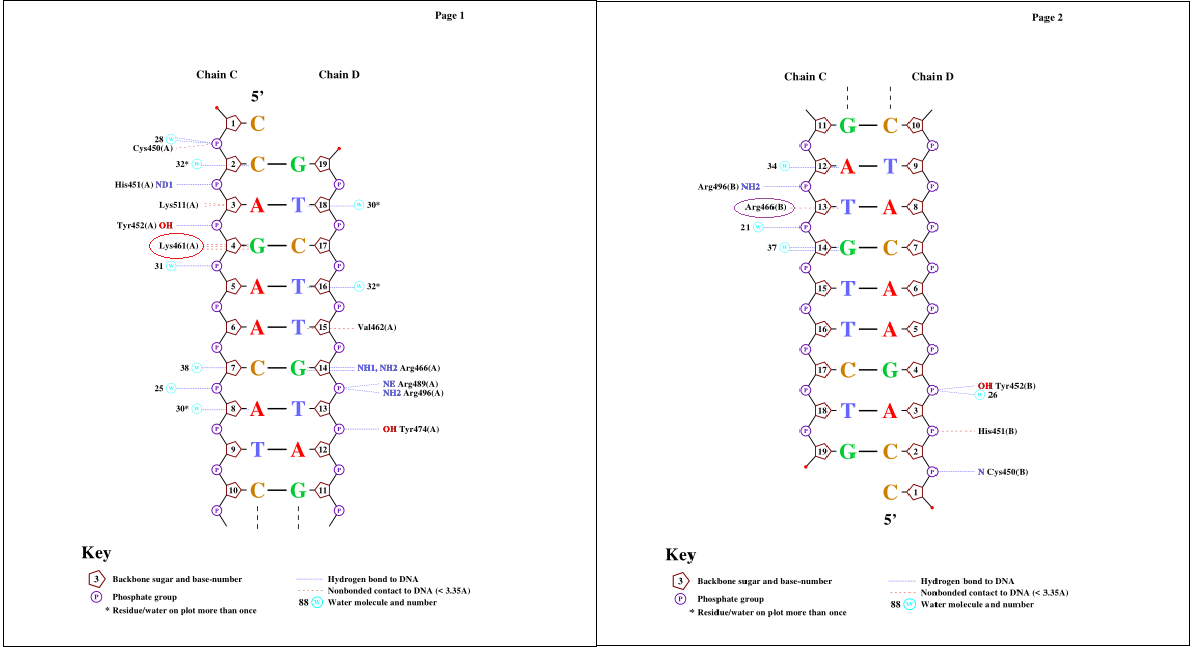
С помощью программы nucplot были найдены днк-белковые контакты. Результаты изображены на рис.2.
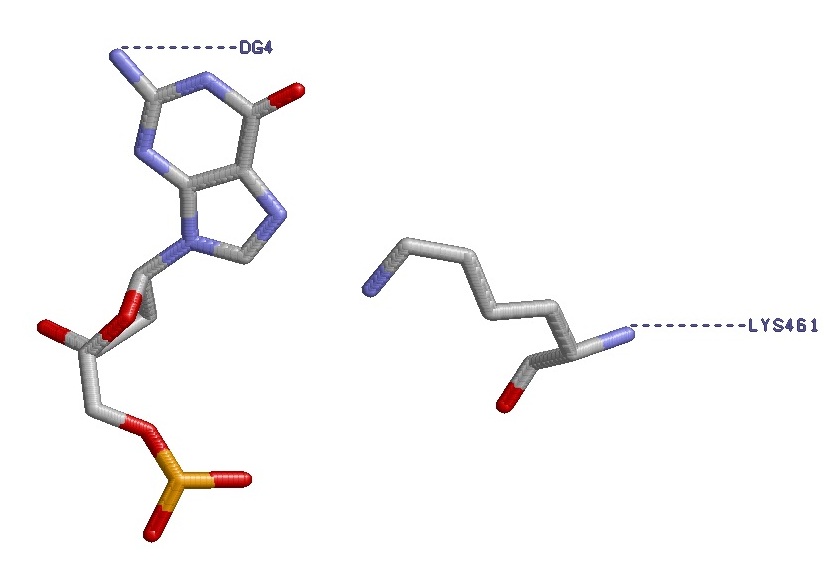
Аминокислотный остаток с наибольшим числом указанных на схеме контактов с ДНК - Lys461 рис.3
Я думаю, что наиболее важный аминокислотный остаток для распознавания ДНК - это Arg466:A, т.к. он связан с самим азотистым основанием, а не с сахарофосфатным остовом, причем двумя водородными связями рис.4.