Задание 1.
Требовалось определить таксономию и функцию последовательности, полученной в практикуме №6. Для этого был запущен blastn с данной последовательностью.
Параметры поиска: алгоритм blastn (Somewhat similar sequences), база данных Nucleotide collection (nr/nt), остальные параметры — по умолчанию.
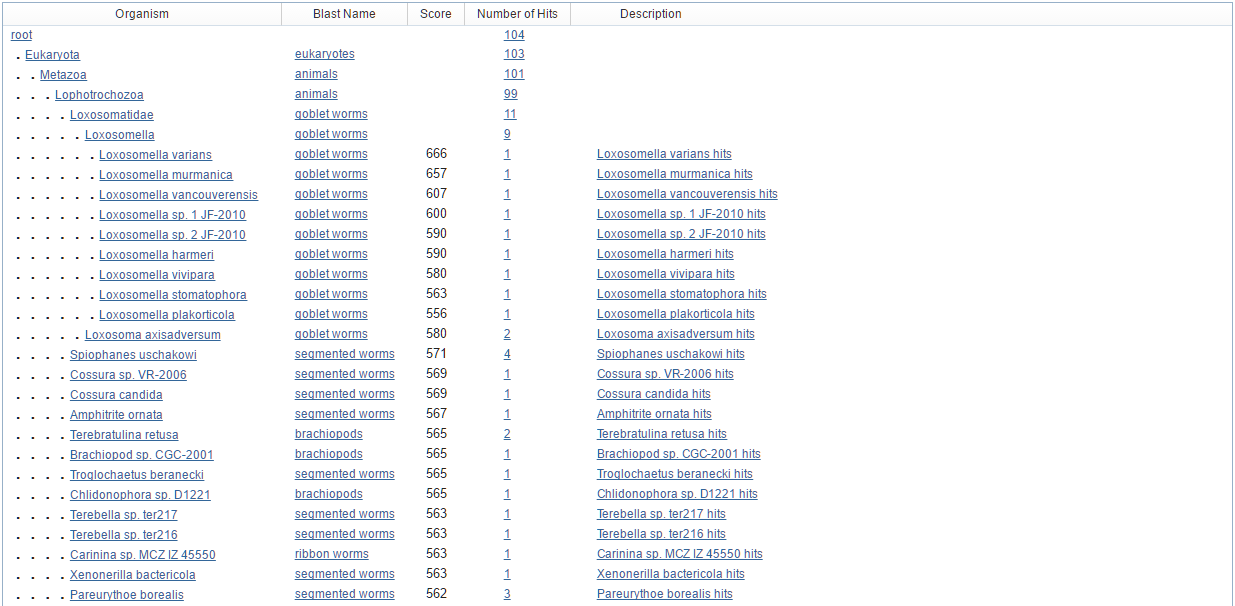
Результаты (рис 1):
E-value худшей находки - 4e-75, лучшей - 0.0. Identities - 92%, лучшей - 99%.
Искомая последовательность является частичной последовательностью гена, кодирующего 18s rRNA (18s рибособная РНК). Эта РНК является структурным компонентом малых субъединиц рибосом эукариот. Нуклеотидная последовательность гена 18s rRNA является частым объектом секвенирования, так как ее испольуют для построения филогенетических деревьев.

Blast также может отображать результаты как taxonomy report (рис.2). Так мы можем увидеть, выравнивания с генами каких видов были найдены и каков их вес. Нам интересны выравнивания с самыми большими значениями Score (рис.2).
Также blast составляет предполагаемое филогенетическое дерево для найденных последовательностей (рис.3).
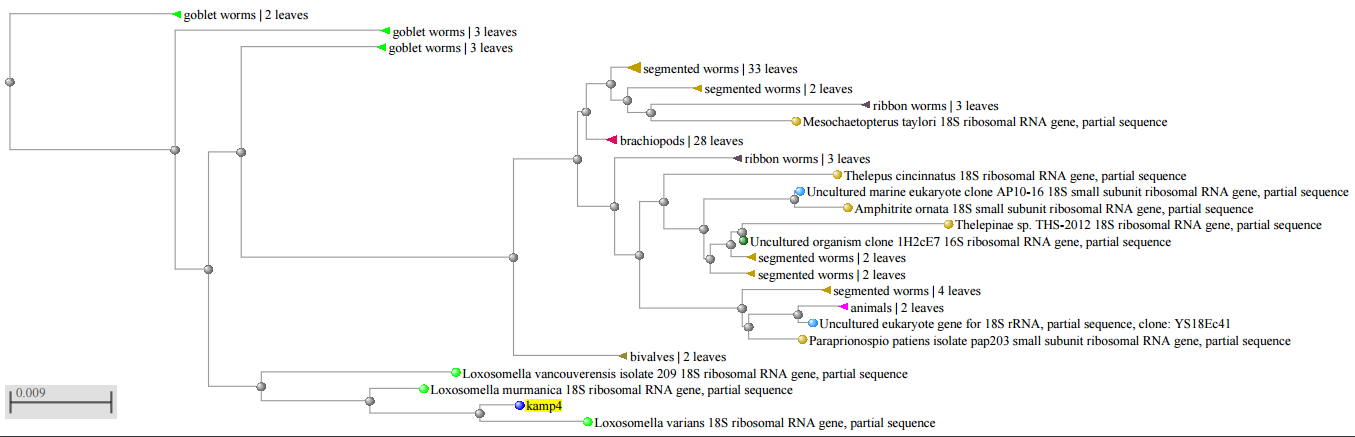
Из приведенных выше данных можно сказать, что нуклеотидная последовательность принадлежит организму из рода червей Loxosomella.
Для большей наглядности я взяла первые пять последоваельностей с лучшими параметрами E-value, Query cover и Ident и построила их множественное выравнивание в Jalview с помощью программы Muscle. (скачать проект jvp)

Проанализировав полученное выравнивание, я могу предположить, что с большой вероятностью данная последовательность может принадлежать организму Loxosomella varians.
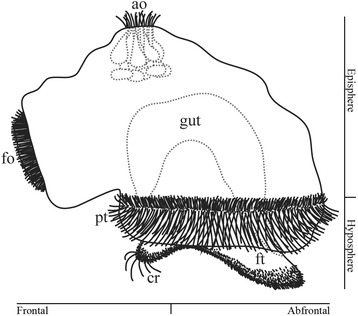
Данный организм относится к семейству Loxosomatidae, которое входит в группу внутрипорошицевых (= Kamptozoa). Представители этой группы микроскопические, обычно морские, сестонофаги. На рис. 5 можно увидеть представителя этого семейства — Loxosomella murmanica

|
|
|
рис. 5.1 3D реконструкция мышечной организации личинки Loxosomella murmanica. Сделано с помощью конфокальной микроскопии.
Источник: Merkel J, Lieb B, Wanninger A. Muscular anatomy of an entoproct creeping-type larva reveals extraordinary high complexity and potential shared characters with mollusks. BMC Evol Biol. 2015; 15: 130. doi: 10.1186/s12862-015-0394-1 [PMC] |
рис. 5.2 Схематическое строение личинки Loxosomella murmanica.
Источник: Merkel J, Lieb B, Wanninger A. Muscular anatomy of an entoproct creeping-type larva reveals extraordinary high complexity and potential shared characters with mollusks. BMC Evol Biol. 2015; 15: 130. doi: 10.1186/s12862-015-0394-1 [PMC] |
Конечная предполагаемая таксономия (до рода):
Задание 2.
Было проведено сравнение трех алгоритмов — blastn (somewhat similar sequences), discontigious megablast (more dissimilar sequences), megablast (highly similar sequences).
Так как для заданной мне последовательности не удавалось настроить поиск так, чтобы количество находок blastn было хотя бы меньше тысячи, была выбрана другая нуклеотидная последовательность (скачать FASTA) — ген, принадлежащий Schistosoma haematobium и кодирующий 18s рРНК.
| Алгоритм поиска | Database | Max target sequences | Expect treshold | Word size | Max matches in a query range | Match/Mismatch scores | Gap costs |
| blastn | Nucleotide collection (nr/nt) | 100 | 10 | 11 | 0 | 2;-3 | 5;2 |
| discontigious megablast | Nucleotide collection (nr/nt) | 100 | 10 | 11 | 0 | 2;-3 | 5;2 |
| megablast | Nucleotide collection (nr/nt) | 100 | 10 | 28 | 0 | 1;-2 | linear |
С приведёнными выше параметрами была запущена работа трёх алгоритмов, при этом область поиска была ограничена таким образом, что поиск проводился в пределах семейства Schistosomatidae, но исключался род Schistosoma. Результаты поиска можно увидеть в таблице 2.
| Алгоритм | Число находок | E-value худшей находки | Ident худшей находки | Query cover худшей находки |
| megablast | 34 | 5e-74 | 85% | 13% |
| discontiguous megablast | 34 | 3e-79 | 85% | 13% |
| blastn | 52 | 5.6 | 100% | 0% |
Алгоритм blastn выдал больше результатов, чем другие два, но все эти находки имеют очень высокий e-value и очень низкое покрытие (рис 6).

Проведенное сравнение позволяет сделать следующие выводы о специфике работы алгоритмов поиска:
- megablast находит самые достоверные и наиболее близкие гомологи
- discontigious megablast хорошо работает в плане поиска гомологов, которые прежде уже дивергировали, и имеют как схожие, так и различающиеся участки
- blastn выдает наиболее широкий спектр последовательностей, которые могут и не быть гомологами исходной или являться очень далекими гомологами.
Задание 3.
В этом задании нужно было проверить наличие гомологов трех белков в заданном геноме Amoeboaphelidium protococarum. Этот организм относится к афелидам, которые родственны грибам протистов из группы опистоконт. Все описанные виды являются внутриклеточными паразитами.
Наличие гомологов устанавливалось с помощью локального BLAST (версия BLAST+) на kodomo. Были выбраны три белка CISY_HUMAN, HSP7C_HUMAN, PABP2_HUMAN и объединены в один файл (смотреть FASTA) для более быстрой работы.
Сначала была создана локальная база данных на основе генома Amoeboaphelidium protococarum, записанного в файле X5.fasta, следующим образом:
makeblastdb -in X5.fasta -dbtype nucl
Запуск поиска с помощью программы blastn:
tblastn -query inprot.fasta -db X5.fasta -out blast.out -outfmt 7
Результаты работы программы обобщены в таблице 3, также можно скачать файл, полученный на выходе.
| Название | Функция | Число находок | Лучшая находка | Ident лучшей | Покрытие лучшей | E-value лучшей |
| Citrate synthase, mitochondrial (CISY_HUMAN) | Белок митохондриальной цитрат-синтазы, принимает участие в метаболизме углеводов, а именно — в цикле трикарбоновых кислот на стадии получения изоцитрата из оксалоацетата. | 6 | scaffold-693 | 69% | 94% | 2e-180 |
| Heat shock cognate 71 kDa protein (HSP7C_HUMAN) | Белок теплового шока. Репрессор активации транскрипции. Шаперон. Является компонентом PRP19-CDC5L комплекса, который формирует внутренню часть сплайсосомы. Этот белок необходим для активации сплайсинга пре-мРНК. Может выполнять структурную функцию в сборке сплайсосомы, поскольку контактирует со всем остальными ее компонентами. Связывает бактериальные липополисахариды и опосредует ЛПС-идуцированную воспалительную реакцию. | 22 | scaffold-199 | 78% | 94% | 0.0 |
| Polyadenylate-binding protein 2 (PABP2_HUMAN) | Участвует в присоединении поли(А)-хвоста к 3'-концу пре-МРНК. Также присутствует на разных стадиях метаболизма мРНК. Полиаденилирование защищает мРНК от ферментативного разрушения в цитоплазме, способствует терминации транскрипции, участвует в экспорте мРНК из ядра и трансляции. | 28 | scaffold-100 | 63% | 31% | 2e-28 |
Параметры лучших находок для первых двух белков довольно неплохие, что позволяет предположить, что в геноме исследуемого организма закодированы белки, гомологичные данным. У лучшей находки третьего белка E-value достаточно большой, что позволяет предположить, что у исследуемого организма нету белков, гомологичных данному.
Задание 4.
Для выполнения задания был выбран из генома Amoeboaphelidium protococarum скэффолд scaffold-698. Предварительно была получена информация о длинах контигов:
infoseq X5.fasta -only -name -length
А затем извлечена последовательность подходящего по длине scaffold-698:
seqret X5.fasta:scaffold-698 -out scaffold-698.fasta
По этой последовательности был запущен алгоритм blastx. Результаты представлены на Рис. 7.

Таким образом, удалось обнаружить ген, кодирующий субъединицу гамма эукариотического трансляционного фактора инициации 2 (eukaryotic translation initiation factor 2 subunit gamma (eIF2-γ)). Эта субъединица совместно с субъединицами eIF2-α и eIF2-β образует эукариотический трансляционный фактор инициации 2 (eIF2), который стабилизирует формирование функциональной рибосомы рядом со старт-кодоном. Он связывается с гуанозин трифосфатом (GTP) и отвечает за доставку заряженной метионином инициирующей тРНК к P-сайту пред-инициирующего комплекса. Как только инициирующая тРНК свяжется со старт-кодоном AUG в P-сайте, так трансляционный фактор инициации 2 гидролизирует ГТФ (GTP) до ГДФ (GDP) и сам диссоциирует. Это позволяет большой субъединице рибосомы связаться и начать элонгацию (удлинение полипептидной цепи). Лучшая находка представлена на Рис. 8