Я искала белки, сходные с моим белком из первого семестра: CRISPR-ассоциированного белка Csn1, всятого из Mycoplasma gallisepticum S6. Поиск только по базе данных SwissProt не дал необходимого разнообразия значений e-value (чтобы увидеть этот запрос загрузите в BLAST файл GHJD7RTG015_search_strategy.asn). Поэтому в дальнейших запросах я не ограничивала базу данных. Для выполнения задания я пользовалась белками, данные которых есть в таблице 1 (это автоматическая таблица, полученная при поиске в BLAST - Hit table. Правда, я убрала из нее первый столбец - query id, поскольку он везде одинаков: gi|564750085|gb|AHB99924.1| и пронумеровала находки).
Некоторые столбцы, вероятно, нуждаются в пояснении. Positives % - число позиций с положительным весом, mismatches - число замен аминокислот (не инделей, а именно замен), q. start и q.end - координаты выравнивания в изначальном белке, s. start и s. end - в сравниваемых белках.
ID сеанса поиска, для которого приведена эта таблица - GGYDV109015 (чтобы посмотреть параметры загрузите в BLAST следующий файл). Поиск проводился при следующих параметрах:
| № | subject ids | % identity | % positives | alignment length | mismatches | gap opens | q. start | q. end | s. start | s. end | e-value | bit score |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | gi|652844792|ref|WP_027123033.1| | 41,29 | 60,65 | 310 | 143 | 6 | 8 | 291 | 6 | 302 | 8,46E-058 | 229 |
| 2 | gi|491504038|ref|WP_005361708.1|;gi|149736675|gb|EDM52561.1| | 26,708 | 46,48 | 966 | 522 | 37 | 12 | 931 | 4 | 829 | 9,82E-048 | 197 |
| 3.1 | gi|1181744525|ref|WP_084829479.1|;gi|585225656|gb|EWM57058.1| | 30,751 | 52,3 | 413 | 234 | 14 | 536 | 936 | 466 | 838 | 1,59E-033 | 152 |
| 3.2 | gi|1181744525|ref|WP_084829479.1|;gi|585225656|gb|EWM57058.1| | 26,91 | 43,19 | 301 | 164 | 9 | 10 | 279 | 3 | 278 | 4,46E-013 | 85,5 |
| 4 | gi|919262384|ref|WP_052796772.1|;gi|950687566|gb|KRS61385.1|; gi|950715890|gb|KRS87309.1|;gi|950719788|gb|KRS90916.1| | 32,132 | 48,05 | 333 | 167 | 12 | 538 | 852 | 438 | 729 | 1,41E-022 | 116 |
| 5 | gi|517844287|ref|WP_019014495.1| | 23,512 | 46,13 | 336 | 209 | 13 | 550 | 874 | 475 | 773 | 3,39E-011 | 79,3 |
| 6 | gi|550259504|ref|WP_022599516.1|;gi|548785101|gb|ERM89468.1| | 25,284 | 44,03 | 352 | 185 | 16 | 545 | 862 | 696 | 1003 | 4,04E-005 | 59,7 |
| 7 | gi|488758052|ref|WP_002681289.1|;gi|42525843|ref|NP_970941.1|; gi|41815893|gb|AAS10822.1|;gi|448955571|gb|EMB36337.1|; gi|448960365|gb|EMB41078.1| | 27,187 | 41,56 | 320 | 187 | 13 | 534 | 842 | 744 | 1028 | 0,005 | 52,8 |
| 8 | gi|1183711884|ref|WP_084938764.1|;gi|1181878277|gb|ORO40105.1| | 24,415 | 41,14 | 299 | 175 | 12 | 549 | 833 | 778 | 1039 | 1,1 | 45,1 |
| 9 | gi|496371126|ref|WP_009080116.1|;gi|400372753|gb|EJP25691.1| | 48,78 | 63,41 | 41 | 16 | 1 | 13 | 48 | 8 | 48 | 1,4 | 44,7 |
Таблица 1.Hit table (автоматическая таблица).
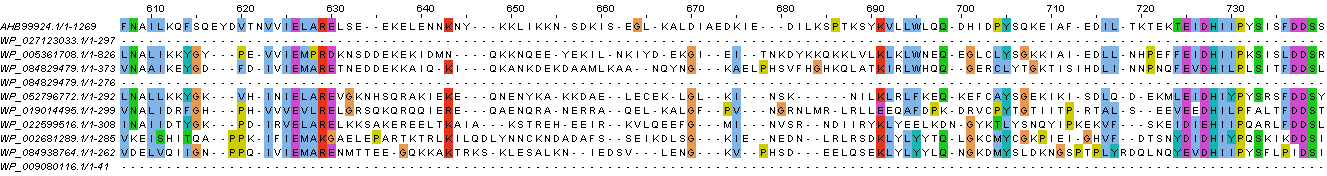
Рисунок 1. Множественное выравнивание.
Можно заметить, что третья последовательность - с ID WP_084829479.1 - имеет 2 выравнивания с разными координатами выравнивыемых фрагментов. То есть программа нашла 2 изначальных шестибуквенных соответствия, вокруг которых начала выравнивание.
На рисунках 2-17 представлены блоки - отрезки выравнивания нескольких последовательностей с достаточным числом абсолютно консервативных позиций (или функционально консервативных - как на рис. 11). Подпись содержит номер рисунка и последовательности, входящие в блок (причем последовательность изначального белка - 0, дальше по порядку, см таблицу 1). Блоки также выделены рамкой. Рамка может содержать строку символов гэпа, однако, очевидно, они не входят в данный блок (я просто решила не делить рамку из-за столь очевидного случая). Толщина рамки большая, если в блок не входят какие-то сосдение последовательности (просто чтобы зрительно было проще отличить, где блок, в где нет). Иногда на одном рисунке можно выделить несколько блоков (как на рис. 4, 10 и 17).
 |  | .png) |  |  |  |
| Рисунок 2. 1 | Рисунок 3. 1, 2, 3.2 | Рисунок 4. 1 и 1, 2, 3.2 | Рисунок 5. 1, 3.2, 9 | Рисунок 6. 1, 2, 3.2, 9 | Рисунок 7. 2, 3.1, 4 |
 |  |  |  |  |  |
| Рисунок 8. 2, 3.1, 4, 5, 8. | Рисунок 9. 2-3.1, 4-8. | Рисунок 10. 2, 4, 7-8 и 7-8. | Рисунок 11. 2, 3.1, 4-5. | Рисунок 12. 2, 3.1, 4, 6-8. | Рисунок 13. 2, 3.1, 4-7. |
 |  |  |  | ||
| Рисунок 14. 2, 3.1, 4-7. | Рисунок 15. 2, 3.1, 4, 6, 8. | Рисунок 16. 5. | Рисунок 17. 1, 3.1-4, 8 и 1-8. |
Задача состояла в том, чтобы определить, гомологичны ли белки или хотя бы их части. Сначала хотелось бы отметить, что все они имеют одинаковое название (см. таблицу 2).
Дальше пойдем по порядку.
Понятно, что чем меньше e-value и нейтральный вес, тем более вероятна гомология. Поэтому первые 6 последовательностей, обладающие e-value меньше 1Е-05 и довольно высоким весом, скорее всего гомологичны. Но по этим параметрам локального выравнивания сложно понять, гомологична ли вся последоватлеьность или лишь ее часть. Для этого нужен параметр coverage.
Если посмотреть на него, то видно, что лишь у второй последовательности он достаточно высок (то есть можно говорить о гомологии именно последовательности, а не домена).
Чтобы сделать вывод про остальные последовательности, полезно посмотреть на третью (3.1 и 3.2). Третья последовательность, к которой BLAST построил 2 выравнивания, очевидна гомологична лишь этими двумя доменами (иначе было бы одно выравнивание).
Причем если посмотреть на координаты выравнивания в изначальной последовательности (таблица 1), то видно, что все остальные последовательности тоже гомологичны примерно этим же доменам (фрагменту примерно от 530 до 800-900 соответствуют выровненные фрагменты п-тей 3.1, 4, 5, 6, 7, 8; от примерно 10 до 200 п-ти 1, 3.2, начало 9-ой).
Потому логично сделать вывод о том, что у всех последовательностей кроме 2 гомологичны лишь домены (домен 1: ≈ 500 - ≈ 850; домен 2: ≈ 10 - ≈ 300).
Интересное наблюдение. Последняя (9-я) находка обладает самым низким весом и самым высоким e-value, однако при этом обладает самым высоким процентом идентичности. Объясняется это тем, что у нее гомологичен малая часть домена 1, и покрытие составляет всего около 3%, однако на этом маленьком фрагменте консервативность последовательности очень велика.
| № | ID | Name | Coverage | % Identity | E-value | Homology | |||
|---|---|---|---|---|---|---|---|---|---|
| S. start | S. end | AlignLength/SeqLength | % | ||||||
| 1 | WP_027123033.1 | type II CRISPR RNA-guided endonuclease Cas9 [Mycoplasma spumans] | 6 | 302 | 297/1076 | 27,6 | 41,29 | 8,46E-058 | да, домен 1 |
| 2 | WP_005361708.1 | type II CRISPR RNA-guided endonuclease Cas9 [Eubacterium ventriosum] | 4 | 829 | 826/1107 | 74,62 | 26,708 | 9,82E-048 | да, п-ть |
| 3.1 | WP_084829479.1 | type II CRISPR RNA-guided endonuclease Cas9 [Streptococcus thermophilus] | 466 | 838 | 373/1121 | 33,27 | 30,751 | 1,59E-033 | да, домен 2 |
| 3.2 | WP_084829479.1 | type II CRISPR RNA-guided endonuclease Cas9 [Streptococcus thermophilus] | 3 | 278 | 276/1121 | 24,62 | 26,91 | 4,46E-013 | да, домен 1 |
| 4 | WP_052796772.1 | MULTISPECIES: type II CRISPR RNA-guided endonuclease Cas9 [Campylobacter] | 438 | 729 | 292/984 | 29,67 | 32,132 | 1,41E-022 | да, домен 2 |
| 5 | WP_019014495.1 | type II CRISPR RNA-guided endonuclease Cas9 [Elioraea tepidiphila] | 475 | 773 | 299/1034 | 28,92 | 23,512 | 3,39E-011 | да, домен 2 |
| 6 | WP_022599516.1 | type II CRISPR RNA-guided endonuclease Cas9 [Coprobacter fastidiosus] | 696 | 1003 | 308/1432 | 21,51 | 25,284 | 4,04E-005 | да, домен 2 |
| 7 | WP_002681289.1 | type II CRISPR RNA-guided endonuclease Cas9 [Treponema denticola] | 744 | 1028 | 285/1395 | 20,43 | 27,187 | 0,005 | да, домен 2 |
| 8 | WP_084938764.1 | type II CRISPR RNA-guided endonuclease Cas9 [Streptococcus oralis] | 778 | 1039 | 262/1420 | 18,45 | 24,415 | 1,1 | да, домен 2 |
| 9 | WP_009080116.1 | type II CRISPR RNA-guided endonuclease Cas9 [Peptostreptococcaceae bacterium AS15] | 8 | 48 | 41/1391 | 2,95 | 48,78 | 1,4 | да, часть домена 1 |
Таблица 2. Параметры и вывод.
Скачать проект можно по ссылке.
| ID | Query ID | Description | Length | Organism |
|---|---|---|---|---|
| V8N5V2_OPHHA (V8N5V2) | Query_197297 | белок главного комплекса гистосовместимости, класс I-related | 474 | Ophiophagus hannah (королевская кобра) |
| A0A0F8BNJ7_LARCR (A0A0F8BNJ7) | Query_197295 | AAA домен-содрержащий белок 2 из семейства ATP-аз | 2081 | Larimichthys crocea (желтый горбыль, отр. окунеобразные) |
Таблица 3. Белки для второго задания.
Почему именно ти белки? Я хотела найти инвертированную последовательность, на соответствующей странице в википедии нашла название каких-то групп белков, у которых встречаются повторы (VNTR) и вбила их в Pfam, где по доменной организации выбрала интересный случай. Далее я воспользоваласась BLAST, чтобы выровнять эти 2 последовательности. Ниже приведены параметры, при которых производилось выравнивание.
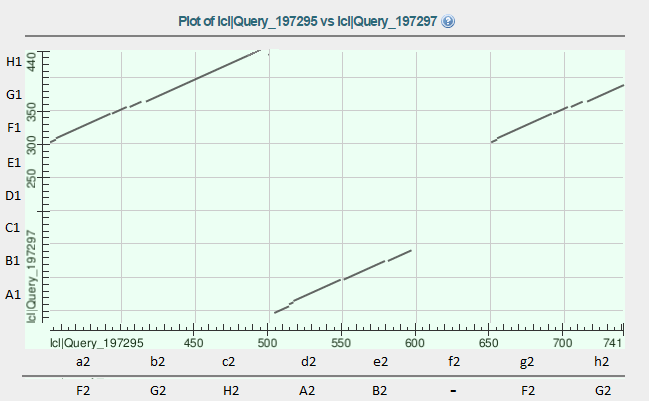
Рисунок 18. Карта выравнивания.
Каждый участок последователньостей я обозначила буквой и цифрой. Вертикалной последовательности соответствуют большие буквы и цифра 1, горизонтальной - маленькие и цифра 2. После этого горизонтальная последовательность была размечена в соответствии с гомологией участков (вторая строка подписей - это гомологичные участки, причем гомологичны те участки, которые обозначенные одной большой буквой). Например, первый и предпоследний участки горизонтальной последовательности соответствуют одному и тому же участку вертикальной, что значит, что все они примерно одинаковы. То есть:
Итого получилось 2 последовательности:
| A1 | B1 | C1 | D1 | E1 | F1 | G1 | H1 |
| F2 | G2 | H2 | A2 | B2 | - | F2 | G2 |
Что можно про них сказать?
Во-первых, начало второй последовательности соответствует концу первой, а началу первой соответствует продолжение второй. То есть 2 домена поменялись местами (то есть произошла транслокация).
Во-вторых, видна дупликация куска FG во второй последовательности, причем интересно, что в двух белках совпадают фрагменты FGH, однако дупликация не включила в себя H.
НАЗАД ➜ |
| © <Рюмина Екатерина>, 2017 |