Выравнивание геномов
Задание 1. Выбор геномов
Выбранные мной в итоге бактерии с полностью секвенированным геномом, состоящем из одной хромосомы - Escherichia coli str. K-12 substr. MG1655 (по горизонтали), Escherichia coli IAI39 (по вертикали на рис. 1)
и Escherichia coli DH1 (по вертикали на рис. 2).
Данные геномы были отобрано потому, что они имеют большие гомологичные участки и в них присутствуют крупные геномные перестановки. Эти параметры проверялись с помощью выравниванием двух последовательностей blast (потому что NPG-explorer работает куда медленнее, к тому же требует подготовительных этапов). Порог E-value про поиске - 0,01 (впрочем, оно все равно было везде равно нулю).
Выравнивание первых двух последовательностей проводился при разной длине слов (28, 64, 128 и 256). Видно, что при максимальной длине (256) глобальные перестройки не пропадают, а вот уровень шума уменьшается. Поэтому при выравнивании вторых двух последовательностей, я сразу выставляла длину слова 256.
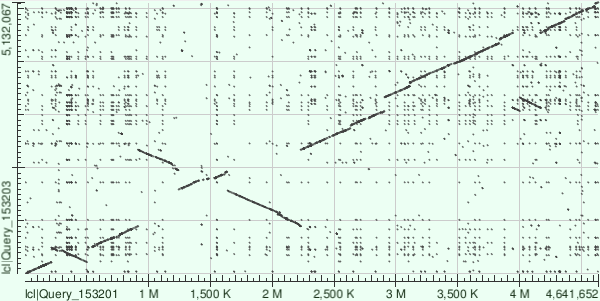
Рисунок 1а. E.coli K12, E.coli IAI39 (длина слова - 28).
|
.png)
Рисунок 1b. E.coli K12, E.coli IAI39 (длина слова - 64).
|
.png)
Рисунок 1c. E.coli K12, E.coli IAI39 (длина слова - 256).
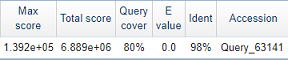
Рисунок 1с'. Информация
|
.png)
Рисунок 2. E.coli K12, E.coli DH1 (длина слова - 256).
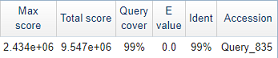
Рисунок 2'. Информация
|

Рисунок 3. E.coli IAI39, E.coli DH1 (длина слова - 256).

Рисунок 3'. Информация
|
Как видно по рисунку 1с, в геноме E.coli IAI39 присутствует 5 инверсий и вставка (около 2,9М по К-12 - горизонтали). На рисунке 1с' приведена краткая информация по этому выравниванию. По рисунку 2 видно, что в E.coli DH1 гомологичен чуть ли не весь геном (весь геном DH1 и 99% генома K-12), однако последовательности начинаются с разных мест.
Задание 2. Идентичность и покрытие.
Значение этих параметров, полученные с помощью blast2seq можно увидеть на рисунках 1c', 2' и 3'. Для получения дальнейших данных была использована пакет NPG-explorer (используемый файл):
| s-блоки | h-блоки | r-блоки | все не минорные,
из >2 фрагментов |
|---|
| Идентичность блоков | min=0.858
median=0.9749
avg=0.9686
max=1 | min=0.9705
median=1
avg=0.9998
max=1 | min=0.85
median=0.9259
avg=0.9323
max=1 | min=0.85
median=0.9787
avg=0.969
max=1 |
| Идентичность соединенных блоков | 0.975811 | 0.999905 | 0.952382 | 0.977783 |
Покрытие (длина и покрытие фрагментов,
тотальная длина и покрытие блоков) | 11920384 (82.75%)
3975792 (73.71%) | 951361 (6.6%)
475681 (8.81%) | 679459 (4.71%)
105561 (1.95%) | 13551204 (94.07%)
4557034 (84.48%) |
| Покрытие всех блоков, кроме уникальных | 4565154 (84,64%) |
Таблица 1. Покрытие, совпадения - NPG-explorer
Задание 3.
Синтеничные участки
используемые файлы: blocks.gbi, blocks.blocks
Всего NPG-explorer нашел 31 g-блок, причем все они состоят из 3 фрагментов. Средняя длина фрагментов этих блоков варьирует от 102.33 п. н. до 842932.33 п. н.
E.coli DH1, E.coli K-12 и E.coli IAI39 содержат по 38 g-блоков (что логично, g-блок по определению присутствует во всех геномах). При этом есть 12 g-блоков, присутствующих у всех трех штаммов на одинаковом месте и в одинаковой ориентации, - консервативных g-блоков (или 14, если считать два g-блока, в которых в одной из последовательностей данный блок инвертирован).
I-блоков, которыми перемежаются g-блоки, найдено 63: 1 i3x1, 30 i2x (со средней длиной от 1 до 31544.50 п. н.) и 32 i1x (со средней длиной от 2 до 46039 п. н.).
На рисунке 4 можно видеть примеры просто инверсии (блок g3x7437) и инверсии с транслокацией (блок g3x219182). На рисунке 5 можно увидеть пример консервативных блоков (блок g3x1611 и блок g3x59409) и опять пример транслокации с инверсией (причем все в том же штамме - блок g3x119).

Рисунок 4. Примеры перестроек

Рисунок 5. Примеры консервативных блоков
Филогения
Используя файл, полученный с помощью пакета NPG-explorer, а также программу просмотра деревьев FigTree, я получила рис. 4, отражающее родство штаммов IAI39, K-12 и DH1 (числами обозначены относительные длины ветвей).

Рисунок 4. Филогенетическое дерево.
Инверсии
По направленным глобальным блокам (направленность обозначается символами > и <) каждого штамма, полученным с помощью NPG-explorer, можно было посмотреть наличие инверсии g-блоков.
Для удобства, я перенесла данные о последовательностях блоков в таблицу ods. С помощью нее были посчитаны инверсии g-блоков - число случаев, когда в каком-то одном штамме блок был направлен не в ту сторону, в которую направлен этот блок в двух других штаммах.
Такой подсчет показал, что инверсии вообще присутствуют только в E.coli IAI39, в которой присутствуют 2 инверсии без перестановки и 14 инверсий с перестановками, причем во всех случаюх, кроме двух (которые в файле помечены комментарием "перестановка и в др"), перестановка данного блока тоже наблюдается только в IAI39. То есть в большинстве случаев в IAI39 произошла одновременная транслокация и инверсия g-блока, в то время как в двух другх штаммах происходила только транслокация, да и то гораздо реже.
Это наблюдение хорошо согласуется с построенным деревом, в котором E.coli IAI39 отстоит от двух других штаммов гораздо дальше, чем они друг от друга.
Делеции, вставки
G-блоки по определению есть во всех последовательностях, в то время как перемежающие их i-блоки - нет. Потому я рассматривала именно их (как и в предыдущем случае, перенеся в таблицу). Результаты все так же подтверждают большое сходство штаммов DH1 и K-12, в отличие от IAI39:
у K-12 и DH1 все i-блоки совпадают (хотя три из них имеют разное положение в этих штаммах), а с IAI39 не совпадает ни один. В ситуации такого полного совпадения сложно говорить о вставке или, наоборот, делеции, тем более, что во всех штаммах эти блоки в основном состоят из повторов.
НАЗАД ➜ |
| | © <Рюмина Екатерина>, 2017 |
|

.png)
.png)

.png)






