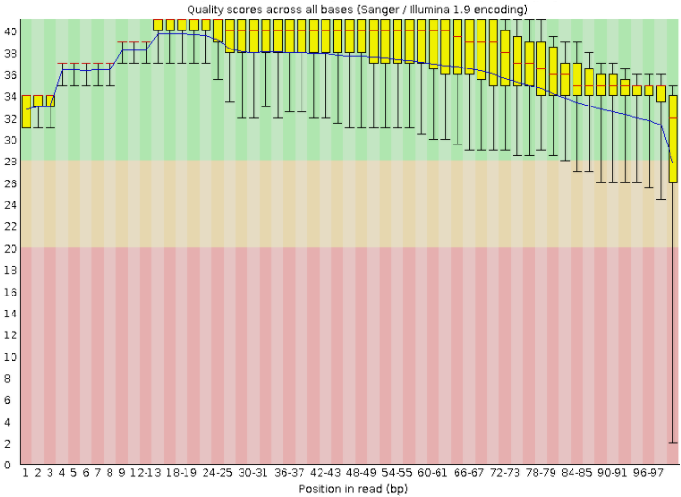
Рисунок 1. До чистки
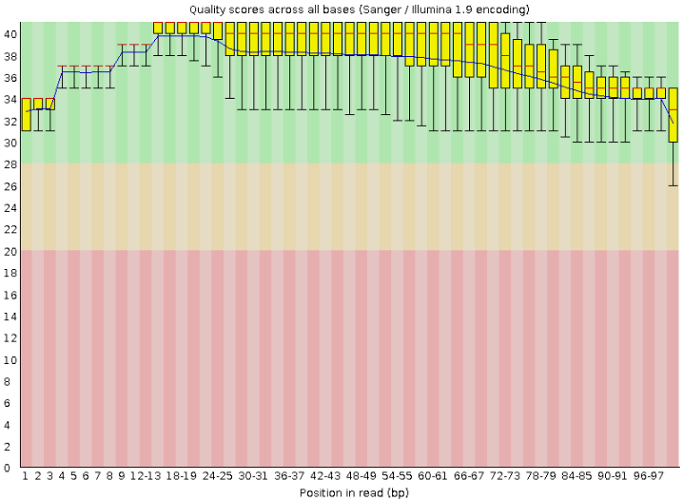
Рисунок 2. После чистки
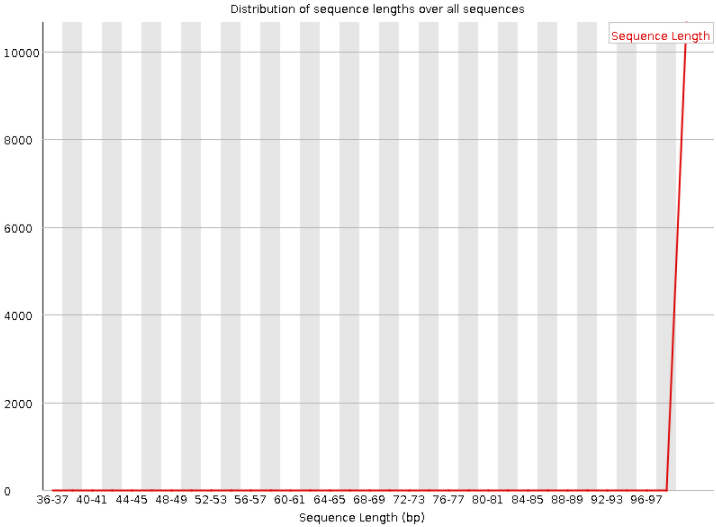
Рисунок 3. Длины ридов до чистки
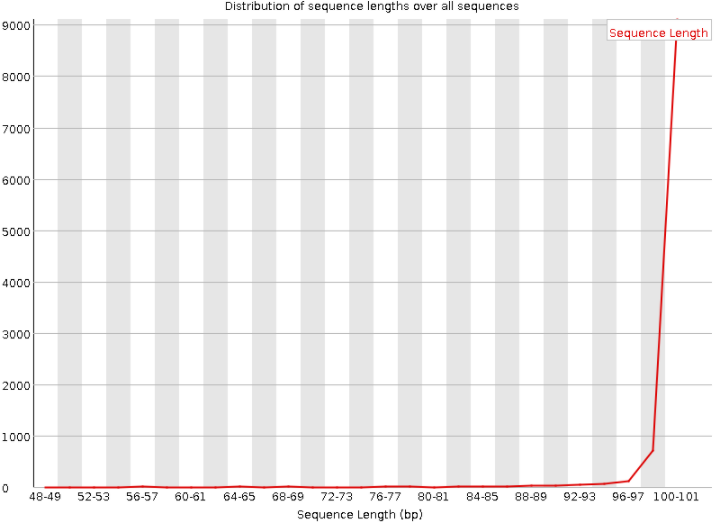
Рисунок 4. Длины ридов после чистки
Сначала я создала требуемую директорию (/nfs/srv/databases/ngs/e.catsrina) и скопировала в нее нужные файлы (chr10.fasta и chr10.fastq).
С помощью программы FastQC (установлена на kodomo) был проведен анализ качества чтений (результат - по ссылке):
| fastqc chr10.fastq
Затем с помощью программы Trimmomatic была проведена очистка чтений: с конца каждого чтения были отрезаны нуклеотиды с качеством ниже 20 (TRAILING:20) и оставлены только чтения длиной не меньше 50 нуклеотидов (MINLEN:50). После чего опять была использована программа FastQC (результат - по ссылке):
| java -jar /usr/share/java/trimmomatic.jar SE -phred33 chr10.fastq chr10_2.fastq TRAILING:20 MINLEN:50 | fastqc chr10_2.fastq
На рисунках 1 и 2 можно увидеть некоторые графических выдачи программы FastQC - Per base sequence quality и длины ридов - до и после чистки соответственно.
 Рисунок 1. До чистки |  Рисунок 2. После чистки |
 Рисунок 3. Длины ридов до чистки |  Рисунок 4. Длины ридов после чистки |
Как видно по этим рисункам, качество ридов (синяя линия) повысилось, особенно в конце ридов, разброс по значениям ("усы") - уменьшился (особенно в конце). Также теперь все позиции попадают желтой областью (все, что между нижним и верхним квартилями) в зеленую зону - в зону с хорошим качеством. Сильнее всего изменился конец, т. к. ридов длиной меньше 50 н было мало, поэтому их выброс повлиял на картину не так сильно, как отрезание некачественных - т. е. как раз концевых - нуклеотидов.
Естественно, изменились длина и число ридов: до очистки было 10666 ридов с длинами 38-100, после - 10526 ридов с длинами 50-100. Изменение распределения длин можно увидеть на рис. 3 и 4: увеличилась минимальная длина (на графике изменилось начало оси абсцисс), из-за уменьшения числа ридов увеличилась доля ридов с длиной от 96 до 100 н (их присутствие стало заметно на графике).
Дальнейшие действия - картирование очищенных чтений по референсной последовательности с помощью программы Hisat2. Действия проводились в директории /nfs/srv/databases/ngs/e.caterina (команда cd /nfs/srv/databases/ngs/e.caterina).
Сначала была извлечена необходимая программа:
| export PATH=${PATH}:/home/students/y06/anastaisha_w/hisat2-2.0.5
Затем была проиндексирована референсная последовательность (программа выдала 8 файлов - chr10.1.ht2 <...> chr10.8.ht2). Ее вывод был записан в файл hisat.txt:
| hisat2-build chr10.fasta chr10 > hisat.txtИ после этого было построено выравнивание очищенных чтений (chr10_2.fastq - обозначается -U) по референсной последовательности (файлам chr10*.ht2 - обозначается -x). Параметры --no-spliced-alignment --no-softclip соответственно означают выравнивание без разрывов и запрет подрезания ридов (обычно оно происходит рядом с концами):
| hisat2 -x chr10 -U chr10_2.fastq --no-spliced-alignment --no-softclip -S chr10.sam
С помощью пакета samtools был произведен анализ выравнивания.
Сначала файл с выравниванием ридов по референсной последовательности (chr10.sam) был переведен в бинарный формат (chr10.bam, перевод в бинарный формат обозначается -b):
| samtools view chr10.sam -o chr10.bam -bЗатем риды в этом файле были отсортированы по своей левой (начальной) координате в референсе (файл chr10_temp.txt - временный файл, необходимый для работы программы, файл chr10_sort.bam - вывод программы):
| samtools sort chr10.bam -T chr10_temp.txt -o chr10_sort.bamПосле чего получившийся файл был проиндексирован:
| samtools index chr10_sort.bamФайлы chr10_sort.bam и chr10_sort.bam.bai (проиндексированный .bam) были визуализированы с помощью программы IGV (см. ниже).
Так же данные для анализа были выданы командой hisat2:
10526 reads; of these: 10526 (100.00%) were unpaired; of these: 131 (1.24%) aligned 0 times 10393 (98.74%) aligned exactly 1 time 2 (0.02%) aligned >1 times 98.76% overall alignment rateОтсюда можно узнать число невыровненных ридов (131 рид, что составляет 1,24% процента от всех ридов), число ридов, выровненных больше одного раза (2 - 0,02%) и, соответственно, число ридов, выровненных ровно 1 раз: 10393 (98.74%).
Файл chr10.sam тоже содержит много информации: для каждого рида в нем написано (помимо его идентификатора, координаты в хромосоме и т. д. и некоторых других параметров):
| samtools depth chr10_sort.bam > depth.txtВ полученном файле depth.txt нуклеотиды были отсортированы по глубине. С наибольшей глубиной (192) оказались нуклеотиды из экзона 12, как следует из программы IGV (см. рис. 5 - в поиск былы вбита ккордината нуклеотида - 63957882). В этой же программе написаны координаты этого экзона: 63952845-63958202. Далее я получила глубины для этого экзона:
| samtools depth chr10_sort.bam -r chr10:63952845-63958202 > depth2.txtПолученный файл был скопирован в .ods, в нем было найдено среднее покрытие. Однако оно оказалось не очень большим - 17,14. Это становится совершенно понятным, если посмотреть на рис. 6, где видно, что это очень большой экзон и далеко не на всей его длине вообще есть риды. Тогда я взяла нуклеотид с максимальным покрытием из другого экзона - им оказался нуклеотид 63995921 (покрытие 188) из экзона 6 того же гена RTKN2 (см. рис. 7). С помощью аналогичной команды и анализа получившегося файла я получила среднее покрытие этого экзона - 154,43.
 Рисунок 5. Окрестность нуклеотида с максимальным покрытием |
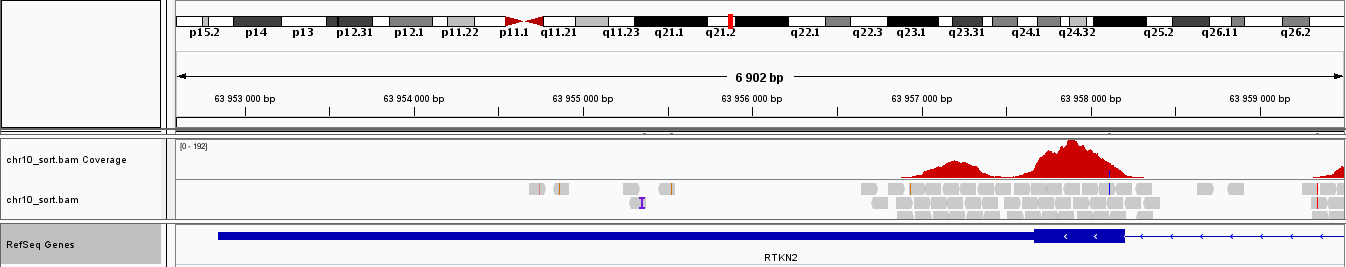 Рисунок 6. Экзон 12 |
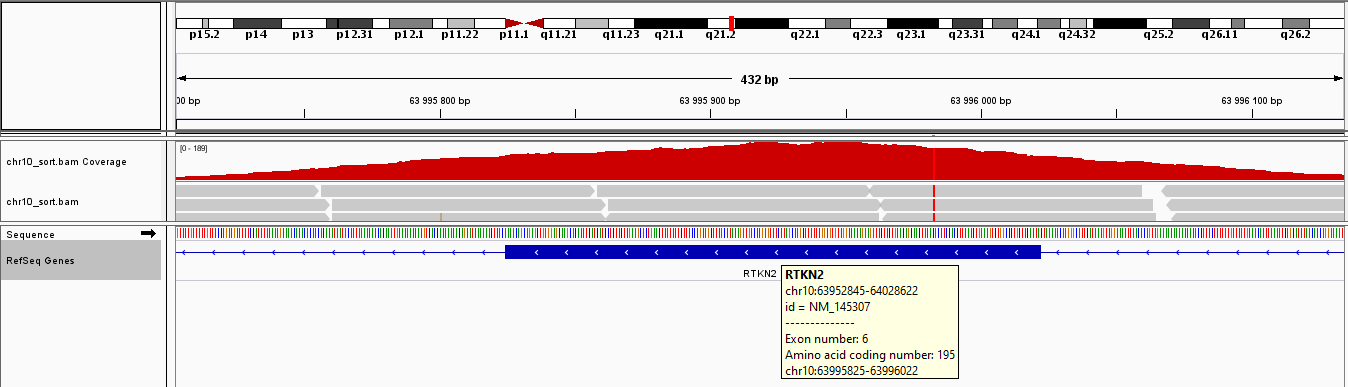 Рисунок 7.Экзон 6 |
Первым делом был создан файл с полиморфизмами (chr10_5.bcf):
samtools mpileup -uf chr10.fasta chr10_sort.bam -o chr10_5.bcfПосле этого с помощью пакета bcftools был создан файл со списком отличий между референсом и чтениями
bcftools call -cv chr10_5.bcf -o chr10_5.vcfАнализ файла chr10_5.vcf позволяет узнать число найденых полиморфизмов - 66 (9 инделей и 57 замен), а также другие параметры - рассмотрим их на примере (визуализацию этих трез случаев с помощью IGV см. ниже):
ID REF ALT QUAL INFO (тип - по умолчанию - замена, DP - глубина прочтения, прочее) FORMAT chr10_sort.bam chr10 5766152 AGTAT AGTATGTAT 92.4668 INDEL;DP=8;IDV=3;IMF=0.375;VDB=0.00746206;SGB=-0.511536;MQ0F=0;AF1=0.5;AC1=1; DP4=5,0,3,0;MQ=60;FQ=95.4767;PV4=1,1,1,1 GT:PL 0/1:130,0,194 chr10 5766337 G A 225.009 DP=93;VDB=0.00111352;SGB=-0.693145;RPB=0.992146;MQB=1;MQSB=1;BQB=0.489669;MQ0F=0; AF1=0.5;AC1=1;DP4=38,11,35,6;MQ=60;FQ=225.007;PV4=0.423127,0.251369,1,1 GT:PL 0/1:255,0,255 chr10 5766572 Catatatatatat CATatatatatatat 10.8393 INDEL;DP=5;IDV=1;IMF=0.2;VDB=0.267404;SGB=-0.379885;MQ0F=0;AF1=0.499996;AC1=1; DP4=0,2,0,1;MQ=60;FQ=13.6582;PV4=1,1,1,1 GT:PL 0/1:48,0,75ID - координата SNP, REF - нуклеотид в референсе, ALT - в выравнивании, QUAL - качество рида, INFO - множество различных параметров (например DP - глубина покрытия в этой позиции, INDEL - то, что это индель (иначе - просто замена нуклеотида)), FORMAT и следующее поле - содержит данные по генотипу (0/1 - диплоидный, гетерозигота (1 - аллель в выравнивании, 0 - в референсе), цифры, соответствующие PL - вероятности этих генотипов).
Файл chr10_5.vcf также был визуализирован с помощью программы IGV (см. ниже).
Аннотирование только что полученных SNP производилось с помощью программы annovar. Сначала вручную были удалены индели из файла chr10_5.vcf (получился файл chr10_6.vcf). Затем этот файл был переведен в нужный формат (perl необходим для получения доступа):
| perl /nfs/srv/databases/annovar/convert2annovar.pl -format vcf4 chr10_6.vcf -o chr10.avinputДалее полученный файл аннотировался по разным базам данных.
По базе данных snp:
| perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -dbtype snp138 -out chr10.snp -build hg19 chr10.avinput /nfs/srv/databases/annovar/humandb/Результат: файлы chr10.snp.hg19_snp138_dropped, chr10.snp.hg19_snp138_filtered, chr10.snp.log. В аннтотациях по типу -filter всегда файл _dropped включает в себя аннотированные snp, _filtered - не аннотированные (не имеющие аннотаций в этой базе данных), .log - сведения о команде. Было получено 54 аннотированных snp (из 57) - т. е. 54 snp имеют rs.
| perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -dbtype 1000g2014oct_all -out chr10_1000g -buildver hg19 chr10.avinput /nfs/srv/databases/annovar/humandb/Результат: файлы chr10_1000g.hg19_ALL.sites.2014_10_dropped, chr10_1000g.hg19_ALL.sites.2014_10_filtered, chr10_1000g.log. Частоту встречаемости см в общей таблице ниже: частоты были найдены для всех snp, кроме трех, минимальная частота - 0.000798722, максимальная - 0.997604, средняя - 0.61, медиана - 0.72.
| perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -dbtype clinvar_20150629 -out chr10_clinvar -buildver hg19 chr10.avinput /nfs/srv/databases/annovar/humandb/Результат: файлы chr10_clinvar.hg19_clinvar_20150629_dropped, chr10_clinvar.hg19_clinvar_20150629_filtered, chr10_clinvar.log. Все аннотированные по этой базе данных snp далжны были быть записаны в файл .hg19_clinvar_20150629_dropped, однако этот файл пуст. Что говорит о том, что никаких известных этой базе данных клинических последствий данные замены не несут.
| perl /nfs/srv/databases/annovar/annotate_variation.pl -regionanno -dbtype gwasCatalog -out chr10_gwas -buildver hg19 chr10.avinput /nfs/srv/databases/annovar/humandb/Результат: Файлы chr10_gwas.hg19_gwasCatalog, chr10_gwas.log. Было найдено всего 3 замены (что составляет всего 5,3% от всех snp), ассоциированные с различными болезнями (остеосаркоме - раку кости, ревматоидному артриту и витилиго - исчезновению меланина с отдельных участков кожи) - см ниже (и в общей таблице). Интересно, что snp, приводящий к витилиго, назодится не в экзоне (по крайней данным refgen), в отличие от двух других snp:
База данных Проявление Координата REF ALT генотип Качество рида Глубина рида gwasCatalog Name=Osteosarcoma chr10 5804531 G A het 225.009 70 gwasCatalog Name=Rheumatoid arthritis chr10 63958112 T C het 225.009 75 gwasCatalog Name=Vitiligo chr10 115481018 C T het 225.009 91
| perl /nfs/srv/databases/annovar/annotate_variation.pl -out chr10_refg -build hg19 chr10.avinput /nfs/srv/databases/annovar/humandb/Результат: Файлы chr10_refg.exonic_variant_function, chr10_refg.variant_function, chr10_refg.log. В интронах содержится 45 замен, в экзонах 10 (причем из них 3 синонимичных и 10 несинонимичных), в UTR3 (нетранслируемый участок на 3'-конце зрелого транскрипта - после стоп-кодона) - 2. Информация о заменах в экзонах (включая получившуюся замену аминокислоты) сведена с таблицу в файле .exonic_variant_function:
Тип Ген (два ID):экзон:замена нукл:замена а. к. Координата REF ALT генотип Качество рида Глубина рида nonsynonymous SNV FAM208B:NM_017782:exon13:c.T1495G:p.C499G, chr10 5781628 T G het 117.008 21 nonsynonymous SNV FAM208B:NM_017782:exon14:c.A2419G:p.I807V, chr10 5784151 A G het 225.009 83 nonsynonymous SNV FAM208B:NM_017782:exon15:c.G3224C:p.R1075P, chr10 5788608 G C het 108.008 14 nonsynonymous SNV FAM208B:NM_017782:exon15:c.T5036C:p.V1679A, chr10 5790420 T C het 135.008 14 nonsynonymous SNV FAM208B:NM_017782:exon20:c.G7211A:p.S2404N, chr10 5804531 G A het 225.009 70 nonsynonymous SNV RTKN2:NM_145307:exon12:c.A1385G:p.H462R, chr10 63958112 T C het 225.009 75 nonsynonymous SNV CASP7:NM_001267057:exon2:c.T236G:p.I79S, CASP7:NM_001227:exon2:c.T12G:p.D4E, CASP7:NM_033338:exon3:c.T111G:p.D37E, CASP7:NM_001267056:exon2:c.T12G:p.D4E, CASP7:NM_033340:exon2:c.T12G:p.D4E, CASP7:NM_033339:exon3:c.T12G:p.D4E, chr10 115457264 T G het 225.009 71 synonymous SNV RTKN2:NM_145307:exon10:c.G1149A:p.K383K, chr10 63964653 C T het 206.009 38 synonymous SNV RTKN2:NM_145307:exon6:c.G528A:p.V176V, chr10 63995983 C T hom 221.999 154 synonymous SNV FAM208B:NM_017782:exon13:c.A1836T:p.T612T, chr10 5781969 A T het 225.009 18
Ниже (в таблице 1) приведены все SNP с аннотациями их этих баз данных (базы Clinvar нет, потому что она не выдала ни одной аннотации).
| Координата | REF | ALT | Качество | Глубина | 1000 genoms частота | SNP (наличие rs) | RefGen (где snp) | Ген | Тип замены | Ген:экзон:замены нукл замена а. к. (REF:номер:ALT) | GWAS |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 5727202 | C | T | 43.0073 | 5 | 0.276757 | rs7091641 | intronic | FAM208B | |||
| 5729603 | T | C | 7.79993 | 1 | 0.919529 | rs4748112 | intronic | FAM208B | |||
| 5729640 | A | G | 11.3429 | 1 | 0.994808 | rs2386625 | intronic | FAM208B | |||
| 5754247 | G | T | 3.0136 | 1 | 0.000798722 | intronic | FAM208B | ||||
| 5757984 | A | G | 11.3429 | 1 | rs60603196 | intronic | FAM208B | ||||
| 5762405 | C | G | 199.009 | 21 | 0.997604 | rs10752093 | intronic | FAM208B | |||
| 5764029 | A | G | 6.20226 | 1 | 0.977835 | rs10737017 | intronic | FAM208B | |||
| 5766337 | G | A | 225.009 | 93 | 0.838259 | rs10737018 | intronic | FAM208B | |||
| 5770873 | T | C | 8.64911 | 1 | intronic | FAM208B | |||||
| 5781628 | T | G | 117.008 | 21 | 0.83746 | rs2254067 | exonic | FAM208B | nonsynonymous SNV | FAM208B:NM_017782:exon13:c.T1495G p.C499G | |
| 5781969 | A | T | 225.009 | 18 | 0.837061 | rs2797486 | exonic | FAM208B | synonymous SNV | FAM208B:NM_017782:exon13:c.A1836T p.T612T | |
| 5784151 | A | G | 225.009 | 83 | 0.140575 | rs45575338 | exonic | FAM208B | nonsynonymous SNV | FAM208B:NM_017782:exon14:c.A2419G p.I807V | |
| 5788608 | G | C | 108.008 | 14 | 0.836062 | rs2797491 | exonic | FAM208B | nonsynonymous SNV | FAM208B:NM_017782:exon15:c.G3224C p.R1075P | |
| 5790420 | T | C | 135.008 | 14 | 0.977436 | rs2669142 | exonic | FAM208B | nonsynonymous SNV | FAM208B:NM_017782:exon15:c.T5036C p.V1679A | |
| 5795413 | T | C | 4.77219 | 1 | 0.836462 | rs2669137 | intronic | FAM208B | |||
| 5795574 | T | C | 8.64911 | 1 | 0.835863 | rs2797494 | intronic | FAM208B | |||
| 5798413 | T | G | 100.008 | 18 | 0.994609 | rs2797495 | intronic | FAM208B | |||
| 5798730 | G | T | 225.009 | 34 | 0.139577 | rs3793707 | intronic | FAM208B | |||
| 5799144 | A | G | 5.46092 | 2 | 0.140575 | rs55837307 | intronic | FAM208B | |||
| 5799286 | T | A | 73.0074 | 12 | 0.545927 | rs2246399 | intronic | FAM208B | |||
| 5804388 | T | C | 172.009 | 25 | 0.98762 | rs2797500 | intronic | FAM208B | |||
| 5804531 | G | A | 225.009 | 70 | 0.854034 | rs2797501 | exonic | FAM208B | nonsynonymous SNV | FAM208B:NM_017782:exon20:c.G7211A p.S2404N | Osteosarcoma |
| 5804865 | C | T | 225.009 | 51 | 0.14397 | rs56352451 | intronic | FAM208B | |||
| 63955526 | A | G | 6.20226 | 1 | 0.222444 | rs12253181 | UTR3 | RTKN2 (NM_145307:c.*2141T>C) | |||
| 63958112 | T | C | 225.009 | 75 | 0.588858 | rs3125734 | exonic | RTKN2 | nonsynonymous SNV | RTKN2:NM_145307:exon12:c.A1385G p.H462R | Rheumatoid arthritis |
| 63959342 | A | T | 100.008 | 8 | 0.194688 | rs7076490 | intronic | RTKN2 | |||
| 63964653 | C | T | 206.009 | 38 | 0.140575 | rs41274060 | exonic | RTKN2 | synonymous SNV | RTKN2:NM_145307:exon10:c.G1149A p.K383K | |
| 63971272 | T | C | 7.79993 | 1 | 0.753994 | rs7092870 | intronic | RTKN2 | |||
| 63974950 | C | G | 196.999 | 38 | 0.753994 | rs4559572 | intronic | RTKN2 | |||
| 63977069 | A | T | 221.999 | 35 | 0.754193 | rs1432414 | intronic | RTKN2 | |||
| 63977757 | C | T | 141.032 | 8 | 0.719249 | rs7895712 | intronic | RTKN2 | |||
| 63978232 | A | T | 11.3429 | 1 | 0.719249 | rs4147232 | intronic | RTKN2 | |||
| 63982984 | A | G | 221.999 | 51 | 0.753994 | rs10740069 | intronic | RTKN2 | |||
| 63983264 | T | G | 40.0075 | 11 | 0.613419 | rs6479792 | intronic | RTKN2 | |||
| 63983266 | C | G | 118.008 | 11 | 0.11881 | rs12359251 | intronic | RTKN2 | |||
| 63983295 | C | A | 144.032 | 8 | 0.753994 | rs6479793 | intronic | RTKN2 | |||
| 63985383 | C | G | 9.52546 | 1 | 0.140775 | rs1545020 | intronic | RTKN2 | |||
| 63987449 | T | C | 47.7647 | 2 | 0.909345 | rs10740070 | intronic | RTKN2 | |||
| 63992502 | C | G | 9.52546 | 1 | 0.717053 | rs10761615 | intronic | RTKN2 | |||
| 63995983 | C | T | 221.999 | 154 | 0.906949 | rs3852448 | exonic | RTKN2 | synonymous SNV | RTKN2:NM_145307:exon6:c.G528A p.V176V | |
| 63999263 | G | A | 165.003 | 13 | 0.715455 | rs2393869 | UTR3 | RTKN2 (NM_001282941:c.*140C>T) | |||
| 64000878 | T | G | 221.999 | 17 | 0.751597 | rs1368158 | intronic | RTKN2 | |||
| 64001114 | T | C | 49.0072 | 5 | 0.570687 | rs1368157 | intronic | RTKN2 | |||
| 64005711 | C | T | 221.999 | 35 | 0.717851 | rs3815999 | intronic | RTKN2 | |||
| 64014521 | A | T | 11.3429 | 1 | 0.0886581 | rs10995095 | intronic | RTKN2 | |||
| 64015918 | T | C | 10.4247 | 1 | 0.7502 | rs6479795 | intronic | RTKN2 | |||
| 64016783 | C | T | 5.46383 | 1 | 0.716454 | rs3852407 | intronic | RTKN2 | |||
| 64018865 | T | C | 9.52546 | 1 | 0.750399 | rs4115469 | intronic | RTKN2 | |||
| 64022747 | A | T | 88.5135 | 5 | 0.716454 | rs3852408 | intronic | RTKN2 | |||
| 64026108 | C | T | 8.64911 | 1 | 0.709864 | rs4266956 | intronic | RTKN2 | |||
| 64026793 | A | G | 9.52546 | 1 | 0.7502 | rs7076565 | intronic | RTKN2 | |||
| 115444419 | A | G | 5.46383 | 1 | 0.178115 | rs11196425 | intronic | CASP7 | nonsynonymous SNV | CASP7:NM_001267057:exon2:c.T236G p.I79S CASP7:NM_001227:exon2:c.T12G p.D4E CASP7:NM_033338:exon3:c.T111G p.D37E CASP7:NM_001267056:exon2:c.T12G p.D4E CASP7:NM_033340:exon2:c.T12G p.D4E CASP7:NM_033339:exon3:c.T12G p.D4E | |
| 115457264 | T | G | 225.009 | 71 | 0.105631 | rs11555408 | exonic | CASP7 | nonsynonymous SNV | CASP7:NM_001267057:exon2:c.T236G p.I79S CASP7:NM_001227:exon2:c.T12G p.D4E CASP7:NM_033338:exon3:c.T111G p.D37E CASP7:NM_001267056:exon2:c.T12G p.D4E CASP7:NM_033340:exon2:c.T12G p.D4E CASP7:NM_033339:exon3:c.T12G p.D4E | |
| 115472719 | G | A | 7.79993 | 1 | 0.696086 | rs10749141 | intronic | CASP7 | |||
| 115480582 | T | C | 65.0073 | 10 | 0.717053 | rs11196449 | intronic | CASP7 | |||
| 115481018 | C | T | 225.009 | 91 | 0.261182 | rs3814231 | intronic | CASP7 | Vitiligo | ||
| 115488800 | C | A | 4.76948 | 2 | intronic | CASP7 |
Таблица 1. SNP и их аннотации
С помощью программы IGV был визуализирован участок, в котором есть индель и две замены (см рис. 5 и 6):
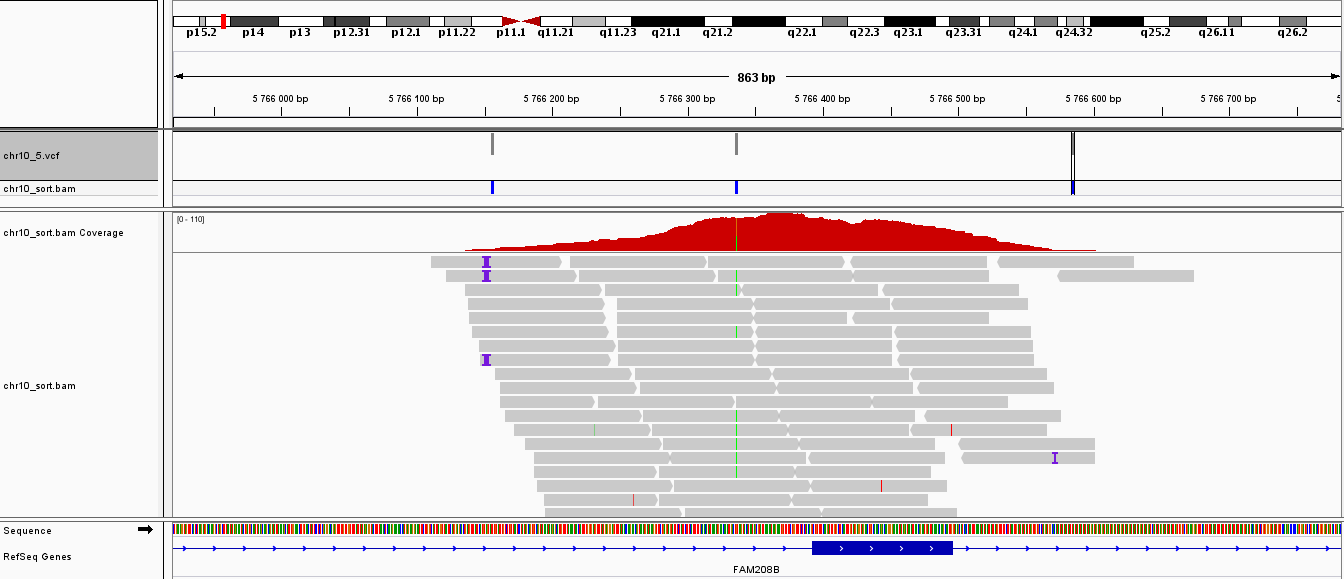
Рисунок 5.Визуализация
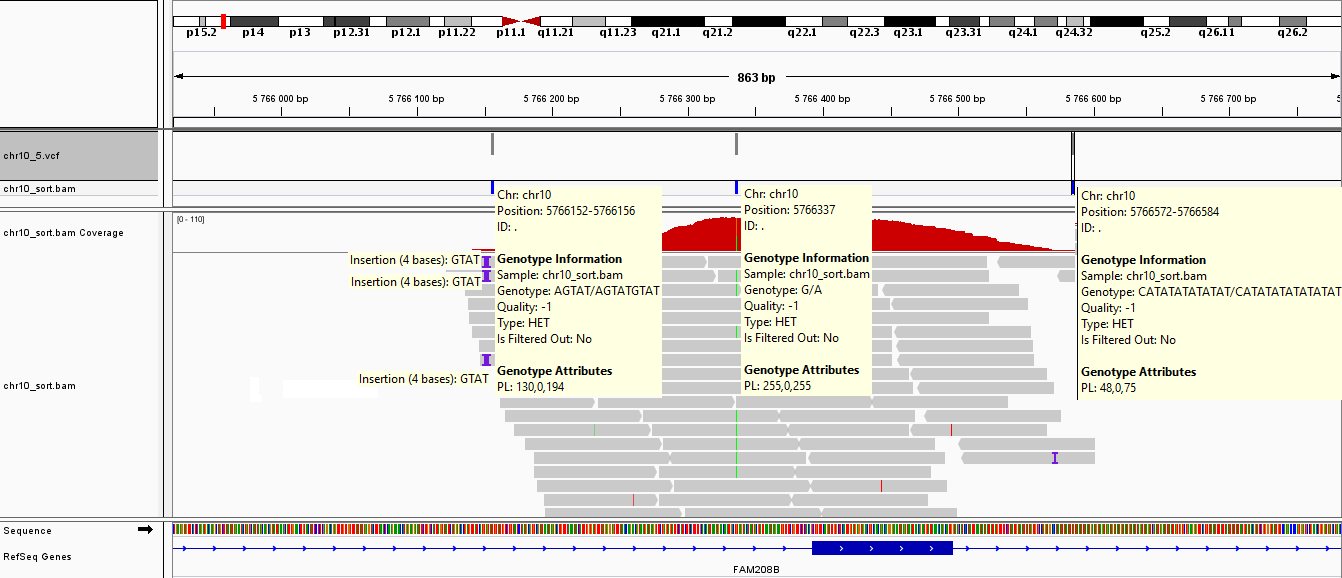
Рисунок 6.Визуализация
НАЗАД ➜ |
| © <Рюмина Екатерина>, 2017 |