Трансмембранные белки
1. База данных OPM
Alpha-helical polytopic Transmembrane classes: Alpha-helical polytopic и Beta-barrel transmembrane. Nitric oxide reductase и Cation efflux system protein CusC.
| Nitric oxide reductase; ID: 4xyd | |
|---|---|
| Толщина гидрофобной части белка в мембране | 31.4 ± 1.1 А |
| Координаты трансмембранных спиралей | A - Tilt: 8° - Segments: 1( 8- 35), 2( 46- 70), 3( 84- 107), 4( 128- 147), 5( 158- 176), 6( 186- 209), 7( 227- 247), 8( 255- 279), 9( 296- 322), 10( 329- 352), 11( 368- 392), 12( 413- 438) B - Tilt: 11° - Segments: 1( 9-30) |
| Среднее количество остатков в одной трансмембранной спирали белка (В) | 22 |
| В какой мембране находится белок | Внутрення мембрана Грам-отрицательных бактерий |
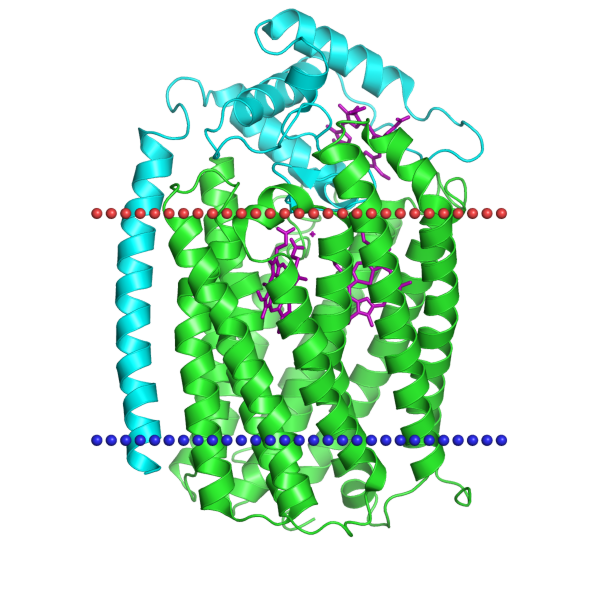
Рис. 1.
| Cation efflux system protein CusC; ID: 3pik | |
|---|---|
| Толщина гидрофобной части белка в мембране | 24.3 ± 1.2 A |
| Координаты трансмембранных спиралей | A - Tilt: 48° - Segments: 1( 83- 93), 2( 107- 117), 3( 289- 299), 4( 315- 326) B - Tilt: 50° - Segments: 1( 83- 93), 2( 107- 117), 3( 289- 299), 4( 315- 326) C - Tilt: 48° - Segments: 1( 83- 93), 2( 107- 117), 3( 289- 299), 4( 315- 326) |
| Среднее количество остатков в одной трансмембранного тяжа белка | 21 |
| В какой мембране находится белок | Внешняя мембрана Грам-отрицательных бактерий |
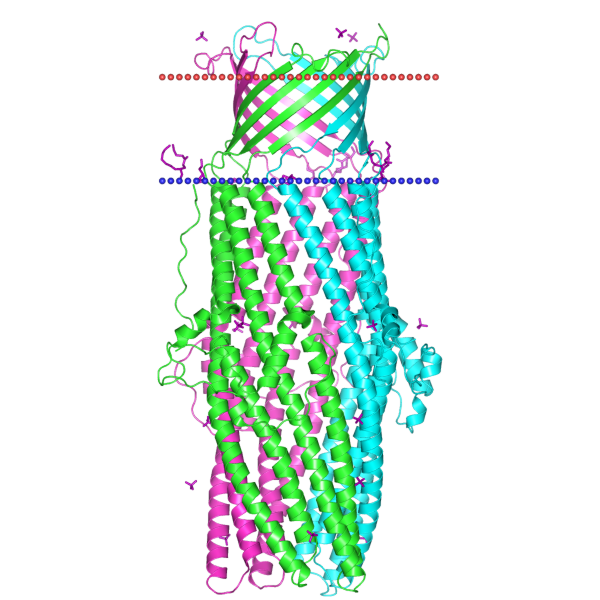
Рис. 2.
2. Анализ предсказания трансмембранных спиралей
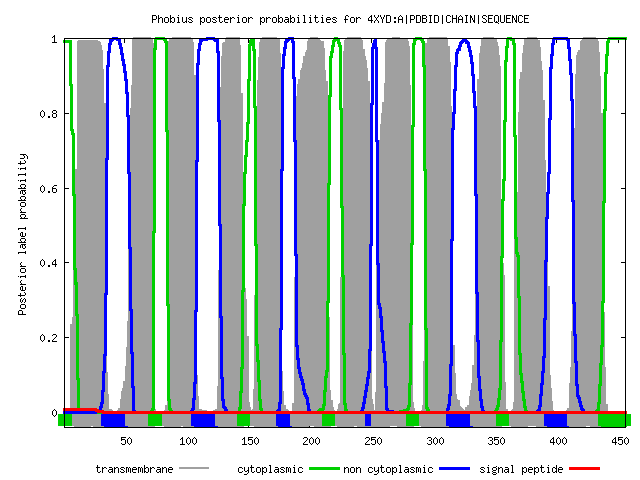 | 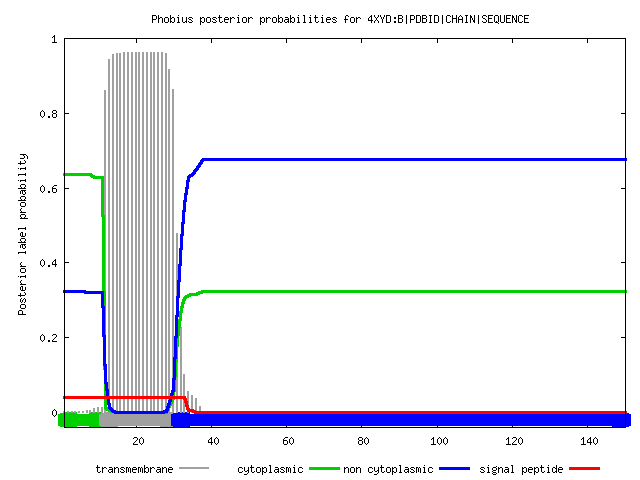 |
Рис. 3. Phobius prediction: весь белок и одна предсказанная спираль. Ось Х - номера аминокислот в последовательности белка, ось У - вероятность нахождения трансмембранной спирали. Разными цветами обозначается линии, соответствующие графику вероятности либо трансмембранного участко (серый цвет), либо выступающего в цитоплазму (зеленый), либо не присутсвующего в цитоплазме (синий), либо сигнальной последовательности (красный, в выбранном белке отсутствует).
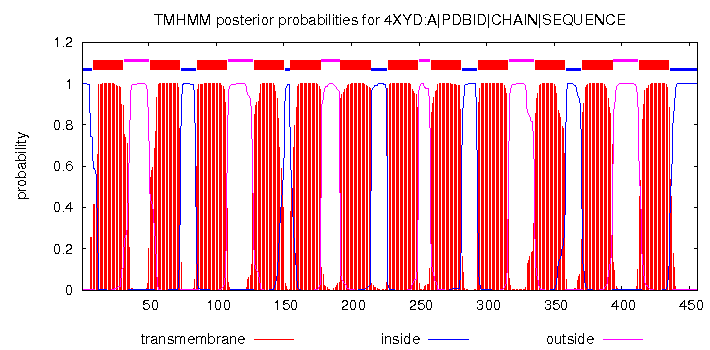 | 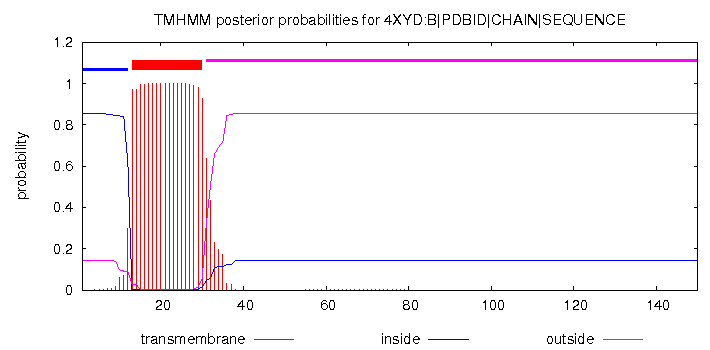 |
Рис. 4. TMHMM result: весь белок и одна предсказанная спираль. Ось Х - номера аминокислот в последовательности белка, ось У - вероятность нахождения трансмембранной спирали. Разными цветами обозначается линии, соответствующие графику вероятности либо трансмембранного участко (красный цвет), либо выступающего внутрь клетки (синий), либо выступающий наружу клетки (розовый)
Программы Phobius и TMHMM выдали одинаковы результат: обе нашли все 12 спиралей субъединицы А, указанные в ОРМ, координаты спиралей примерно совпадают с таковыми в ОРМ - см. табл. 1.
| Phobius prediction | TMHMM result | OPM | |||
|---|---|---|---|---|---|
| TRANSMEM | 12 35 | TMhelix | 9 31 | 1( 8- 35) | |
| TRANSMEM | 55 73 | TMhelix | 51 73 | 2( 46- 70) | |
| TRANSMEM | 85 108 | TMhelix | 86 108 | 3( 84- 107) | |
| TRANSMEM | 128 145 | TMhelix | 128 150 | 4( 128- 147) | |
| TRANSMEM | 157 177 | TMhelix | 155 177 | 5( 158- 176) | |
| TRANSMEM | 189 214 | TMhelix | 192 214 | 6( 186- 209) | |
| TRANSMEM | 226 249 | TMhelix | 227 249 | 7( 227- 247) | |
| TRANSMEM | 255 282 | TMhelix | 259 281 | 8( 255- 279) | |
| TRANSMEM | 294 315 | TMhelix | 294 316 | 9( 296- 322) | |
| TRANSMEM | 335 355 | TMhelix | 336 358 | 10( 329- 352) | |
| TRANSMEM | 367 394 | TMhelix | 371 393 | 11( 368- 392) | |
| TRANSMEM | 414 438 | TMhelix | 413 435 | 12( 413- 438) | |
Таблица 1. Координаты трансмембранных доменов - Phobius, TMHMM и ОРМ.
3. База данных TCDB
Из двух белков из пункта 1 был найден только один: Cation efflux system protein CusC (3pik) - TCID: 1.B.17.3.5. Это переносчик одновалентных ионов меди и серебра, расположенный во внешней мембране. Известна его 3D структура (из кристаллографии), касающаяся механизма сворачивания белка, в результате которого получается канал.
Расшифровка TC-кода:
- Позиция 1: число - класс транспортера (например, канал)
- Позиция 2: буква - подкласс
- Позиция 3: число - семейство
- Позиция 4: число - подсемейство
- Позиция 5: число - конкретный белок (транспортер)
