PSI-BLAST
С помощью PSI-BLAST и поиска по банку Swiss-Prot было составлено семейство гомологов для белка P17265 (RecName: Full=Ribosome hibernation promotion factor; Short=HPF). Результат представлен в таблице 1.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 17 | P0A147.1 | 7e-04 | P26983.1 | 0.027 |
| 2 | 27 | P33987.1 | 1e-08 | P9WMA8.1 | 0.015 |
| 3 | 28 | P9WMA8.1 | 0.002 | Q6P9R4.2 | 0.028 |
| 4 | 28 | P24694.1 | 3e-18 | P27321.3 | 0.22 |
| 5 | 28 | P24694.1 | 2e-18 | P27321.3 | 0.19 |
Таблица 1. Семейство гомологичных белков (PSI-BLAST)
Уже на четвертой итерации список находок выше порога не поменялся по сравнению с предыдущей итерацией, что говорит о том, что это семейство четко обособлено. Также для оценки качества семейства ниже приведены названия (функции) полученных 28 белков:
- Ribosome hibernation promotion factor; Short=HPF - 24 белка
- Ribosome-associated factor Y; Short=pY - 2 белка
- Ribosome-binding factor PSRP1, chloroplastic; Short=CS5; - 1 белок
- Dormancy associated translation inhibitor; Short=DATIN - 1 белок
Видно, что подавляющее число белков имеют ту же функцию (Ribosome hibernation promotion factor), что и выбранный изначально белок, остальные же либо тоже связаны с рибосомой (либо непосредственно, либо функциоонально).
Помимо этого ступенька между худшей "правильной" находкой и "лучшей" неправильной весьма значительна, что так же свидетельствует в пользу того, что это действительно семейство гомологичных белков.
Prosite
Цель: уточнить паттерн семейств белков из практикума 2 так, чтобы он описывал не все белки данного семейства, а только белки протеобактерий. Паттерн искался с помощью сканирования белка RS2_ECOLI. Было найдена 2 паттерна, далее расматривался первый из них: RIBOSOMAL_S2_1, PS00962; Ribosomal protein S2 signature 1 (PATTERN). Последовательность данного паттерна: MrDMLKAGVHFG. По выравниванию из практикума 2 (Muscle with Default) были добавлены 2 консервативные колонки: MrDMLKAGVHFGHQT (см. рис. - до и после добавления).
 Рисунок 1. Результаты поиска Prosite | 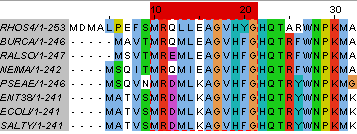 Рисунок 2. Паттерн в выравнивании  Рисунок 3. Более строгий паттерн |
- TP = 76
- FP = 1 (P34831)
- NF = 328
