Реконструкция эволюции доменной архитектуры
1. Выбор семейства доменов
Мной было выбрано семейство Kazal_2 (AC: PF07648), включающее 395 архитектур, 7303 последовательностей и 330 видов. Наличие этого домена обычно указывает на функцию ингибирования серин протеазы, однако он есть и в некоторых белках без этой активности. Список разных доменных архитектур с выбранным доменом и указанием числа последовательностей можно посмотреть по ссылке.
2. Выбор архитектур
Выбранные архитектуры и число представителей для каждой указаны на рисунках 1 и 2.

Рисунок 1. Архитектура 1 (далее - "yel")
Рисунок 2. Архитектура 2 (далее - "red")
Первая архитектура содержит еще один домен: Organic anion-transporting polypeptide (OATP, AC: PF03137). Вторая содержит еще два домена: Secreted protein acidic and rich in cysteine Ca binding region (SPARC_Ca_bdg, AC: PF10591) и Thyroglobulin type-1 repeat (Thyroglobulin_1, AC: PF00086).
Далее с помощью скрипта была получена информация о всех последовательностях данного семейства:
| python swisspfam-to-xls.py -z -i /srv/databases/pfam/swisspfam.gz -p PF07648 -o PF07648.xls
По полученной таблице была построена сводная таблица (строки – AC последовательностей, столбцы – домены Pfam). После чего по сводной таблице были выбраны последовательности, соответствующие выбранным архитектурам и записаны их АС (в сводной таблице последовательности, соответствующие архитектуре 1 выделены желтым, архитектуре 2 - красным, откуда их обозначения).
3. Выбор таксона и подтаксонов
Для полуучение информации о таксономической принадлежности последовательностей сначала были скачаны полные записи всех последовательностей. Для этого список выбранных ранее АС бы загружен в Uniprot - Retrieve и сохренен в файл uniprot-yourlist.txt. После чего был использован скрипт для получения таксономии:
| python uniprot-to-taxonomy.py -i uniprot-yourlist.txt -o taxonomy.xls
Полученные данные были добавлены к основной таблице с помощью VLOOKUP (OpenOffice). После чего были выбраны таксон - Metazoa (многоклеточные животные), и два его подтаксона - Chordata (хордовые) и Ecdysozoa (линяющие). С помощь скрипта были выбраны последовательности, соответствующие данным подтаксонам:
| python filter-alignment.py -i align.fasta -m select.txt -o selected.fasta -a "_"
Полученные последовательности были переименованы (E = Ecdysozoa, C = Chordata, yel = архитектура 1 (см. рис. 1), red = архитектура 2 (см. рис. 2)) и загружены в Jalview, где были объединены в группы по архитектурам. В полученном выравнивании были удалены пустые столбцы (внутри групп), N- и C-концевые участки в том случае, если в них очевидно не было хорошего выравнивания, а также в некоторых местах выравнивания были подправлены. К последовательностям также была добавлена последовательность FST_HUMAN с известной 3D архитектурой. Проект можно скачать по ссылке, часть его представлена на рис. 3 (выравнивание включает только фрагменты, представляющие выбранный домен, а не все последовательности белков целиком).
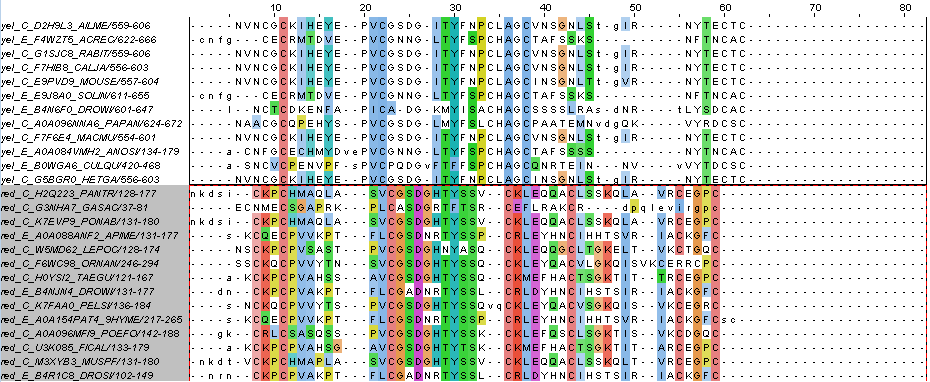
Рис. 3. Выравнивание
Построение филогенетического дерева домена
С помощью программы Mega методом Neighbor-Joining были построены филогенетические деревья для двух архитектур (см. рис. 4 и 5), для проверки достоверности ветвей был использован метод Bootstrap (100 реплик). Красным цветом отмечены ветви, принадлежащие подтаксону Chordata, зеленым - Ecdysozoa.
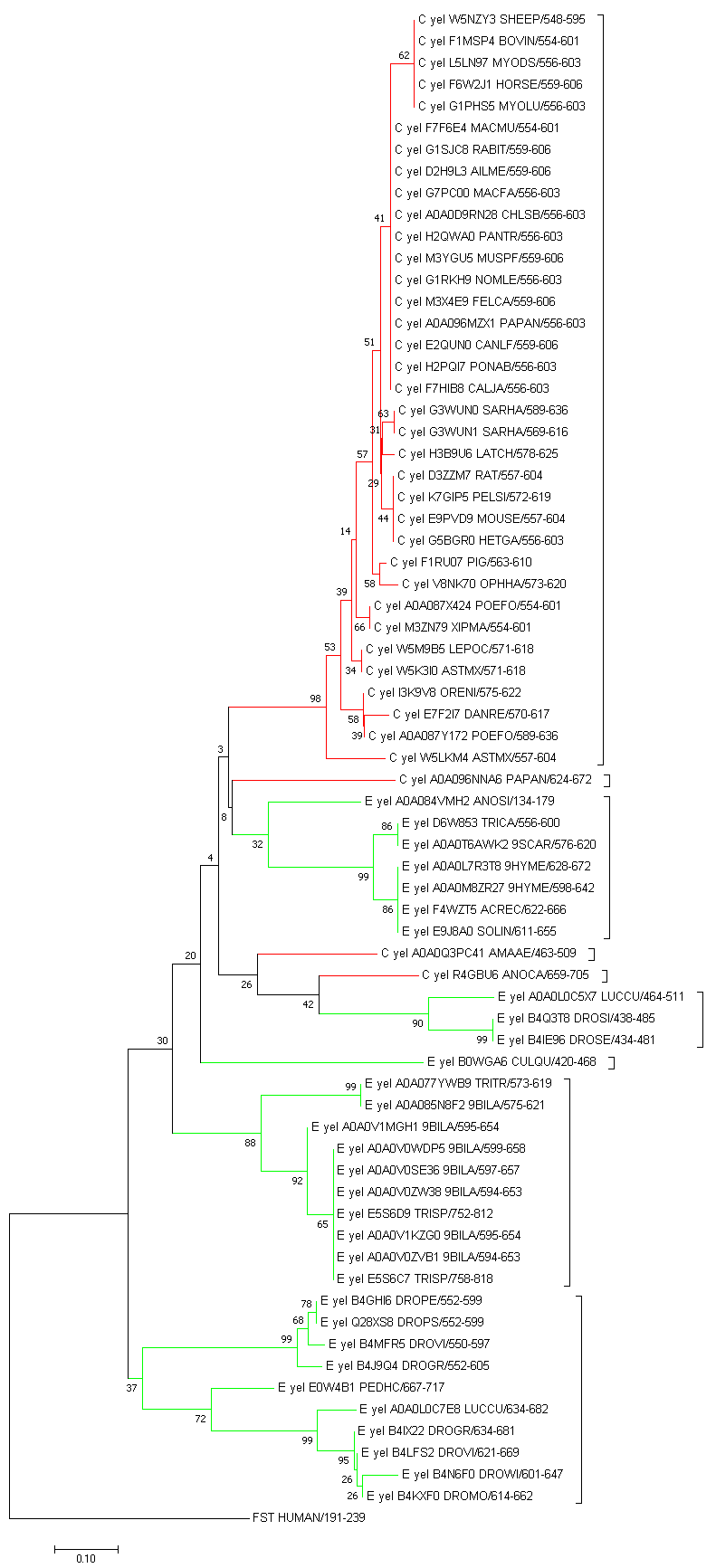 Рис. 4. Дерево первой (yel) структуры | Рис. 5. Дерево второй (red) структуры |
| Скобочная формула | Скобочная формула |
Видно, что в архитектуре red, все домены Chordata являются ортологами по отношению к доменам Ecdysozoa (разделение произошло 1 раз). В архитектуре yel все сложнее: после разделения на Chordata и Ecdysozoa (произошеднее на ветви с достоверностью 4) в кладе, соответствующей Chordata, есть две клады с доменами Ecdysozoa.
