



 |  |  |  |
Выравнивание как отражение эволюции. JalView |
Вернуться на страницу семестра Выравнивание белков семейства HSP70В данной работе мы проводим выравнивание 6 белков семейства HSP70 из эукариот (P11141, O24581), архей (Q6L0S7, A0RZ01), бактерий (Q8YW74, B2UKV6). Общие сведения о белках и организмах приведены в таблице 1. Таблица 1. Идентификаторы и общие сведения
Ниже приведено выравнивание, полученное с помощью JalView, с выделенными абсолютно консервативными позициями и дополнительной строкой "Разметка". Её назначение будет описано ниже. Выравнивание последовательностей белков 1)Cтоит отметить, что на картинке уже исправленный вручную вариант, потому что Jalview иногда подгоняет 100%-ное совпадение там, где это маловероятно. Часто это происходит в участках, где много гэпов и инделей. Особенно много гэпов можно наблюдать в концах цепей, но нужно понимать, что они не все достоверные. Также в некоторых участках значки G и С совпадали, в таком случае я оставляла только C. 2)На разметке отмечены типы консервативности: консервативные на 80% или более (C), абсолютно функциональные консервативные позиции (F), позиции с гэпами (G). Следует дополнительно пояснить для F - это колонки, где аминокислотные остатки имеют похожие свойства и относятся в одинаковые группы классификации аминокислот. Так, например, можно объединить аспарагиновую и гмутаминовую кислоты; алифатические аминокислоты и тд. В концевом участке я не ставила гэпы, потому что этот участок недостоверный и гэпы не могут свидетельствовать о делециях. 3)Длина выравнивания - 727 c учётом гэпов, которые вставляет программа. На выравнивании можно увидеть, какие колонки я посчитала недостоверными. Также хорошо заметны блоки - обратите внимание на участки дополнительной разметки, где нет G, но подряд идут C или F. Блок - это прямоугольник в выравнивании, для которого предполагается гомология между остатками из всех последовательностей, входящих в блок, для каждой колонки блока. Фрагменты последовательностей, входящих в блок, должны быть похожи, т.е. блок должно содержать много абсолютно консервативных или функционально консервативных позиций [7] Таблица 2. Параметры консервативности белков
Искусственная эволюция белкаЭволюцию белка подчиняется следующим условиям:
Геном первого предка, выбранный для эксперимента
При помощи программы Jalview проводим выравнивание и пытаемся отследить созданную нами эволюционную линию Результат, выданный программой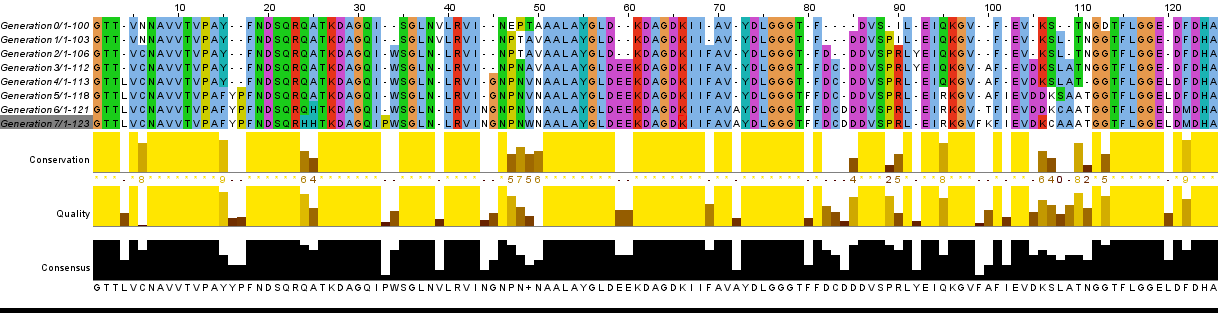 Выравнивание, после исправлений вручную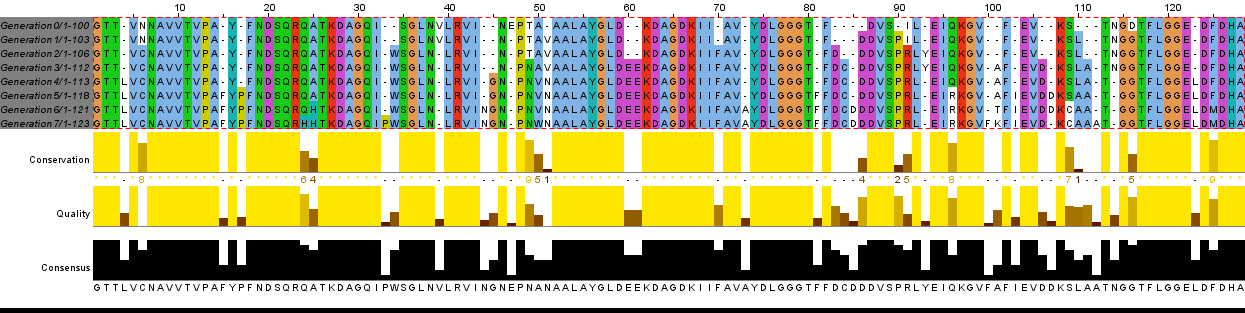 Тирозины в поколениях 0 — 4 с 15 места перенесены на 16 с появдением гэпа. Видно, что тирозин созраняется в этой колонке во всех 7 поколениях, поэтому и было сделано исправление Глобальная перестановка нуклеотидов с 45 по 50. Программа не поняла делецию E в первом же поколении, из-за этого выравнивание вышло не совсем удачным на этом участке Устранены явные ошибки программы в районе 110 а. м. о. Вставлены недостающие гэпы Ход эволюцииПосле исправления ошибок программы, проще заметить мутации в поколениях (описаны первые 10)1 поколение: среди первых 10 мутаций не встретилось мутаций в 1 поколении 2 поколение: замена аспарагина на цистеин в 6 позиции, вставка триптофана в 34 позиции, делеция валина 39 3 поколение: среди первых 10 мутаций не встретилось 4 поколение: вставка лейцина в 4 позиции 5 поколение: вставка фенилаланина в 15 позиции, вставка пролина в 17 позиции 6 поколение: замена аланина на гистидин в 25 позиции, вставка аспарагина 44 7 поколение: замена глутамина на гистидин в 24 позиции, вставка пролина в 33 позиции Искусственная эволюция белка с изменением нуклеотидной последовательностиЭволюция белка [8] подчиняется следующим условиям:
msbar -count 3 -point 1 -block 1 -codon 1
Последовательность белка первого предка
Нуклеотидная последовательность белка
Результат выравнивания, выданный программой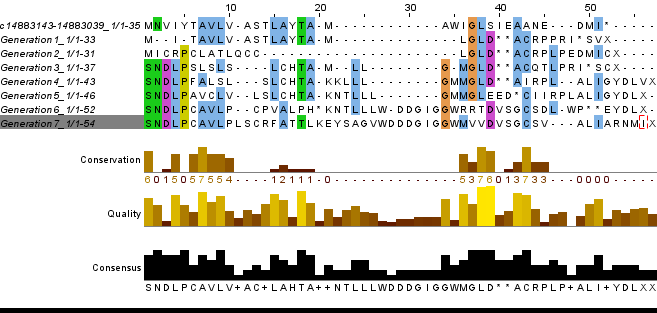 Исправления вручную автоматического выравнивания не было сделано, тк из-за множественных замен в нуклеотидной последовательности последовательность аминокислот изменилась до неузнаваемости, выравнивание нельзя считать удачным из-за огромного количества гэпов, поэтому и исправить что-либо проблематично. Абсолютно консервативной не осталась ни одна колонка. Конечно, из-за большого количества случайных мутаций произошло смещение рамки считывания, поэтому относительную консервативность можно проследить только в начале выравнивания в 1-5 позициях. При этом из-за того, что программе было разрешено изменять целые блоки нуклеотидов, проследить изменения через поколения не так просто как в 1 случае. Остаются видны лишь некоторые мутации, например, замена метионина в 3 поколении на серин, что может стать для белка летальным, так как пропадёт старт-кодон, либо увеличить длину цепи, если впереди есть другой метионин. Также значком * помечены стоп-кодоны, (мутация с появлением стоп-кодона - нонсенс) которые также нарушат начальную структуру белка. Take home messagesСпорные утверждения из лекции:Только мутации в половых клетках наследуются: Неверно, потому что существуют ещё, например, соматические мутации – изменения наследственного материала в соматических клетках. Такая мутация передается только потомкам мутантной клетки в процессе митоза. К примеру, такие мутации могут наследоваться при вегетативном размножении растений. Последовательность белка обычно под стабилизирующим отбором: Не всегда верно, потому что в неблагоприятных условиях белок находится под движущим отбором. Из-за бурного развития фармацевтики и, в частности, антибиотиков такому отбору подвергается большое количество бактерий, прямо сейчас пытающихся выработать приспособленность к новому лекарству. Можно считать, что данные патогены обычно находятся в неблагоприятных условиях, следовательно, под влияние движущего отбора. Скрипт имитации эволюции для 7 поколений аминокислотной последовательности Скрипт имитации эволюции для 7 поколений нуклеотидной последовательности Ссылка на скачивание выравниваний заданий 1, 2, 3 Источники [1] UniProtKB - Q6L0S7 (DNAK_PICTO) [2] UniProtKB - A0RZ01 (DNAK_CENSY) [3] UniProtKB - Q8YW74 (DNAK2_NOSS1) [4] UniProtKB - B2UKV6 (DNAK_AKKM8) [5] UniProtKB - P11141 (HSP7F_CAEEL) [6] UniProtKB - O24581 (BIP3_MAIZE) [7] Словарь терминов kodomo [8] Nematode Specific Peptide family, group A [ Caenorhabditis elegans ], NCBI |