Вернуться на страницу семестра
Задача: Найти и описать полиморфизмы у пациента
Часть I: подготовка чтений
0. Создание рабочей директории.
В директории /nfs/srv/databases/ngs/ создана директория e.mironova, в неё скопированы файлы с ридами (chr21.fastq) и хромосомой (chr21.fasta).
Подробнее о формате fastq - это тот же fasta формат, но в нём используются дополнительные символы, которые говорят о качестве прочтения и степени, с которой можно доверять каждому отдельному нуклеотиду.
Рис.1. Пересчёт качества в вероятность ошибки

1. Анализ качества чтений.
Контроль качества чтений с помощью программы FastQC. На выходе получена html страница с отчётом. Пример выдачи этой программы вы можете увидеть в пункте 2.
Для лучшего понимания происходящего - картинка ниже.
Рис.2. Ящик с усами / диаграмма размахов / boxplot (жёлтый прямоугольник на картинке выдачи FastQC)
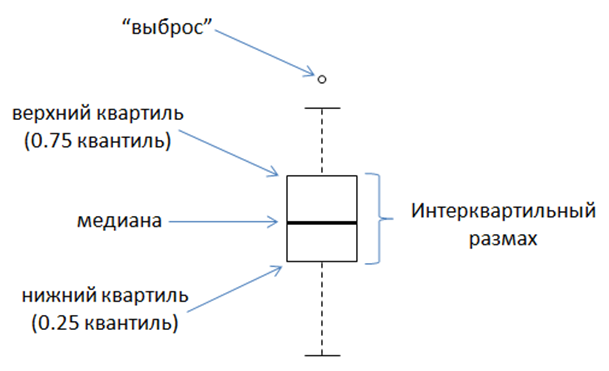
Дополнительное объяснение: желтый прямоугольник - интерквартальный размах (разница между верхней и нижней квартилями качества).
Это диапазон значений качества, при котором 25% ридов на данной позиции имет качество выше нижней границы и 25% меньше верхней, то есть половина ридов на данном нуклеодите имеет качество, которое попадает в данный отрезок.
Красная линия это медиана, синяя - среднее качество чтений.
2. Очистка чтений
Очистку чтений проведём с помощью программы Trimmomatic. С конца каждого чтения удалены нуклеотиды с качеством ниже 20, затем оставлены только чтения длиной не меньше 50 нуклеотидов.
Использовалась программа, установленная на kodomo, команду можно увидеть в таблице ниже. Программа выдала, что из 8158 ридов осталось 7858 (96,32%) и 300 (3,68%) были откинуты.
Для полученного улучшенного fastq файла снова была проведена проверка качества чтения и получена html страница. Итог и сравнение качества двух fastq файлов можно увидеть ниже.
| Рис. 1. Контроль качества ридов до тримминга | Рис. 2. Контроль после тримминга |
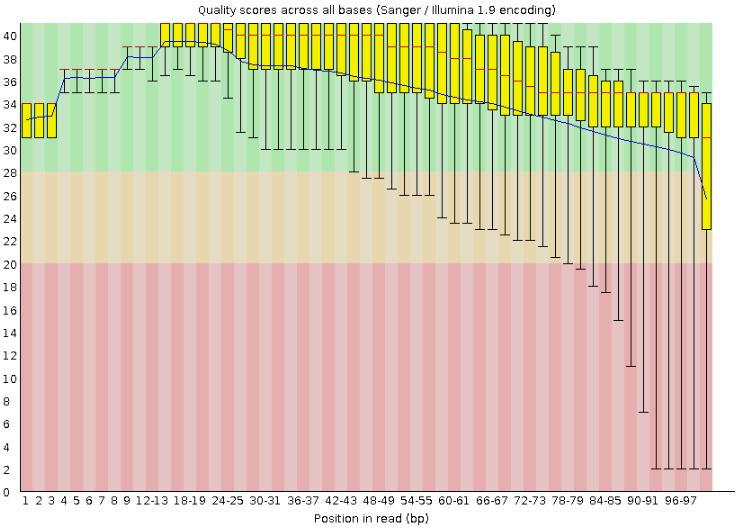 | 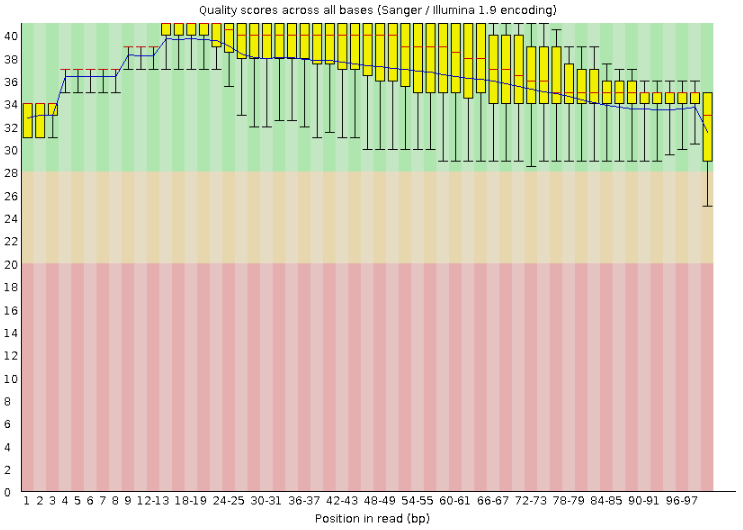 |
После тримминга (удаления «плохих» фрагментов чтений) мы видим, что все интерквантильные размахи качества позиций в ридах теперь в зелёной области (самый доверительный интервал).
Можно бы было сказать так не только о интерквантильном размахе, но и вообще о значениях качества, если бы минимум качества в последней позиции не ушёл в жёлтую область, но остальные минимумы качества заметно выросли,
конечно же, следом выросло и среднее значение - синяя линяя заметно поднялась. В некоторых позициях интерквантильные размахи уменьшились, но их уменьшения связано с поднятием нижней квартили, а не падением верхней, что также говорит об увеличении общего качества ридов.
Часть II: картирование чтений
3. Картирование чтений
Откартировать очищенные чтения с помощью программы Hisat2.
Этапы:
Сначала необходимо проиндексировать референсную последовательность; команда hisat2-build
Некоторые параметры hisat2:
-х – путь к индексу
-U – путь к чтениям
--no-softclip – запрет подрезания чтений
--no-spliced-alignment – картирование без разрывов
Затем построить выравнивание прочтений и референса в формате .sam. Запустите hisat2 с параметрами --no-spliced-alignment и --no-softclip
Сохранить вывод программы hisat2 в отдельный файл (вывод можно увидеть ниже)
|
7858 reads; of these:
7858 (100.00%) were unpaired; of these:
47 (0.60%) aligned 0 times
7808 (99.36%) aligned exactly 1 time
3 (0.04%) aligned >1 times
99.40% overall alignment rate
|
4. Анализ выравнивания
Переведите выравнивание чтений с референсом в бинарный формат .bam. Используйте пакет samtools, команда view: samtools view; Руководство по samtools.
Отсортируйте выравнивание чтений с референсом (получившийся после картирования .bam файл) по координате в референсе начала чтения; команда samtools sort;
Проиндексируйте отсортированный .bam файл командой samtools index
Выясните, сколько чтений откартировано на геном; загляните в вывод программы Hisat2. Видим, что 47 ридов не откартировано, 7808 встретились 1 раз, а 3 больше 1 раза.
В .sam файле также можно увидеть:
SRR2776256.15395984 – ID чтения
chr12 9822304 - хромосома и координата, куда «легло» чтение
100M – СIGAR: сжато кодирует информацию о выравнивании чтения
NM:i – расстояние до генома
NH:I – количество картирований для данного чтения
Часть III: Анализ SNP
5. Поиск SNP (single nucleotide polymorphism - однонуклеотидный полиморфизм) и инделей.
Создайте файл с полиморфизмами в формате .bcf; команда samtools mpileup -uf.
|
[mpileup] 1 samples in 1 input files
Set max per-file depth to 8000
|
Создайте файл со списком отличий между референсом и чтениями в формате .vcf. Используйте команду "bcftools call -cv" пакета bcftools.
Таблица 1. Три полиморфизма из .vcf файла
| Координата | Тип полиморфизма | Референс | Риды | Глубина покрытия данного места (сколько чтений пересекло конкретную позицию) | Качество чтений в данном месте |
| 45397408 | Делеция | CTGTGT | CTGT | 8 | 31.4691 |
| 45398160 | Вставка | gttgttttgttttgtttt | gTTGTTttgttttgttttgtttt | 14 | 55.4663 |
| 16334963 | Замена | T | G | 150 | 225.009 |
6. Аннотация SNP
Для работы с программой annovar из .vcf файла необходимо получить файл, с которым умеет работать эта программа. Сделать это можно с помощью скрипта convert2annovar.pl.
|
NOTICE: Finished reading 148 lines from VCF file
NOTICE: A total of 116 locus in VCF file passed QC threshold, representing 110 SNPs (70 transitions and 40 transversions) and 8 indels/substitutions
NOTICE: Finished writing 110 SNP genotypes (70 transitions and 40 transversions) and 6 indels/substitutions for 1 sample
|
Было найдено 110 snp и 6 инделей.
С помощью программы annovar проаннотируйте только полученные snp (индели не надо!). Используйте базы данных: refgene, dbsnp, 1000 genomes, GWAS, Clinvar.
узнать, какие из Ваших snp имеют rs, можно с помощью команды
|
annotate_variation.pl -filter -out outputfile -build hg19 -dbtype snp138 inputfile.human humandb/ |
где inputfile.human — входной файл, полученный после обработки .vcf с помощью convert2annovar.pl (расширение не имеет значения);
outputfile — выходной файл; humandb/ — директория, в которой лежат базы данных (все необходимые базы данных уже есть на kodomo, пользоваться опцией -downdb не надо!);
snp138 — база данных, с которой вы работаете. Базы данных в annovar часто обновляются, для корректного запуска программы всегда нужно знать, какая версия какой базы данных у Вас скачена.
Для вас: refgene — refGene; dbsnp — snp138; 1000 genomes — 1000g2014oct; GWAS — gwasCatalog; Clinvar — clinvar_20150629.
В Annovar существуют 3 типа аннотаций по базам данных, основанных на: генной разметке (gene-based annotation); разметке других регионов генома (region-based annotation);
фильтрации (filter-based annotation). Команды, с помощью которых можно проаннотировать полиморфизмы по необходимым базам данных есть в итоговой таблице ниже.
|
Вывод программы для аннотации по refgene
NOTICE: The --geneanno operation is set to ON by default
NOTICE: Reading gene annotation from /nfs/srv/databases/annovar/humandb/hg19_refGene.txt ... Done with 50914 transcripts
(including 11516 without coding sequence annotation) for 26271 unique genes
NOTICE: Reading FASTA sequences from /nfs/srv/databases/annovar/humandb/hg19_refGeneMrna.fa ... Done with 4 sequences
WARNING: A total of 345 sequences will be ignored due to lack of correct ORF annotation
NOTICE: Finished gene-based annotation on 110 genetic variants in polymorf.annovar
NOTICE: Output files were written to poly_refgene.variant_function, poly_refgene.exonic_variant_function
|
|
Вывод программы для аннотации по dbsnp
NOTICE: Variants matching filtering criteria are written to poly_dbsnp.hg19_snp138_dropped,
other variants are written to poly_dbsnp.hg19_snp138_filtered
NOTICE: Processing next batch with 110 unique variants in 110 input lines
NOTICE: Database index loaded. Total number of bins is 2894320 and the number of bins to be scanned is 75
NOTICE: Scanning filter database /nfs/srv/databases/annovar/humandb/hg19_snp138.txt...Done
|
|
Вывод программы для аннотации по 1000 genomes
NOTICE: Variants matching filtering criteria are written to poly_1000g.hg19_ALL.sites.2014_10_dropped,
other variants are written to poly_1000g.hg19_ALL.sites.2014_10_filtered
NOTICE: Processing next batch with 110 unique variants in 110 input lines
NOTICE: Database index loaded. Total number of bins is 2824642 and the number of bins to be scanned is 75
NOTICE: Scanning filter database /nfs/srv/databases/annovar/humandb/hg19_ALL.sites.2014_10.txt...Done
|
|
Вывод программы для аннотации по GWAS
NOTICE: Reading annotation database /nfs/srv/databases/annovar/humandb/hg19_gwasCatalog.txt ... Done with 18027 regions
NOTICE: Finished region-based annotation on 110 genetic variants in polymorf.annovar
NOTICE: Output file is written to poly_gwas.hg19_gwasCatalog
|
|
Вывод программы для аннотации по Clinvar
NOTICE: the --dbtype clinvar_20150629 is assumed to be in generic ANNOVAR database format
NOTICE: Variants matching filtering criteria are written to poly_clinvar.hg19_clinvar_20150629_dropped,
other variants are written to poly_clinvar.hg19_clinvar_20150629_filtered
NOTICE: Processing next batch with 110 unique variants in 110 input lines
NOTICE: Database index loaded. Total number of bins is 49139 and the number of bins to be scanned is 2
NOTICE: Scanning filter database /nfs/srv/databases/annovar/humandb/hg19_clinvar_20150629.txt...Done
|
Таблица 2. Команды, выполненные в течение практикума
| Командра | Функция | Выходные файлы |
| 1. Анализ качества чтений |
| fastqc chr21.fastq (аналогично для imp_chr21.fastq) | Вызов программы FastQC, которая контролирует качество чтений | chr21_fastqc.html chr21_fastqc.zip (отчет о программе в виде html файла) |
| 2. Очистка чтений |
| java -jar /usr/share/java/trimmomatic.jar SE -phred33 chr21.fastq impr_chr21.fastq TRAILING:20 MINLEN:50 | Запускает программу Trimmomatic. Отрезать с конца каждого чтения нуклеотиды с качеством ниже 20, оставьте чтения длиной не меньше 50 нуклеотидов. | impr_chr21.fastq |
| 3. Картирование чтений |
| export PATH=${PATH}:/home/students/y06/anastaisha_w/hisat2-2.0.5 | Пакет программ, которые в данной папке, теперь доступны к вызову в putty | - |
| hisat2-build chr21.fasta chr21 | Индексирует референсную последовательность | Индексированный chr21.fasta |
| hisat2 --no-spliced-alignment --no-softclip -x chr21 -U impr_chr21.fastq -S result.sam | Выравнивание референсной последовательности и ридов после тримминга | result.sam |
| 4. Анализ выравнивания |
| samtools view result.sam -b -o result.bam | Перевод выравнивания чтений с референсом в бинарный формат .bam | result.bam |
| samtools sort result.bam -T a.txt -o sort_result.bam | Сортировка выравнивания ридов с референсом (.bam файл) по координате в референсе начала чтения | sort_result.bam |
| samtools index sort_result.bam | Индексировать отсортированный .bam файл | Индексированный sort_result.bam |
| 5. Поиск SNP и инделей |
| samtools mpileup -uf chr21.fasta sort_result.bam --output polymorf.bcf | Создать файл с полиморфизмами | Бинарный файл polymorf.bcf, который содержит информацию об отличиях исследуемого образца от референса |
| bcftools call -cv polymorf.bcf -o polymorf.vcf | Перевод файла с полиморфизмами из формата bcf в vcf | polymorf.vcf |
| 6. Аннотация SNP |
| perl /nfs/srv/databases/annovar/convert2annovar.pl -format vcf4 polymorf.vcf > polymorf.annovar | Получен файл, с которым умеет работать программа annovar (чтобы работать с SNP, нужно вручную удалить индели из файла) | polymorf.annovar |
| perl /nfs/srv/databases/annovar/annotate_variation.pl -out poly_refgene -build hg19 polymorf.annovar /nfs/srv/databases/annovar/humandb/ | Аннотация snp по базе данных refgene | poly_refgene.variant_function, poly_refgene.exonic_variant_function |
| perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -out poly_dbsnp -build hg19 -dbtype snp138 polymorf.annovar /nfs/srv/databases/annovar/humandb/ | Аннотация snp по базе данных dbsnp (snp138) | poly_dbsnp.hg19_snp138_dropped poly_dbsnp.hg19_snp138_filtered |
| perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -dbtype 1000g2014oct_all -out poly_1000g -buildver hg19 polymorf.annovar /nfs/srv/databases/annovar/humandb/ | Аннотация snp по базе данных 1000 genomes | poly_1000g.hg19_ALL.sites.2014_10_dropped, poly_1000g.hg19_ALL.sites.2014_10_filtered |
| perl /nfs/srv/databases/annovar/annotate_variation.pl -regionanno -dbtype gwasCatalog -out poly_gwas -build hg19 polymorf.annovar /nfs/srv/databases/annovar/humandb/ | Аннотация snp по базе данных GWAS | poly_gwas.hg19_gwasCatalog |
| perl /nfs/srv/databases/annovar/annotate_variation.pl -filter -out poly_clinvar -dbtype clinvar_20150629 -buildver hg19 polymorf.annovar /nfs/srv/databases/annovar/humandb/ | Аннотация snp по базе данных Clinvar | poly_clinvar.hg19_clinvar_20150629_dropped, poly_clinvar.hg19_clinvar_20150629_filtered |
В отчете укажите:
команды
описание трех полиморфизмов из .vcf файла
cколько snp и сколько инделей Вы получили?
Хорошее ли покрытие и качество у найденных полиморфизмов?
О покрытии и качестве полиморфизмов можно судить по таблице 6.
Мы видим, что более, чем у половины (62 nps) плохое покрытие - < 5, у 38 nps глубина покрытия больше или равна 10, максимальное покрытие - 150.
У 37 snp качество меньше 28 (красная область в программе FastQC). У 29 - качество очень хорошее - больше 100.
На какие категории делит snp база данных refseq в annovar? Сколько snp у Вас попало в каждую группу?
Таблица 3. Категории refseq в annovar
| Обозначение |
Количество |
Значение |
| exonic |
4 |
Экзонные SNP |
| splicing |
0 |
SNP сайтов сплайсинга |
| ncRNA |
0 |
SNP в генах некодирующих РНК |
| UTR5 |
0 |
SNP в области 5' нетранслируемых областей |
| UTR3 |
9 |
SNP в области 3' нетранслируемых областей |
| intronic |
96 |
интронные SNP |
| upstream |
1 |
SNP находится в области 1 kb до сайта начала транскрипции |
| downstream |
0 |
SNP находится в области 1 kb до сайта начала транскрипции |
| intergenic |
0 |
SNP расположены в межгенных областях |
В какие гены попали Ваши snp? Гены, можно увидеть в таблице 6: NRIP1, UBASH3A, AGPAT3.
К каким нуклеотидным и аминокислотным заменам привели snp?
Это можно увидеть в аннотации по RefGene, таблица приведена ниже.
Таблица 4. Замены аминокислот
| Синонимичность | Ген | Координата | Референс | Рид | Замена |
| Да | NRIP1 | 16340289 | C | T | Нет. Остался глицин |
| Нет | UBASH3A | 43824106 | A | G | Серин -> Глицин |
| Да | UBASH3A | 43824123 | G | C | Нет. Остался серин |
| Да | UBASH3A | 43863521 | A | G | Нет. Остался пролин |
Сколько snp имеет rs? В аннотации по банку dbsnp (snp138) можно увидеть, что всего 67 snp имеют rs.
Что Вы можете сказать о частоте найденных snp? См. таблицу 6. Частота встречаемости находится в интервале от 0,01 до 0,8 (но для одного нуклеотида она равна 1).
Что Вы можете сказать о клинической аннотации snp? Базы GWAS, Clinvar предоставляют клиническое описание SNP. Поиск по Clinvar не дал результатов, результат по GWAS:
Таблица 5. Клиническая аннотация
| Заболевание | Координата | Референс | Рид | Гетеро-/Гомозигота | Качество |
| Name=Cognitive performance | 16340289 | C | T | het | 225.009 |
| Name=Type 1 diabetes | 43836390 | A | G | hom | 137.009 |
| Name=Phospholipid levels (plasma) | 45404338 | A | G | het | 35.0083 |
Составьте сводную таблицу, в которую бы вошли все snp и их характеристики по использованным для аннотации базам данных. По желанию можно выделить и/или описать наиболее интересные на Ваш взгляд полиморфизмы.
Таблица 6. Информация о snp, собранная с ПОЧТИ ВСЕХ аннотаций
| Координата | Референс | Рид | Гетеро-/Гомозигота | Качество | Глубина | Располежен (refgene) | Ген (refgene) | rs (dbsnp) | Встречаемость (1000genome) |
| 16334658 | C | T | het | 212.009 | 32 | UTR3 | NRIP1(NM_003489:c.*2379G>A) | rs1056947 | 0.159345 |
| 16334963 | T | G | het | 225.009 | 150 | UTR3 | NRIP1(NM_003489:c.*2074A>C) | rs2822988 | 0.321486 |
| 16335402 | C | T | het | 42.0073 | 24 | UTR3 | NRIP1(NM_003489:c.*1635G>A) | rs2142450 | 0.148562 |
| 16335515 | C | T | het | 225.009 | 54 | UTR3 | NRIP1(NM_003489:c.*1522G>A) | rs1041403 | 0.316693 |
| 16336647 | A | T | het | 28.0137 | 6 | UTR3 | NRIP1(NM_003489:c.*390T>A) | | |
| 16336804 | T | C | hom | 221.999 | 62 | UTR3 | NRIP1(NM_003489:c.*233A>G) | rs1056930 | 0.772364 |
| 16340289 | C | T | het | 225.009 | 60 | exonic | NRIP1 | rs2229741 | 0.599441 |
| 16359397 | C | T | hom | 37.7652 | 2 | intronic | NRIP1 | | |
| 16379074 | C | A | hom | 43.7647 | 2 | intronic | NRIP1 | | |
| 16396991 | A | G | hom | 37.7652 | 2 | intronic | NRIP1 | rs9636612 | 0.590455 |
| 16403246 | G | A | hom | 39.765 | 2 | intronic | NRIP1 | rs62220722 | |
| 16403296 | G | A | hom | 35.7656 | 2 | intronic | NRIP1 | rs4817585 | |
| 16405589 | A | G | hom | 39.765 | 2 | intronic | NRIP1 | rs7279087 | 0.199281 |
| 16418176 | C | T | hom | 26.7735 | 2 | intronic | NRIP1 | rs9979082 | 0.132788 |
| 16432068 | T | C | hom | 41.7648 | 2 | intronic | NRIP1 | rs13052454 | 0.219249 |
| 43823968 | C | T | het | 13.2188 | 10 | upstream | UBASH3A | | |
| 43824106 | A | G | het | 210.009 | 16 | exonic | UBASH3A | rs2277798 | 0.545327 |
| 43824123 | G | C | het | 225.01 | 24 | exonic | UBASH3A | rs2277799 | 0.39976 |
| 43824193 | G | A | het | 215.009 | 24 | intronic | UBASH3A | rs11909229 | 0.0517173 |
| 43826112 | C | T | hom | 17.8363 | 2 | intronic | UBASH3A | rs56268047 | 0.00998403 |
| 43826344 | C | T | hom | 197.974 | 20 | intronic | UBASH3A | rs3746923 | 0.429313 |
| 43826391 | T | C | hom | 221.999 | 32 | intronic | UBASH3A | rs3746924 | 0.466254 |
| 43826618 | C | T | het | 128.008 | 26 | intronic | UBASH3A | rs3746925 | 0.169529 |
| 43827270 | C | A | hom | 84.5134 | 4 | intronic | UBASH3A | | |
| 43829758 | G | A | hom | 221.999 | 32 | intronic | UBASH3A | rs11702374 | 0.403754 |
| 43829832 | A | C | het | 7.79563 | 10 | intronic | UBASH3A | | |
| 43829895 | C | G | het | 17.0924 | 6 | intronic | UBASH3A | | |
| 43830958 | A | T | het | 4.12848 | 4 | intronic | UBASH3A | | |
| 43831151 | G | A | hom | 117.514 | 4 | intronic | UBASH3A | rs13047735 | 0.189497 |
| 43832844 | G | T | het | 26.0177 | 6 | intronic | UBASH3A | | |
| 43832856 | G | C | hom | 142.007 | 10 | intronic | UBASH3A | rs3761377 | 0.202276 |
| 43832918 | T | C | hom | 171.998 | 18 | intronic | UBASH3A | rs3761378 | 0.403355 |
| 43836251 | C | A | hom | 23.7825 | 2 | intronic | UBASH3A | | |
| 43836390 | A | G | hom | 137.009 | 34 | intronic | UBASH3A | rs9976767 | 0.409145 |
| 43836835 | G | A | hom | 126.133 | 6 | intronic | UBASH3A | rs1977952 | 0.209465 |
| 43838403 | T | A | hom | 221.999 | 46 | intronic | UBASH3A | rs9974660 | 0.210463 |
| 43846956 | T | G | hom | 177.998 | 24 | intronic | UBASH3A | rs2839510 | 0.184704 |
| 43847011 | G | T | het | 16.1143 | 10 | intronic | UBASH3A | | |
| 43852015 | T | C | het | 33.0105 | 4 | intronic | UBASH3A | rs73905659 | 0.13758 |
| 43852037 | T | C | het | 128.009 | 10 | intronic | UBASH3A | rs12482947 | 0.536142 |
| 43855180 | T | A | het | 4.76875 | 12 | intronic | UBASH3A | | |
| 43859064 | C | T | hom | 43.7647 | 2 | intronic | UBASH3A | rs2839514 | 0.560703 |
| 43860659 | A | G | hom | 2915337 | 2 | intronic | UBASH3A | rs2839515 | 0.526957 |
| 43862466 | G | A | het | 225.009 | 54 | intronic | UBASH3A | rs2254368 | 0.496206 |
| 43863247 | T | C | het | 4.76875 | 10 | intronic | UBASH3A | rs17114945 | 0.0397364 |
| 43863293 | T | C | het | 9.52088 | 12 | intronic | UBASH3A | rs17114946 | 0.0155751 |
| 43863521 | A | G | het | 200.009 | 26 | exonic | UBASH3A | rs868092 | 0.616813 |
| 43864519 | G | A | het | 125.008 | 24 | intronic | UBASH3A | rs4920104 | 0.487021 |
| 43865046 | G | A | hom | 15.8779 | 2 | intronic | UBASH3A | | |
| 43865590 | C | G | hom | 43.7647 | 2 | intronic | UBASH3A | rs3788015 | 0.166733 |
| 43867059 | C | T | het | 225.009 | 36 | intronic | UBASH3A | rs3827232 | 0.245008 |
| 45289508 | G | A | hom | 41.7648 | 2 | intronic | AGPAT3 | | |
| 45289542 | G | T | hom | 37.7652 | 2 | intronic | AGPAT3 | | |
| 45294162 | T | C | hom | 43.7647 | 2 | intronic | AGPAT3 | rs8133789 | 0.81869 |
| 45299029 | G | A | hom | 41.7648 | 2 | intronic | AGPAT3 | | |
| 45302889 | G | A | hom | 39.765 | 2 | intronic | AGPAT3 | | |
| 45309646 | C | G | hom | 35.7656 | 2 | intronic | AGPAT3 | | |
| 45317365 | A | G | het | 54.0072 | 12 | intronic | AGPAT3 | rs6518335 | 0.810903 |
| 45319619 | C | T | hom | 23.7825 | 2 | intronic | AGPAT3 | rs2070541 | 0.400559 |
| 45323989 | G | C | het | 22.0341 | 8 | intronic | AGPAT3 | | |
| 45324137 | A | G | hom | 3.98131 | 2 | intronic | AGPAT3 | | |
| 45325916 | G | T | hom | 35.7656 | 4 | intronic | AGPAT3 | rs2838429 | 0.389177 |
| 45339066 | A | G | hom | 43.7647 | 2 | intronic | AGPAT3 | rs420826 | 1 |
| 45340268 | G | A | hom | 15.8779 | 2 | intronic | AGPAT3 | | |
| 45342711 | G | T | hom | 30.768 | 2 | intronic | AGPAT3 | | |
| 45344373 | C | A | hom | 35.7656 | 2 | intronic | AGPAT3 | | |
| 45345133 | G | T | hom | 2915337 | 2 | intronic | AGPAT3 | | |
| 45348068 | G | T | hom | 2915337 | 2 | intronic | AGPAT3 | | |
| 45349756 | G | A | hom | 23.7825 | 2 | intronic | AGPAT3 | rs2838439 | 0.776957 |
| 45349784 | A | G | hom | 24.7788 | 2 | intronic | AGPAT3 | rs2070547 | 0.775958 |
| 45354316 | C | T | hom | 41.7648 | 2 | intronic | AGPAT3 | | |
| 45354744 | G | A | hom | 37.7652 | 2 | intronic | AGPAT3 | rs1888525 | 0.76278 |
| 45354997 | G | T | het | 9.52088 | 6 | intronic | AGPAT3 | | |
| 45356073 | T | G | hom | 35.7656 | 2 | intronic | AGPAT3 | rs9306165 | 0.775958 |
| 45356183 | A | G | hom | 28.7701 | 2 | intronic | AGPAT3 | rs9306166 | 0.762979 |
| 45356294 | G | A | het | 4.12848 | 6 | intronic | AGPAT3 | | |
| 45356761 | C | A | hom | 30.768 | 2 | intronic | AGPAT3 | | |
| 45360045 | G | A | het | 149.008 | 40 | intronic | AGPAT3 | rs9981554 | 0.0339457 |
| 45361117 | A | C | hom | 3.98131 | 2 | intronic | AGPAT3 | | |
| 45363892 | C | A | het | 15.1417 | 4 | intronic | AGPAT3 | | |
| 45364210 | G | C | hom | 30.768 | 2 | intronic | AGPAT3 | rs73906678 | 0.033746 |
| 45366893 | A | G | het | 17.0924 | 6 | intronic | AGPAT3 | rs11911486 | 0.459864 |
| 45372453 | G | C | hom | 3.98131 | 2 | intronic | AGPAT3 | | |
| 45373020 | A | G | hom | 28.7701 | 2 | intronic | AGPAT3 | rs9974326 | 0.395966 |
| 45373499 | G | C | hom | 43.7647 | 2 | intronic | AGPAT3 | | |
| 45373938 | G | T | hom | 39.765 | 2 | intronic | AGPAT3 | | |
| 45374071 | C | T | hom | 43.7647 | 2 | intronic | AGPAT3 | | |
| 45375397 | C | A | hom | 3.98131 | 2 | intronic | AGPAT3 | | |
| 45376207 | A | G | hom | 28.7701 | 2 | intronic | AGPAT3 | rs915876 | 0.498403 |
| 45378476 | G | A | het | 22.0341 | 8 | intronic | AGPAT3 | | |
| 45378552 | A | G | hom | 80.5134 | 4 | intronic | AGPAT3 | rs61033068 | 0.497604 |
| 45378963 | T | C | het | 135.043 | 16 | intronic | AGPAT3 | rs200253938 | 0.173922 |
| 45379795 | G | A | het | 43.0073 | 32 | intronic | AGPAT3 | rs73906687 | 0.0219649 |
| 45380130 | C | T | hom | 13.943 | 2 | intronic | AGPAT3 | rs2838450 | 0.391773 |
| 45380213 | G | A | hom | 9.30613 | 2 | intronic | AGPAT3 | rs12626709 | 0.279353 |
| 45381944 | C | T | hom | 39.765 | 2 | intronic | AGPAT3 | rs4818875 | 0.402756 |
| 45385041 | G | T | het | 24.0241 | 6 | intronic | AGPAT3 | | |
| 45389261 | A | G | het | 132.008 | 30 | intronic | AGPAT3 | rs73906692 | 0.11861 |
| 45389348 | T | C | hom | 35.7656 | 2 | intronic | AGPAT3 | rs2838454 | 0.254193 |
| 45391448 | G | T | het | 7.79563 | 12 | intronic | AGPAT3 | | |
| 45391752 | T | C | hom | 100.514 | 4 | intronic | AGPAT3 | rs9976628 | 0.694289 |
| 45392846 | A | G | hom | 43.7647 | 2 | intronic | AGPAT3 | rs3788094 | 0.403355 |
| 45393034 | C | T | hom | 43.7647 | 2 | intronic | AGPAT3 | | |
| 45397335 | C | G | hom | 24.7788 | 2 | intronic | AGPAT3 | rs2838457 | 0.151158 |
| 45397393 | G | A | het | 22.0341 | 8 | intronic | AGPAT3 | | |
| 45400664 | G | T | het | 33.0091 | 4 | intronic | AGPAT3 | | |
| 45401246 | G | C | het | 16.1143 | 12 | intronic | AGPAT3 | | |
| 45404338 | A | G | het | 35.0083 | 20 | UTR3 | AGPAT3(NM_020132:c.*2065A>G,NM_001037553:c.*2065A>G) | rs7435 | 0.365415 |
| 45406095 | C | G | hom | 39.765 | 2 | UTR3 | AGPAT3(NM_020132:c.*3822C>G,NM_001037553:c.*3822C>G) | rs14151 | 0.464657 |
| 45406878 | C | T | het | 106.008 | 16 | UTR3 | AGPAT3(NM_020132:c.*4605C>T,NM_001037553:c.*4605C>T) | rs56798348 | 0.0131789 |
|









