Вернуться на страницу семестра
Задача: картировать чтения, полученные в результате секвенирования транскриптома (человек, версия сборки генома hg19)
Часть I: подготовка чтений
0. Создание рабочей директории и скачивание данных.
Код доступа проекта по секвенированию бактерии Buchnera aphidicola: SRR4240360. Cам проект доступен по адресу http://www.ebi.ac.uk/ena/data/view/SRR4240360. SRR4240379.fastq.
1. Подготовка чтений программой trimmomatic
Прежде всего надо удалить возможные остатки адаптеров. Для этого можно использовать следующий "step" программы trimmomatic: ILLUMINACLIP:adapters.fasta:2:7:7, где adapters.fasta –
файл с адаптерами. Адаптеры для Illumina собраны в файлах в директории /P/y16/term3/block3/adapters. Я создала свой файл, в котором объединила все адаптеры из этих файлов вместе.
Вывод программы при удалении адаптеров - Input Reads: 8254632 Surviving: 8212761 (99,49%) Dropped: 41871 (0,51%)
После этого удалите плохие буквы с концов чтений, оставив только чтения длиной не менее 30. Вывод программы: Input Reads: 8212761 Surviving: 7935083 (96,62%) Dropped: 277678 (3,38%), размер файла SRR4240360.fastq до очистки - 832 Мб, после - 798 Мб.
Часть II: реализация сборки с помощью графа де Брайна через программу Velvet
Запустите программу velveth, сначала с опцией -help. Разберитесь, как запустить её в данном случае. чтобы она подготовила k-меры длины k=29 (максимально возможной при нашей длине чтений). Практически во всём можно разобраться, читая help, но можно почитать и руководство. Длина k-мера называется hash_length, чтения в нашем случае короткие и не парные (short).
2. Подготовка k-меров длины 29 velveth
Есть два основных типа алгоритмов сборки: OLC = overlap-layout-consensus (алгоритмы OLC работают непосредственно с ридами), de Bruijn graph (алгоритмы, использующие граф де Брайна, сначала составляют список k-меров (слов длины k, например k = 30), встретившихся в ридах.
3. Сборка velvetg
Запускаю velvetg (сборка на основе k-меров). Вывод программы: Final graph has 1944 nodes and n50 of 34768, max 80212, total 710306, using 0/8254632 reads
N50 - 34768, длины трёх самых длинных контигов: ID 11 - 80212, ID 7 - 70305, ID 9 - 68353; и соответственно их покрытие: 44.44, 49.82, 47.30.
Есть контиги с аномально большим: ID 1563 - 1026604, ID 1565 - 72483, ID 1568 - 23411; и аномально малым покрытием: ID 540 - 1.
О том, как считается покрытие и длина: у каждого k-мера есть покрытие (сколько раз он встретился в ридах), покрытие контига это среднеарифметическое для покрытия всех k-меров, из которых он состоит.
Длина также тесно связана с k-мерами: длина 1 означает, что контиг длины 1 k-мер, то есть 29, длина 3 означает, что в контиге есть 3 k-мера, то есть 31 нуклеотид.
Часть III: Анализ. Сравнение программой megablast
4. Выравнивание в blastn
Программой megablast каждый из трёх самых длинных контигов сравнивался с хромосомой Buchnera aphidicola (GenBank/EMBL AC — CP009253).
Координаты участка хромосомы, соответствующего контигу, характеристики выравнивания или выравниваний
(число однонуклеотидных различий, число гэпов).
Про каждый контиг требуется понятное описание того, как именно он "ложится" на банковский геном.
5. Анализ результатов
Все сводные данные по выравниваниям в таблице.
Рисунок 1. Выравнивание контига 11 с хромосомой
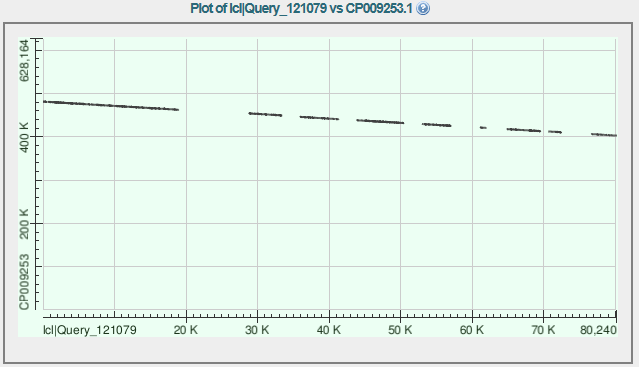
Таблица 1. Данные о выравниваниях
| % identity | length | mismatches | gap opens | q. Start | q. End | s. Start | s. End | evalue |
| 82.460 | 439 | 1490 | 14 | 1 | 434 | 481301 | 480874 | 1.53e-101 |
| 74.125 | 5971 | 817 | 196 | 640 | 6514 | 480660 | 474844 | 0.0 |
| 77.020 | 7389 | 991 | 168 | 6617 | 13930 | 474667 | 467412 | 0.0 |
| 77.009 | 5015 | 542 | 135 | 13960 | 18901 | 467421 | 462496 | 0.0 |
| 75.465 | 4732 | 1295 | 112 | 28792 | 33444 | 454069 | 449411 | 0.0 |
| 80.255 | 3054 | 1009 | 52 | 36016 | 39043 | 445895 | 442877 | 0.0 |
| 79.044 | 1694 | 837 | 24 | 39173 | 40848 | 442817 | 441135 | 0.0 |
| 88.947 | 190 | 368 | 6 | 40970 | 41151 | 440944 | 440755 | 9.77e-59 |
| 97.561 | 82 | 566 | 1 | 41241 | 41322 | 440732 | 440652 | 4.71e-32 |
| 77.082 | 2954 | 710 | 86 | 43947 | 46870 | 438139 | 435267 | 0.0 |
| 75.193 | 3495 | 320 | 123 | 46932 | 50361 | 435241 | 431839 | 0.0 |
| 76.199 | 4151 | 154 | 115 | 52963 | 57041 | 429483 | 425412 | 0.0 |
| 79.113 | 857 | 326 | 7 | 61151 | 62004 | 421327 | 420477 | 1.81e-165 |
| 79.528 | 4665 | 170 | 109 | 64936 | 69530 | 417677 | 413081 | 0.0 |
| 80.799 | 1828 | 61 | 28 | 70617 | 72431 | 412321 | 410512 | 0.0 |
| 82.361 | 2177 | 13 | 15 | 76679 | 78847 | 406218 | 404050 | 0.0 |
| 85.924 | 1158 | 1 | 5 | 79090 | 80240 | 403823 | 402668 | 0.0 |
Координаты участка хромосомы, соответствующего контигу:402668-481301, выравнивание по - цепи
Рисунок 2. Выравнивание контига 7 с хромосомой
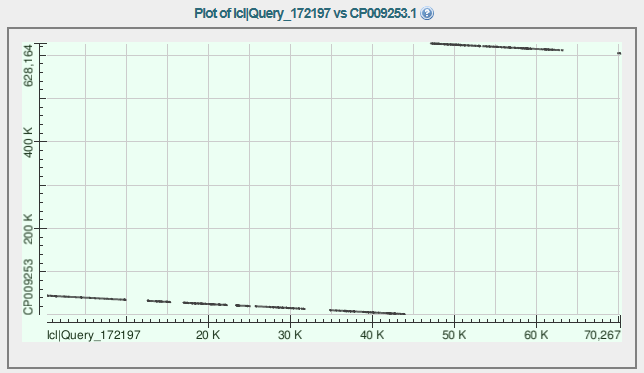
Таблица 2. Данные о выравниваниях
| % identity | length | mismatches | gap opens | q. Start | q. End | s. Start | s. End | evalue |
| 82.851 | 9633 | 1522 | 100 | 330 | 9895 | 44693 | 35124 | 0.0 |
| 77.422 | 2777 | 1738 | 71 | 12611 | 15347 | 32745 | 30013 | 0.0 |
| 76.551 | 5433 | 1350 | 166 | 16955 | 22304 | 28363 | 23067 | 0.0 |
| 81.524 | 1851 | 1247 | 41 | 23329 | 25153 | 22183 | 20358 | 0.0 |
| 85.253 | 2231 | 1055 | 23 | 25732 | 27942 | 20182 | 17962 | 0.0 |
| 75.976 | 3226 | 299 | 68 | 28020 | 31190 | 17919 | 14727 | 0.0 |
| 82.218 | 478 | 687 | 8 | 31350 | 31824 | 14465 | 13994 | 1.33e-111 |
| 78.380 | 9223 | 543 | 201 | 34834 | 43923 | 11103 | 2004 | 0.0 |
| 75.782 | 6173 | 291 | 207 | 47089 | 53136 | 627104 | 621055 | 0.0 |
| 79.211 | 7379 | 395 | 144 | 53366 | 60670 | 620926 | 613658 | 0.0 |
| 77.900 | 2086 | 82 | 42 | 60779 | 62845 | 613671 | 611633 | 0.0 |
| 79.461 | 297 | 76 | 2 | 62977 | 63272 | 611524 | 611229 | 3.10e-53 |
| 82.222 | 495 | 59 | 5 | 69778 | 70267 | 604795 | 604302 | 3.66e-117 |
Координаты участка хромосомы, соответствующего контигу:2004-44693 и 604302-627104, выравнивание по - цепи, на самом деле, мы видим такую картину не из-за перенесения крупного участка генома, а из-за того, что ДНК кольцевая
Рисунок 3. Выравнивание контига 9 с хромосомой
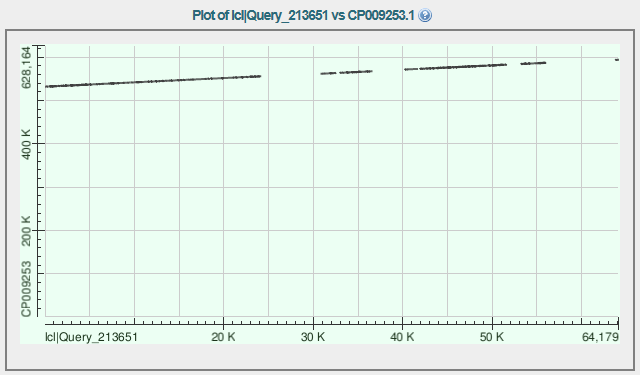
Таблица 3. Данные о выравниваниях
| % identity | length | mismatches | gap opens | q. Start | q. End | s. Start | s. End | evalue |
| 79.464 | 18553 | 3322 | 356 | 1 | 18334 | 531936 | 550219 | 0.0 |
| 80.866 | 5655 | 955 | 97 | 18541 | 24178 | 550361 | 555905 | 0.0 |
| 78.982 | 1689 | 2141 | 17 | 30812 | 32478 | 561741 | 563423 | 0.0 |
| 77.808 | 3731 | 698 | 85 | 32948 | 36626 | 563837 | 567489 | 0.0 |
| 84.446 | 1453 | 220 | 5 | 40266 | 41715 | 571558 | 573007 | 0.0 |
| 73.466 | 9825 | 569 | 361 | 41969 | 51557 | 573092 | 582686 | 0.0 |
| 75.684 | 2776 | 327 | 84 | 53245 | 55963 | 584329 | 587055 | 0.0 |
| 80.501 | 359 | 66 | 4 | 63823 | 64179 | 593743 | 594099 | 3.79e-72 |
Координаты участка хромосомы, соответствующего контигу:531936-594099, выравнивание по + цепи
Таблица 4. Команды, выполненные в течение практикума
| Команда | Функция | Выходные файлы |
| 1. Подготовка чтений |
| gunzip SRR4240360.fastq.gz | Разархивировать | SRR4240360.fastq |
| cat /P/y16/term3/block3/adapters/*.fa > adapters.fasta | Создать свой файл, в котором объединить все адаптеры | adapters.fasta со всеми адаптерами |
| java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240360.fastq noad_SRR4240360.fastq ILLUMINACLIP:adapters.fasta:2:7:7 | Удалить возможные остатки адаптеров, которые записаны в adapters.fasta | noad_SRR4240360.fastq |
| java -jar /usr/share/java/trimmomatic.jar SE -phred33 noad_SRR4240360.fastq impr_SRR4240360.fastq TRAILING:20 MINLEN:30 | Запускает программу Trimmomatic. Отрезать с конца каждого чтения нуклеотиды с качеством ниже 20, оставьте чтения длиной не меньше 30 нуклеотидов | impr_SRR4240360.fastq |
| 2. Сборка с помощью velvet |
| velveth /nfs/srv/databases/ngs/e.mironova/pr14 29 -short -fastq SRR4240360.fastq | Подготовлены k-меры длиной k=29 | Файлы Log, Roadmaps и Sequences |
| velvetg . | Сборка контигов в текущей папке | Graph, PreGraph, LastGraph, contigs.fa, stats.txt |
|










